はじめに
RDKitで、SMARTSで反応パターンを定義し、そのパターンに基づき反応物から生成物を生成してみたい。
環境
- Windows 10
- RDkit 2019.03.3.0
方法
参考文献を参考にしてほしい。
今回ためしてみた反応
今回は炭素原子の水酸化反応を試してみたい。SMARTSのパターンでいうと以下のようなものになる。
[C:1]>>[C:1][OH]
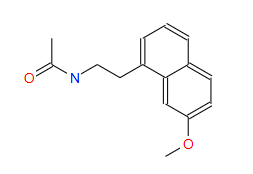
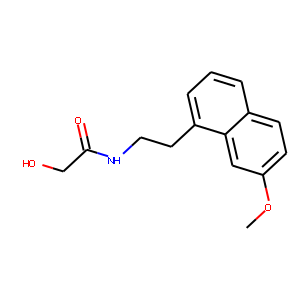
反応物は以下の化合物を試してみた。
ソース
ソースは以下の通りだ。反応により生成された生成物を全て画像に出力してみた。
from rdkit.Chem import AllChem
from rdkit.Chem.Draw import rdMolDraw2D
from io import BytesIO
from cairosvg import svg2png
from IPython.display import SVG
from rdkit import Chem
# 画像出力
def generate_image(mol, size, path, name):
image_data = BytesIO()
view = rdMolDraw2D.MolDraw2DSVG(size[0], size[1])
tm = rdMolDraw2D.PrepareMolForDrawing(mol)
view.DrawMolecule(tm)
view.FinishDrawing()
svg = view.GetDrawingText()
SVG(svg.replace('svg:', ''))
print(path + "/" + name)
try:
svg2png(bytestring=svg, write_to=path + "/" + name + ".png")
except:
pass
def main():
reactant_1 = Chem.MolFromSmiles('COc2ccc1cccc(CCNC(C)=O)c1c2')
reaction_pattern = '[C:1]>>[C:1][OH]'
rxn = AllChem.ReactionFromSmarts(reaction_pattern)
x = rxn.RunReactants([reactant_1])
for i, mol in enumerate(x):
print(mol[0])
try:
Chem.SanitizeMol(mol[0])
generate_image(mol[0], (300, 300), "tmp", "metablite_{0}".format(i))
except:
print("Error")
print(Chem.MolToMolBlock(mol[0]))
if __name__ == "__main__":
main()
確認
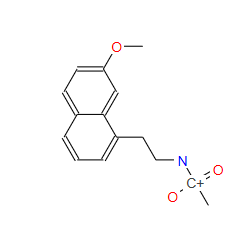
生成物は4つ得られた。1つはサニタイジングする際に電荷の関係でエラーになった。
結果を画像で確認してみよう。
生成物1
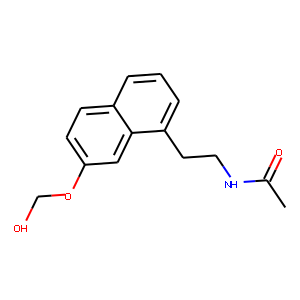
生成物2
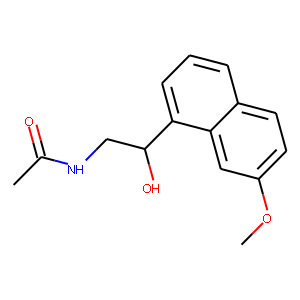
生成物3
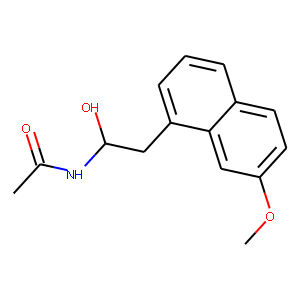
生成物4
生成物5(サニタイジング処理で電荷エラーになったもの)
考察
- 生成されたものは予想通りの結果である。
- 芳香環のC原子に対する反応はなかったが、その理由はよくわからない。仕様の確認が必要である(今後の課題)
- いずれにせよ、生成された反応物が、妥当な化合物などうかの判定は必要になりそうだ。