はじめに
フィンガープリントを使って予測モデルを作成した場合、説明変数として利用されたフィンガープリントのビットがどういうものか分からないと、予測のメカニズムの理解が難しいだろう。
Morganフィンガープリントについては、参考文献に示したようにビットを可視化するための情報がいくつかある.
今回は、情報が少ない AtomPairFingerprint についての可視化方法を検討したので共有したい。
AtomPairFingerprintとは?
AtomPairFingerprintとは、簡単に言うと分子内における原子のペアの出現に関するフィンガープリントだ。もう少し詳しく言うと、C原子とN原子が同時に出現したというような単純なものではなく、同じC原子であっても結合の違いを考慮したり、同じペアであってもペア同士の距離の違いを考慮したりしている。
詳しくは、アトムペアやドナーアクセプターペアフィンガープリントは分子の形状を捉えた化学表現 を見てほしい(丸投げ)
本記事の目的
本記事では、AtomPairFingerprintのビットとして「689188」のようなコードが与えられたときに、そのコードが化合物のどの部分に該当するのかを可視化する方法をお伝えすることである。
環境
- RDKit 2021.09.2
- cairosvg
やり方
準備
例えば、
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Descriptors, Draw
from rdkit.ML.Descriptors import MoleculeDescriptors
from rdkit.Chem.AtomPairs import Pairs, Utils
fp = AllChem.GetAtomPairFingerprint(mol)
d = fp.GetNonzeroElements()
for key in d.keys():
explained = Pairs.ExplainPairScore(key)
print(key, explained)
のように、分子のAtomPairFingerprintを計算し、ExplainPairScore関数を使うと、
541730 (('C', 1, 0), 2, ('C', 1, 0))
541734 (('C', 1, 0), 6, ('C', 1, 0))
590881 (('C', 1, 0), 1, ('C', 4, 0))
590885 (('C', 1, 0), 5, ('C', 4, 0))
590980 (('C', 4, 0), 4, ('C', 4, 0))
689187 (('C', 1, 0), 3, ('C', 2, 1))
689188 (('C', 1, 0), 4, ('C', 2, 1))
689191 (('C', 1, 0), 7, ('C', 2, 1))
689193 (('C', 1, 0), 9, ('C', 2, 1))
689194 (('C', 1, 0), 10, ('C', 2, 1))
689195 (('C', 1, 0), 11, ('C', 2, 1))
のように、分子に含まれるAtomPairFingerprintのビットを表す各コードについて、それがどのような原子ペア、距離を表しているかを得ることができる。具体的には、ExplainPairScoreの戻り値のタップルの1番目は、アトムペアの1つめの原子コード、2番目はアトムペアの距離、3番目はアトムペアの2つめの原子コードである。
従って、分子内の原子の組み合わせを総当たりで調べ、原子コード・距離と、コードにExplainPairScore関数をかましたタプルを照合することで、コードに対応する原子ペアを容易に特定できるのである。
実装コード
以下はkey「689188」に該当するペア特定し、そのペア間に存在する原子のインデックス番号の一覧をresutlsに格納する処理である。molには可視化対象のRDKitのMOLオブジェクトが格納されている前提としている。
結果が複数ある場合もあるため、resutls はリストのリストになっている。
key = 689188
explained = Pairs.ExplainPairScore(key)
results = []
for i, atom1 in enumerate(mol.GetAtoms()):
code1 = Utils.GetAtomCode(mol.GetAtomWithIdx(i))
explain1 = Utils.ExplainAtomCode(code1)
for j, atom2 in enumerate(mol.GetAtoms()):
code2 = Utils.GetAtomCode(mol.GetAtomWithIdx(j))
explain2 = Utils.ExplainAtomCode(code2)
if j > i:
pathLength = len(AllChem.GetShortestPath(mol, i, j)) - 1
if ((explain1 == explained[0] and explain2 == explained[2] or
explain2 == explained[0] and explain1 == explained[2]) and
(pathLength == explained[1])):
results.append(AllChem.GetShortestPath(mol, i, j))
これを実行すると次のように、results変数に「689188」に該当する原子ペアを結ぶパス上にある原子のリストが複数分格納される。
[(0, 1, 4, 9, 8),
(2, 1, 4, 9, 8),
(3, 1, 4, 9, 8),
(8, 7, 6, 25, 26),
(8, 7, 6, 25, 27),
(8, 7, 6, 25, 28)]
該当箇所の原子が特定できたため、これを可視化してみよう。まず、可視化関数を用意する。
from rdkit.Chem.Draw import rdMolDraw2D
from IPython.display import SVG
from io import BytesIO
from PIL import Image
from cairosvg import svg2png
def generate_image(mol, highlight_atoms, size, isNumber=False):
view = rdMolDraw2D.MolDraw2DSVG(size[0], size[1])
tm = rdMolDraw2D.PrepareMolForDrawing(mol)
option = view.drawOptions()
if isNumber:
for atom in mol.GetAtoms():
option.atomLabels[atom.GetIdx()] = atom.GetSymbol() + str(atom.GetIdx() + 1)
view.DrawMolecule(tm, highlightAtoms=highlight_atoms)
view.FinishDrawing()
svg = view.GetDrawingText()
SVG(svg.replace('svg:', ''))
ret = svg2png(bytestring=svg.encode('utf-8'))
img = Image.open(BytesIO(ret))
return img
次に、上の関数にresultsの0番目のリストを与えて可視化してみる。
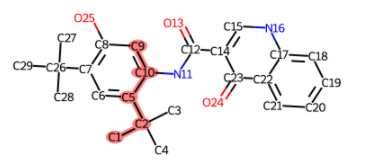
img = generate_image(mol, results[0], (400,200), True)
img
Jupyter Labであればこんな感じで表示されるはずだ。
これにより当該のリストが、ハイライトされている部分の両端であるC1とC9のアトムペアを示していることが一目に分かる。
同様にresults内の全リストについて色を変更する等して可視化すれば、分子中における「689188」に該当する全アトムペアを一目で表示させることができるはずだ。