はじめに
掲題の件を実現するにあたって、CycloPsというライブラリを見つけたので今回試してみた。
環境
- Python3.x
- RDKit 2020.X
- CycloPs
CycloPsとは
アミノ酸配列から環状ペプチドのバーチャルライブラリを生成してくれるツールである。
1次元のアミノ酸配列から、残基同士を結合することで、環状のペプチドを生成してくれる。
GUIとコマンドラインの両方で使える。
詳しくは参考文献の論文を参照のこと。
残基同士を結合するパターンは次の5種類である。それぞれ細かい条件があるようなので詳しくは論文を見てほしい。
- head-to-tail bond
アミノ酸配列のC端とN端を結合する。 - disulfide bond
システイン残基同士を結合する。 - side-chain to side-chain bond
側鎖同士を結合する。 - side-chain to N-terminal bond
側鎖とN端を結合する - side-chain to C-terminal bond
側鎖とC端を結合する
CycloPsのインストール
元々はPython2系で開発されているが、自分の環境だとRDkitがうまく動作しなかったため、3系でイントールする。
$ conda create -n cyclops python==3.6
$ conda activate cyclops
$ conda install -c rdkit rdkit
$ git clone https://github.com/fergaljd/cyclops.git
インストール後の作業
Python3系ではCycloPsが動作しないためソースを修正する必要がある。今回は、コマンドラインのみ使えればよい。
そこで PepLibGen/StructGen/StructGen.py を以下の通り修正することで、Python3でも動作するようにする。
修正内容
- print a, b のような記載を print(a,b) のように修正する。
- Except a, b のような記載を Except a as b のように修正する。
- 940行目あたりでエラーが出るため、以下のように修正する
# print(out,mollstr)
# print(out, '$$$$')
out.write(molstr)
out.write('$$$$')
out.write('\n')
- その他実行時エラーとなるところを、Python3で動作するよう修正する(手抜き)。
実行例
CPPsite 2.0 に登録されている環状ペプチドのアミノ酸配列と出力SDFを指定して実行する。
するとSDFに、5種類のパターンにより生成された環状ペプチドのリストが出力される。
以下実行例である。
python PepLibGen/StructGen/StructGen.py CSKSSDYQC hoge.sdf
実行結果例
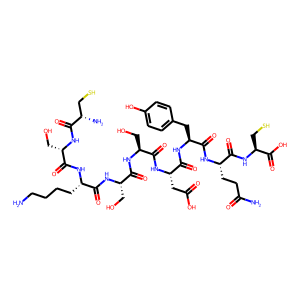
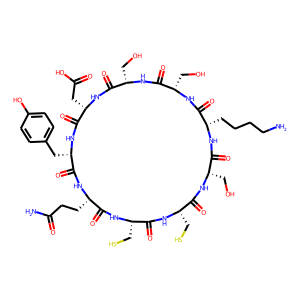
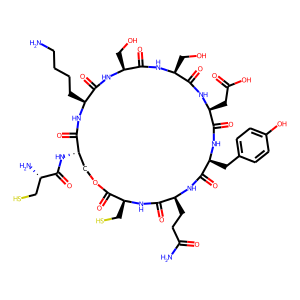
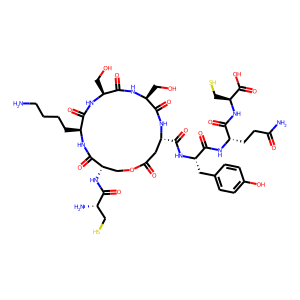
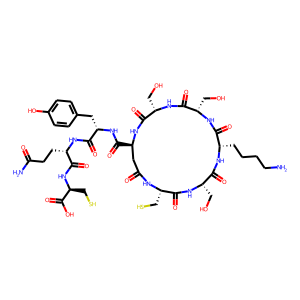
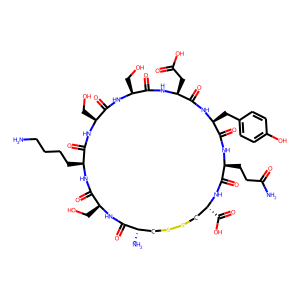
アミノ酸配列 "CSKSSDYQC" を指定して得られた6つの構造を以下に列挙する。
1番目
これは線形タイプである。環状ではないものも出力される仕様のようである。