まぁ、よくこういう事をするわけです。
library(tidyverse)
library(magrittr)
dat_pca <- iris %>%
cbind(., select_at(., 1:4) %>% prcomp(scale = T) %>% .$x) %>%
select(Species, everything()) %T>%
plot(col = .$Species, pch = 20)

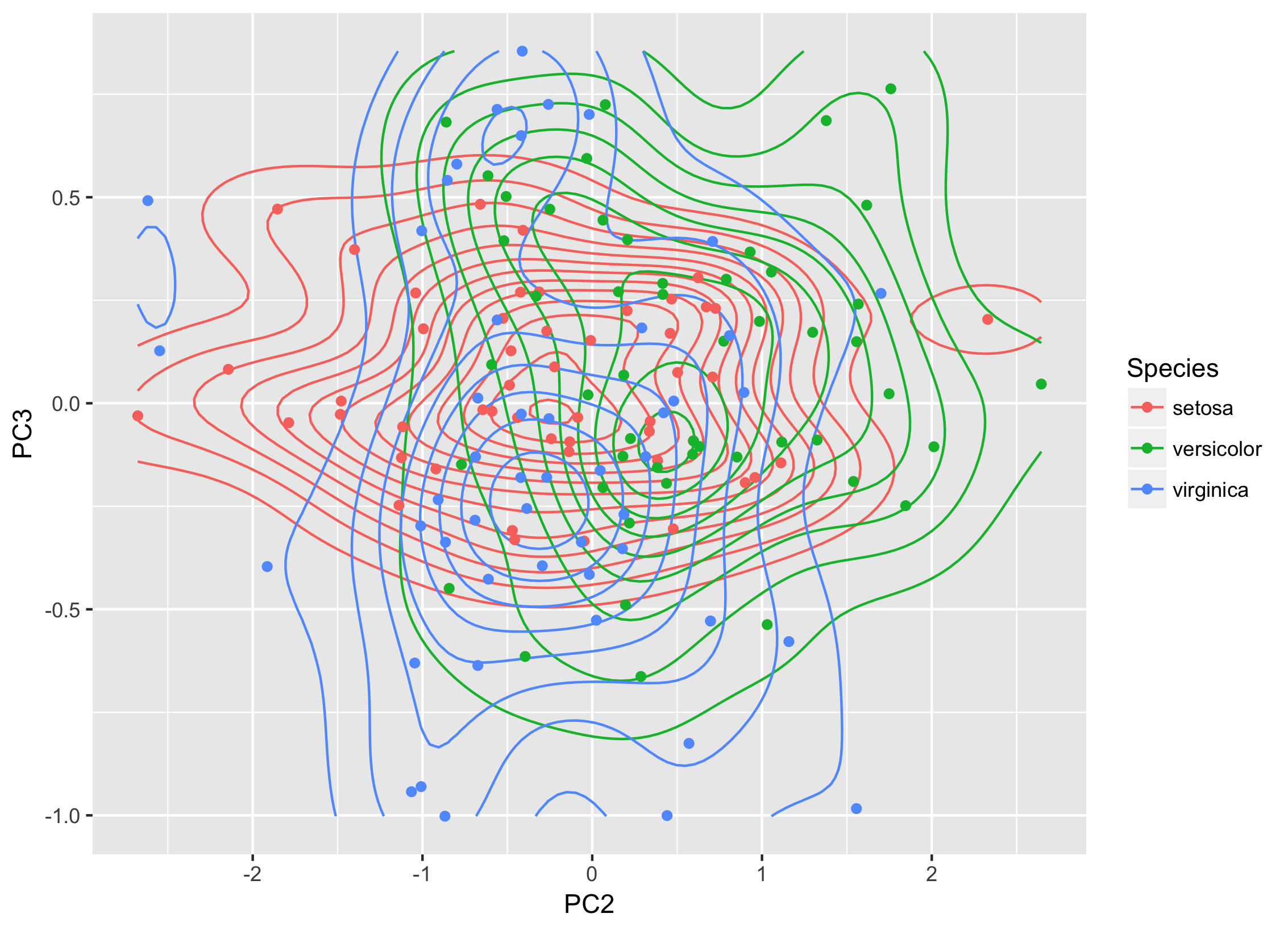
で、こーゆーのを描くわけですね。

ggplot(dat_pca, aes(PC1, PC2, color = Species))+
geom_density2d()+
geom_point()
これは各群の分布を可視化するために、プロットを正規分布をカーネル関数としてフィッティングしてるわけです。
カーネルについては以前にだいぶ書きましたのでここでは割愛。
で、このプロットを見ると、例えば青と緑は重なっている事は分かるんですが、具体的に新規の値に対して緑群・青群のどちらへの帰属率が高いのか、となると読み取るのが苦しいですね(ですね?)。
ま、いずれにせよ推定値を取り出せると何かと捗ります。
今回はこの平面をグリッドに区切って、網羅的にカーネル密度推定を行ないます。
2次元のカーネル密度推定を計算するRパッケージはいくつかありますが、高次元への拡張性が高いことから{ks}を愛用しています。
library(ks)
# 空間グリッドを準備
N <- 101
bound <- seq(-3, 3, length = N)
grid_kde <- expand.grid(PC1 = bound, PC2 = bound)
# Speciesでnestしておいて、mapでまとめてkdeを実行
dat_kde <- dat_pca %>%
select(Species, PC1, PC2) %>%
mutate_if(is.numeric, ~scale(.)) %>% # 主成分スコアをscaleしておきます。(グリッド範囲を統一したい)
group_by(Species) %>%
nest %>%
mutate(res_kde = map(data, ~kde(., eval.points = grid_kde)))%>% # kdeの実行
mutate(estimate = map(res_kde, ~ .$estimate %>% {. / max(.)} %>% c)) %>% # 以下、結果の整形
mutate(eval = map(res_kde, ~ .$eval.points),
eval = map2(.x = estimate, .y = eval, .f = ~ data.frame(estimate = .x, .y)))
ま、こんな感じで..。
> dat_kde
# A tibble: 3 x 5
Species data res_kde estimate eval
<fct> <list> <list> <list> <list>
1 setosa <tibble [50 × 2]> <S3: kde> <dbl [10,201]> <data.frame…
2 versicolor <tibble [50 × 2]> <S3: kde> <dbl [10,201]> <data.frame…
3 virginica <tibble [50 × 2]> <S3: kde> <dbl [10,201]> <data.frame…
こういう風になります。
nestを使うことで、元のデータがdataカラムに残っているのと、res_kdeにkdeの結果がS3クラスで全て保持されているのが良いです(後で立ち戻って調理できる)。
どうでもいいですが、<dbl [10,201]>って表記紛らわしくないですか?
<dbl [10201]> にして欲しい。
推定結果はevalカラムにまとまっていて、
> dat_kde$eval[[1]] %>% str
'data.frame': 10201 obs. of 3 variables:
$ estimate: num 7.92e-132 3.17e-121 4.28e-111 1.96e-101 3.04e-92 ...
$ PC1 : num -3 -2.94 -2.88 -2.82 -2.76 -2.7 -2.64 -2.58 -2.52 -2.46 ...
$ PC2 : num -3 -3 -3 -3 -3 -3 -3 -3 -3 -3 ...
こんな感じで、PC1, PC2にグリッド座標、その座標の推定値がestimateに入っています。
10201 obs.いいですね。うん。
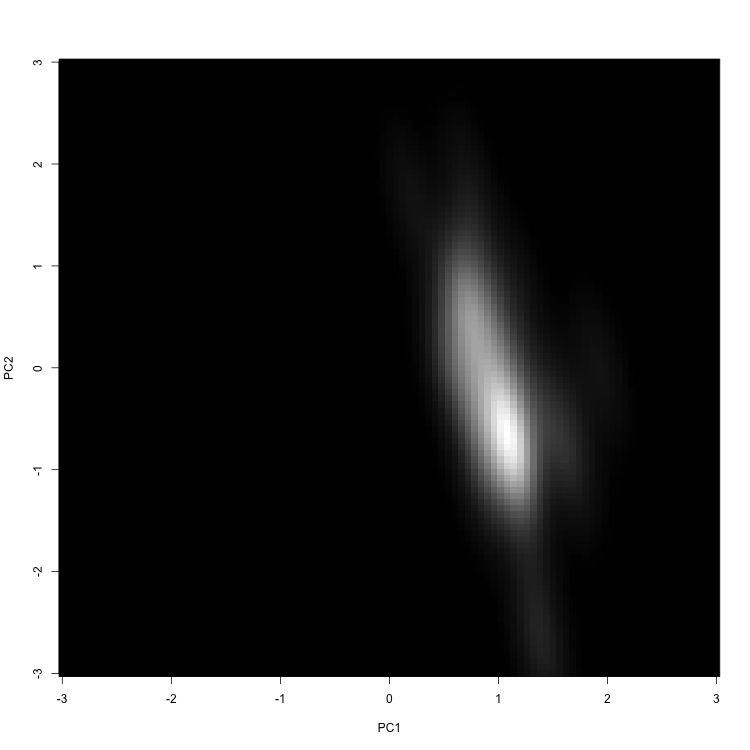
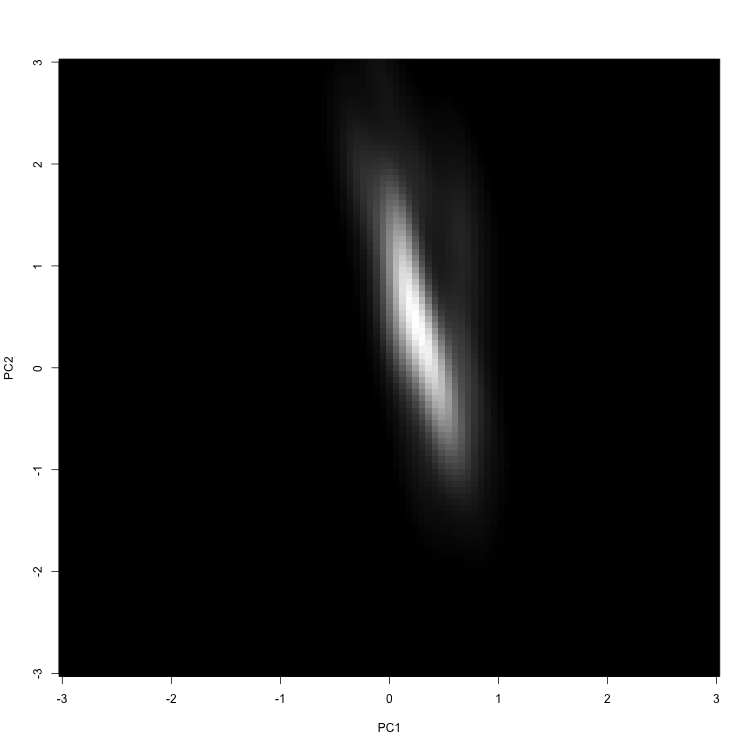
可視化は普通にimageを使って、
dat_i <- dat_kde$eval[[i]] %>% .$estimate %>% matrix(N, N)
image(bound, bound, dat_i, col = grey(seq(0, 1, by = 1/255)), xlab = "PC1", ylab = "PC2")
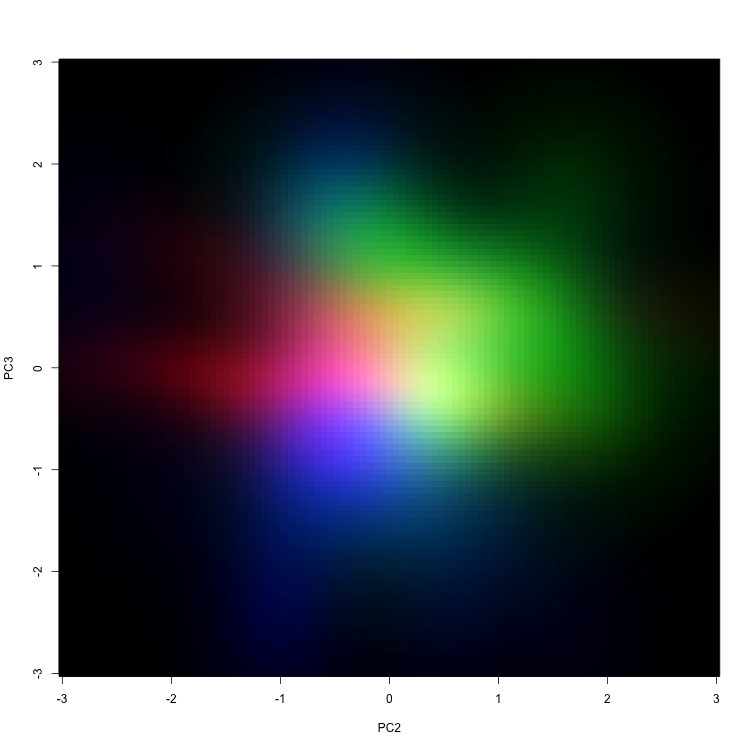
これを別個に保存して、それぞれにRGBを割り当てて結合する。
画像周りはmagickに移行中といいつつ、慣れたEBImageを使っちゃう。
library(EBImage)
a4 <- rgbImage(red = readImage("img_1.png") %>% channel("grey"),
blue = readImage("img_2.png") %>% channel("grey"),
green = readImage("img_3.png") %>% channel("grey"))
writeImage(a4, "marge.png")
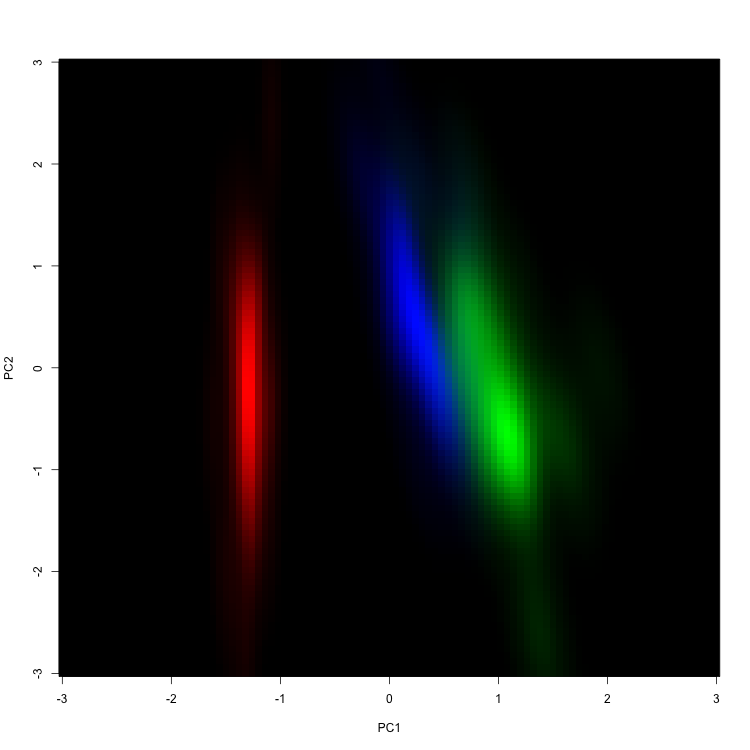
お。こんな感じですね。
分離が悪い平面では、
わぁキレイ。
って、苦労した?かいがあった様な無かった様な。