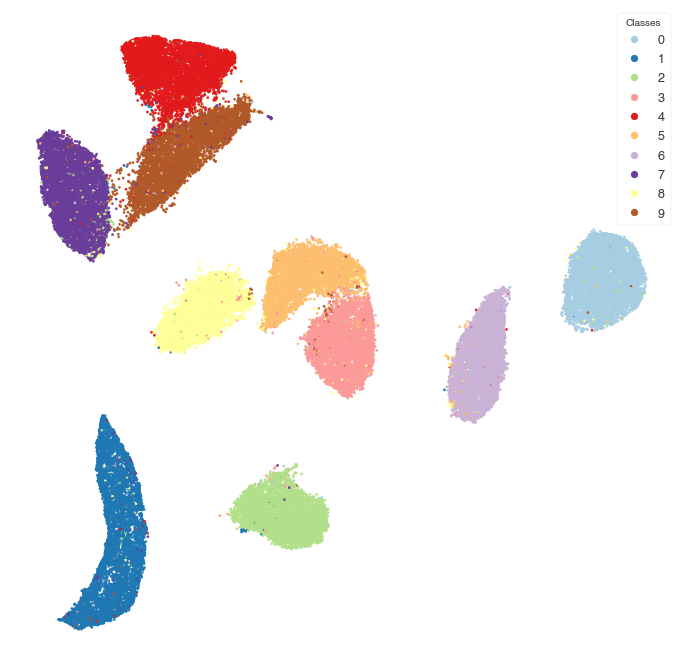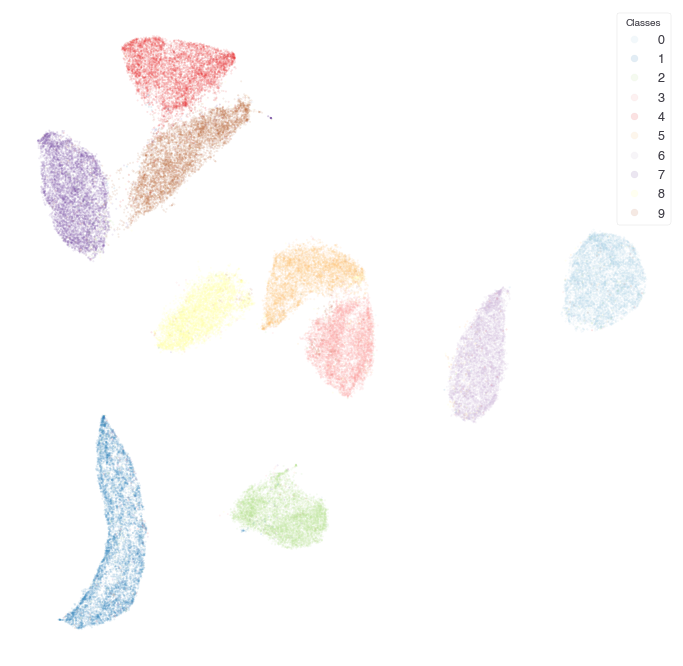大量のデータ点で散布図を描くと、あまりに密集してしまって、ある領域にどの程度のデータが存在するのかよくわからなくなる。
例として、手書き数字画像データセット(MNIST)をUMAPで二次元に圧縮した次のようなデータを考える。
import pandas as pd
df = pd.read_csv('./mnist_embedding.csv', index_col=0)
display(df)
| x | y | class | |
|---|---|---|---|
| 0 | 1.273394 | 1.008444 | 5 |
| 1 | 12.570375 | 0.472456 | 0 |
| 2 | -2.197421 | 8.652475 | 4 |
| 3 | -5.642218 | -4.971571 | 1 |
| 4 | -3.874749 | 5.150311 | 9 |
| ... | ... | ... | ... |
| 69995 | -0.502520 | -7.309745 | 2 |
| 69996 | 3.264405 | -0.887491 | 3 |
| 69997 | -4.995078 | 8.153721 | 4 |
| 69998 | -0.226225 | -0.188836 | 5 |
| 69999 | 8.405535 | -2.277809 | 6 |
70000 rows × 3 columns
xがX座標、yがY座標、classはそれぞれのラベル(0~9のどの数字を書いた画像か)。
普通にmatplotlibで散布図を描いてみる。ちなみに本筋ではないけど、最近追加されたlegend_elements関数によって、複数カテゴリの散布図はfor文をまわさずとも簡単に凡例が作れるようになった。
import matplotlib.pyplot as plt
fig, ax = plt.subplots(figsize=(12, 12))
sc = ax.scatter(df['x'], df['y'], c=df['class'], cmap='Paired', s=6, alpha=1.0)
ax.add_artist(ax.legend(*sc.legend_elements(), loc="upper right", title="Classes"))
plt.axis('off')
plt.show()
7万個の点がプロットされている。それぞれの数字ごとにクラスタが別れているのはいいんだけど、これだけデータサイズが大きいと点があまりに密集して、オーバーラップして塗り潰されてしまい、それぞれのクラスの中の構造がほとんど見えない。これをなんとかしたい。
解決策1: sizeやalphaを調整してがんばる
オーバーラップを避けるために、点のサイズを小さくする、あるいは点の透明度を調整して密度をわかりやすくする。試行錯誤が必要だし、必ずしも見やすくなるとは限らない。
fig, ax = plt.subplots(figsize=(12, 12))
sc = ax.scatter(df['x'], df['y'], c=df['class'], cmap='Paired', s=3, alpha=0.1)
ax.add_artist(ax.legend(*sc.legend_elements(), loc="upper right", title="Classes"))
plt.axis('off')
plt.show()
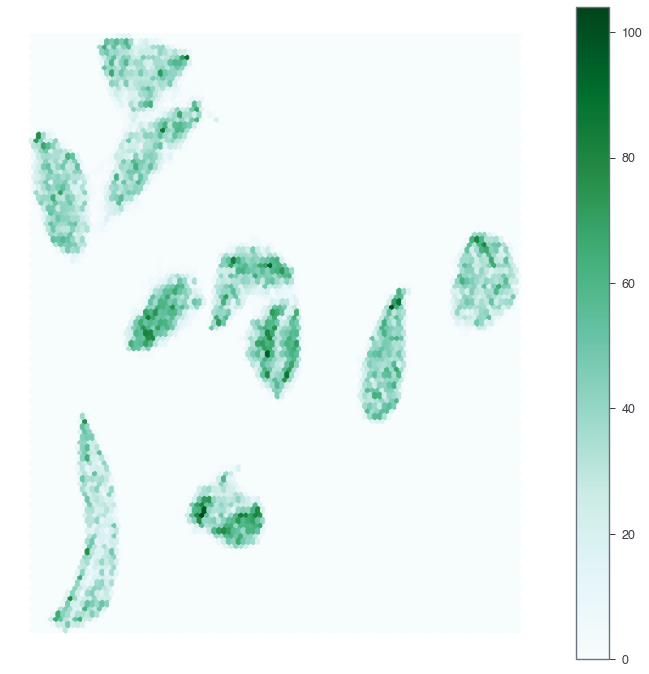
解決策2: Hexagonal Binning
これもよくやる方法。キャンバスを六角形のグリッドで敷き詰めて、それぞれの中に入るデータ点の数を集計して色の濃さで表現する。Pandasのプロット関数を使うのが簡単。
fig, ax = plt.subplots(figsize=(12, 12))
df.plot.hexbin(x='x', y='y', gridsize=100, ax=ax)
plt.axis('off')
plt.show()
解決策3: Datashaderを使う
応用が効いて使いやすい。使い方に慣れさえすれば。
Datashaderは大規模なデータセットについて「ラスタライズされたプロット」を高速に生成するライブラリ。
最初に出力する図の解像度(ピクセル数)を決めてしまってから、各ピクセルにデータを集計して、画像として出力する、という3つのステップで描画する。それぞれのステップで細かく調整ができるので自由度が高い。
各ステップは後述するけど、全部デフォルト設定でちぢめて書くと次のようになる。
import datashader as ds
from datashader import transfer_functions as tf
tf.shade(ds.Canvas().points(df,'x','y'))
各ステップの設定
Datashaderでは、
-
キャンバスを設定
-
集計関数の設定と計算
-
画像へ変換
の三つのステップでプロットを作る。以下、それぞれ説明。
1. キャンバスを設定
datashader.Canvasでキャンバスのもろもろを設定する。縦と横の解像度(ピクセル)、対数軸か否か、数値のレンジ(matplotlibでいうxlim, ylim)など。
canvas = ds.Canvas(plot_width=600, plot_height=600, # 縦横600ピクセル
x_axis_type='linear', y_axis_type='linear', # 'linear' or 'log'
x_range=(-10,15), y_range=(-15,10))
2. 集計関数の設定と計算
上で(600 x 600)ピクセルのキャンバスを作った。このピクセルひとつひとつについて、データをどのように集計するかをここで設定する。たとえば、ピクセルに入るデータ点のカウントに応じて色の濃さを変える、データ点がひとつでも入るか否かの二値にする、など。
たとえば上で設定したcanvas変数に対して以下のように、データフレーム、x軸座標(のカラム名)、y軸座標、集計関数を入れて計算を実行する。datashader.reductions.count関数の場合は、ピクセルに入るデータ点の個数をカウントする。
canvas.points(df, 'x', 'y', agg=ds.count())
<xarray.DataArray (y: 600, x: 600)>
array([[0, 0, 0, ..., 0, 0, 0],
[0, 0, 0, ..., 0, 0, 0],
[0, 0, 0, ..., 0, 0, 0],
...,
[0, 0, 0, ..., 0, 0, 0],
[0, 0, 0, ..., 0, 0, 0],
[0, 0, 0, ..., 0, 0, 0]], dtype=int32)
Coordinates:
* x (x) float64 -9.979 -9.938 -9.896 -9.854 ... 14.85 14.9 14.94 14.98
* y (y) float64 -14.98 -14.94 -14.9 -14.85 ... 9.854 9.896 9.938 9.979このように、(600 x 600)のサイズの行列で、データ点の個数をカウントした描画用のデータが生成された。
カウントではなくデータ点が入るか否かの二値で集計したい場合は、datashader.reductions.any関数を使って次のようにすればいい。
canvas.points(df, 'x', 'y', agg=ds.any())
<xarray.DataArray (y: 600, x: 600)>
array([[False, False, False, ..., False, False, False],
[False, False, False, ..., False, False, False],
[False, False, False, ..., False, False, False],
...,
[False, False, False, ..., False, False, False],
[False, False, False, ..., False, False, False],
[False, False, False, ..., False, False, False]])
Coordinates:
* x (x) float64 -9.979 -9.938 -9.896 -9.854 ... 14.85 14.9 14.94 14.98
* y (y) float64 -14.98 -14.94 -14.9 -14.85 ... 9.854 9.896 9.938 9.9793. 画像への変換
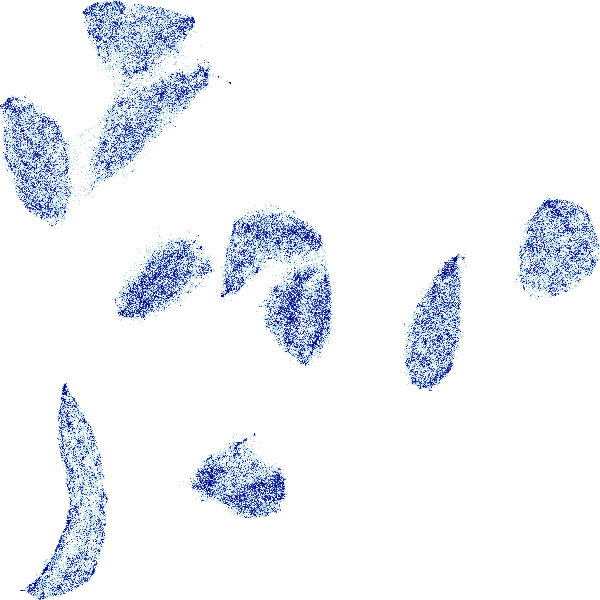
画像への変換はdatashader.transfer_functionsのshade関数を使う。shade関数の引数に、上で計算した集計済みの行列データを渡せばいい。また他にも様々なtransfer_functionsが用意されていて、画像出力の微調整ができる。ここではカウント集計した結果をset_background関数で白背景にして画像化してみる。
tf.set_background(tf.shade(canvas.points(df,'x','y', agg=ds.count())), 'white')
データ点の密度に応じて濃淡が表現されてだいぶ構造が見やすくなった。
同じようにデータ点が入るか否かの二値で集計した場合もやってみる。
tf.set_background(tf.shade(canvas.points(df,'x','y', agg=ds.any())), 'white')
他の補助データで集計する
これまではデータの座標情報だけ使って集計をしたけど、データ点それぞれになんらかのカテゴリのラベルがついていたり、連続値が割り振られていたりすることもよくある。
単にピクセルに入るデータ点を数えるだけだとそういった情報が反映されないので、それぞれに応じた特別な集計関数が存在する。
補助データがカテゴリカル変数の場合の集計
MNISTの場合は正解クラスのラベルがついているので、それでちゃんと色分けをしてプロットしたい。そのための集計関数として、datashader.reductions.count_catがある。この関数は、それぞれのラベルごとにピクセルに入るデータ点の個数をカウントする。つまりMNISTの場合は(600 x 600)の集計行列が10個できあがることになる。
count_catを使うためには、ラベルデータがPandasのcategory型である必要があるので(int型じゃダメ)、まずはデータフレームのラベル列をcategory型に変換する。
df['class'] = df['class'].astype('category')
count_catで集計する。countやanyの集計関数と違って、データフレームのどのカラムがラベルを表しているのか、カラム名を指定する必要がある。
agg = canvas.points(df, 'x', 'y', ds.count_cat('class'))
それぞれのラベルの色は、ラベルをキーとした辞書で定義しておく。冒頭で描画したときの図の色と合わせるためにmatplotlibから"Paired"の色を取り出す。辞書型のリスト内包を使うと簡単。
import matplotlib
color_key = {i:matplotlib.colors.rgb2hex(c[:3]) for i, c
in enumerate(matplotlib.cm.get_cmap('Paired', 10).colors)}
print(color_key)
{0: '#a6cee3', 1: '#1f78b4', 2: '#b2df8a', 3: '#fb9a99', 4: '#e31a1c', 5: '#fdbf6f', 6: '#cab2d6', 7: '#6a3d9a', 8: '#ffff99', 9: '#b15928'}
画像化してみる。各ピクセルの色は、ピクセルに入るデータ点のラベルの数に応じてそれぞれの色がミックスされて描画されるらしい。
tf.set_background(tf.shade(agg, color_key=color_key), 'white')
補助データが連続値の場合の集計
データ点のひとつひとつに、なんらかの連続値が紐づいていることがある。シングルセル解析とかで、数万の細胞の次元圧縮した図に関して、細胞ごとになんらかの遺伝子発現量で色の濃さを変える場合とか。
ピクセルには複数のデータ点が入るので、なんらかの方法で代表値を決めなくてはならない。そのための集計関数として、max, mean, modeなど簡単な統計量は一通り揃えてくれている。
MNISTは連続値補助データがないので、適当に作ってみる。わかりやすい量として、画像の中心のエリアの平均的な輝度を計算してみる。ゼロだと(画像の真ん中を線が走ることはあまりないから)暗くなり、1だと明るくなるはず。
data = pd.read_csv('./mnist.csv').values[:, :784]
data.shape
(70000, 784)
# 28 x 28のサイズの画像なので。
upper_left = 28 * 13 + 14
upper_right = 28 * 13 + 15
bottom_left = 28 * 14 + 14
bottom_right = 28 * 14 + 15
average_center_area = data[:, [upper_left, upper_right,
bottom_left, bottom_right]].mean(axis=1)
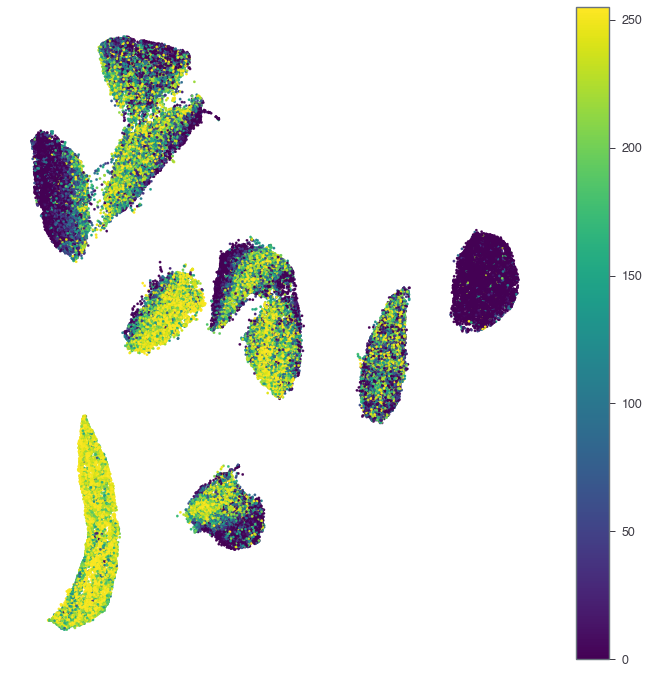
まずは普通にmatplotlibで描いてみる。
fig, ax = plt.subplots(figsize=(12, 12))
sc = ax.scatter(df['x'], df['y'], c=average_center_area, cmap='viridis',
vmin=0, vmax=255, s=6, alpha=1.0)
plt.colorbar(sc)
plt.axis('off')
plt.show()
やはりつぶれてしまってよくわからない。
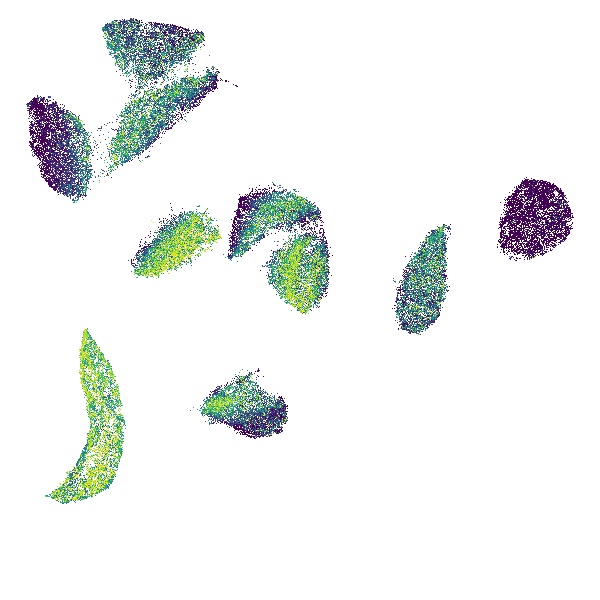
Datashaderに渡して、各ピクセルに入ったデータ点の「最大値」で塗り分けてみる。datashader.reductions.max関数で集計できる。
df['value'] = average_center_area
agg = canvas.points(df, 'x', 'y', agg=ds.max('value'))
tf.set_background(tf.shade(agg, cmap=matplotlib.cm.get_cmap('viridis')), 'white')
見やすくなった。matplotlibのscatterでサイズを小さく調整する場合とあまり変わらないかもしれないが、細かい試行錯誤なしでも綺麗に描画できるのが便利。
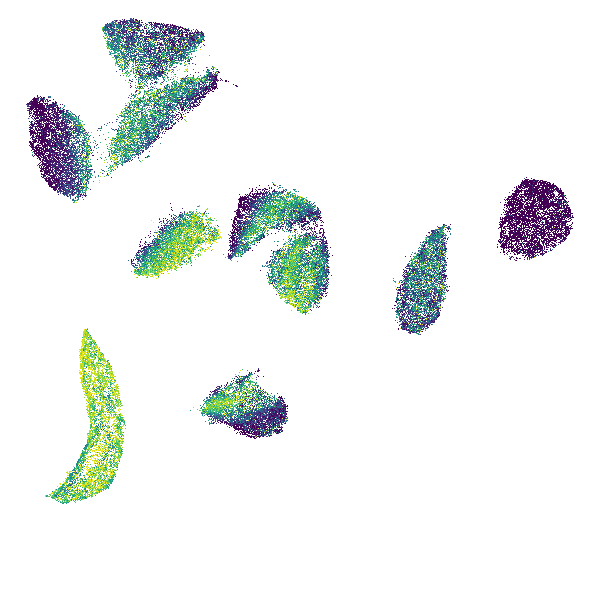
あとデータサイズが巨大でも高速なので、平均値で集計する場合はどうなるか、などいろいろと調整してみるのもストレスにならない。
agg = canvas.points(df, 'x', 'y', agg=ds.mean('value'))
tf.set_background(tf.shade(agg, cmap=matplotlib.cm.get_cmap('viridis')), 'white')