はじめに(この記事について)
Aidemyのデータ分析講座で学習した筆者が、実際のデータに対して試行錯誤しながらデータ分析にトライした記録です。本記事内で実施した手順は必ずしも適切なものではありませんのでご注意ください。
下記サイトで提供されている、オンライン性格診断の生データを使用させてもらいました。
実際に使用したのは、ビッグファイブ性格診断結果(BIG5)になります。
また、コード全体はGitHubにて確認可能です。
目次
1.目的
2.実行環境
3.データの確認と可視化
4.データの前処理
5.モデルの作成と予測
6.おわりに(まとめ)
1. 目的
感覚的に「性別」と「性格」には関係性はないように思える。
しかし、"男らしい性格"や"女らしい性格"という言葉が存在することから、
もしかしたら、なんらかの関係性を持つのかもしれない。
ということで今回は、ビッグファイブ診断の回答結果から、
性別を特定可能かどうかを検証してみることにする。
2. 実行環境
検証時の環境は下記の通り。
- GoogleColaboratory
- Python==3.10.12
- pandas==1.5.3
- numpy==1.23.5
- matplotlib==3.7.1
- seaborn==0.12.2
- scikit-learn==1.2.2
- imbalanced-learn==0.10.1
- tensorflow==2.12.0
3. データの確認と可視化
ダウンロードしたデータをGoogleドライブに保存し、データを取り込む。
# データセットはタブ区切りなのでsepに\tを指定して読み込む
raw_df = pd.read_csv("/content/drive/MyDrive/dataset/BIG5/data.csv", sep="\t")
print(f"データ件数:{raw_df.shape[0]}/列数:{raw_df.shape[1]}")
print(raw_df.dtypes)
データ件数:19719/列数:57
race int64
age int64
engnat int64
gender int64
hand int64
source int64
country object
E1 int64
E2 int64
E3 int64
E4 int64
E5 int64
E6 int64
E7 int64
E8 int64
E9 int64
E10 int64
N1 int64
N2 int64
N3 int64
N4 int64
N5 int64
N6 int64
N7 int64
N8 int64
N9 int64
N10 int64
A1 int64
A2 int64
A3 int64
A4 int64
A5 int64
A6 int64
A7 int64
A8 int64
A9 int64
A10 int64
C1 int64
C2 int64
C3 int64
C4 int64
C5 int64
C6 int64
C7 int64
C8 int64
C9 int64
C10 int64
O1 int64
O2 int64
O3 int64
O4 int64
O5 int64
O6 int64
O7 int64
O8 int64
O9 int64
O10 int64
dtype: object
提供元で説明されている内容
country以外は数値データである
race : 人種
age : 年齢(13歳以上である)
engnat : 母国語は英語か
gender : 性別
hand : 利き手
source : オンライン診断にアクセスした手段
country : オンライン診断にアクセスした国(ISOコード)
E1~O10はリッカート尺度(1:Disagree, 3:Neutral, 5:Agree)の診断項目
# 統計量の確認と入力ミスの確認
print(raw_df.describe(exclude="number"))
print("countryの欠損",raw_df["country"].isnull().sum(),"件")
lists = ["race","age","engnat","gender","hand","source"]
print(raw_df[lists].describe())
print()
for col in lists:
print(col,"入力ミス",(raw_df[col]==0).sum(),"件")
country
count 19710
unique 158
top US
freq 8753
countryの欠損 9 件
race age engnat gender hand \
count 19719.000000 1.971900e+04 19719.000000 19719.000000 19719.000000
mean 5.324205 5.076703e+04 1.365130 1.616918 1.130128
std 4.019064 7.121272e+06 0.488796 0.499122 0.413663
min 0.000000 1.300000e+01 0.000000 0.000000 0.000000
25% 3.000000 1.800000e+01 1.000000 1.000000 1.000000
50% 3.000000 2.200000e+01 1.000000 2.000000 1.000000
75% 8.000000 3.100000e+01 2.000000 2.000000 1.000000
max 13.000000 1.000000e+09 2.000000 3.000000 3.000000
source
count 19719.00000
mean 1.95228
std 1.50477
min 1.00000
25% 1.00000
50% 1.00000
75% 2.00000
max 5.00000
race 入力ミス 153 件
age 入力ミス 0 件
engnat 入力ミス 70 件
gender 入力ミス 24 件
hand 入力ミス 100 件
source 入力ミス 0 件
国情報の欠損値は、人種情報から推測できるかもしれない。
しかし種類が多く、また文字列情報のため数値化の必要がある。
今回は使用しない方向で考える。
ageに異常な値が存在している(max=$10^9$)。
グラフで確認する
import seaborn as sns
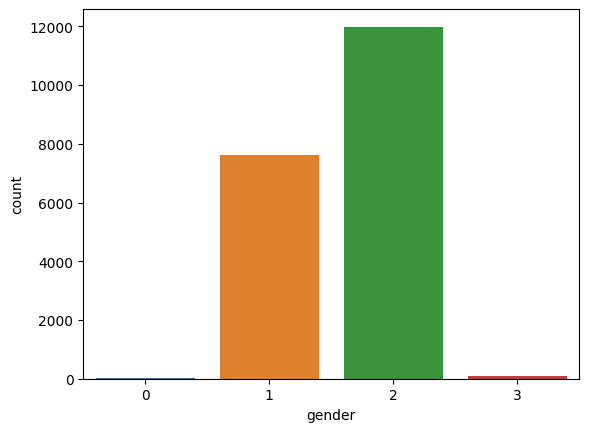
sns.countplot(x="gender", data=raw_df)
データの男女比は1:1.5。
入力ミス(=0)とその他(=3)が含まれている。入力ミスとその他を回答したデータは取り除く。
# ageについて可視化
# ひとまず100歳以下のみのデータの分布を確認
age_df = raw_df[raw_df["age"]<=100]
print(age_df["age"].describe()) # 統計量の再確認
print()
fig = sns.FacetGrid(age_df, col="gender", hue="gender", height=4)
fig.map(sns.histplot, "age", bins=30, kde=False)
count 19636.000000
mean 26.263801
std 11.567487
min 13.000000
25% 18.000000
50% 22.000000
75% 31.000000
max 100.000000
Name: age, dtype: float64
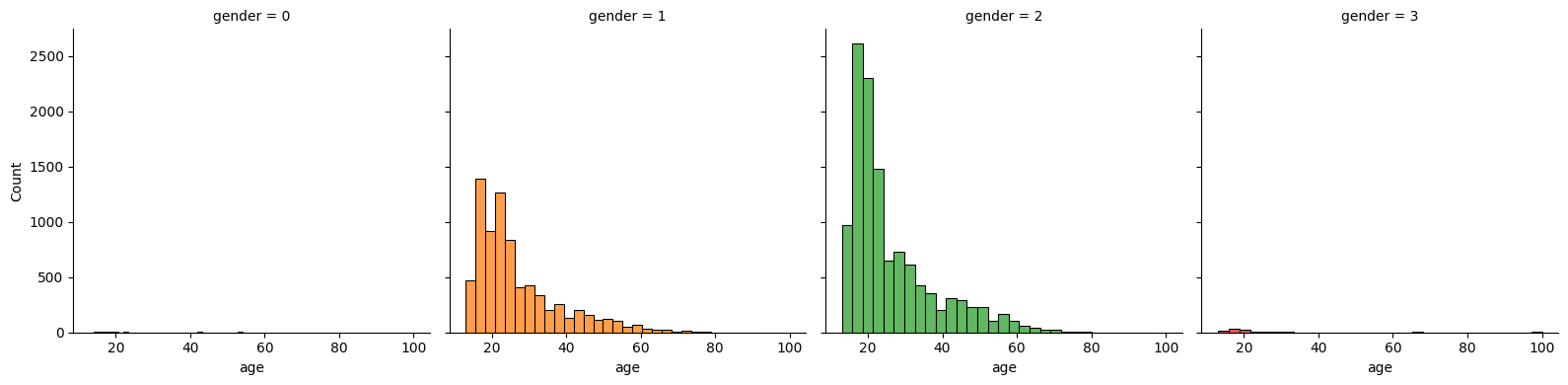
この診断の回答者のほとんどは20代で、80歳よりも大きい回答はほぼない。
80より大きい回答は平均値に置き換える方針で進める。
 |
 |
 |
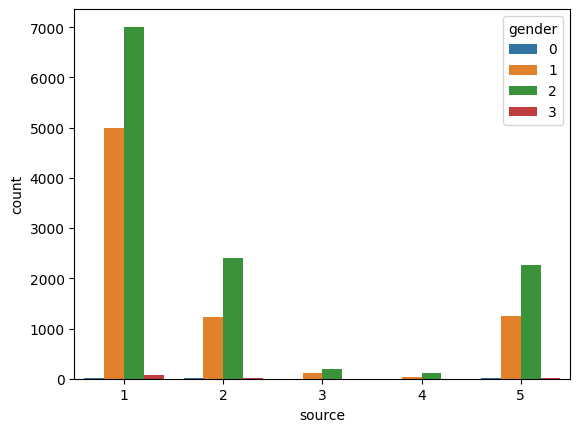 |
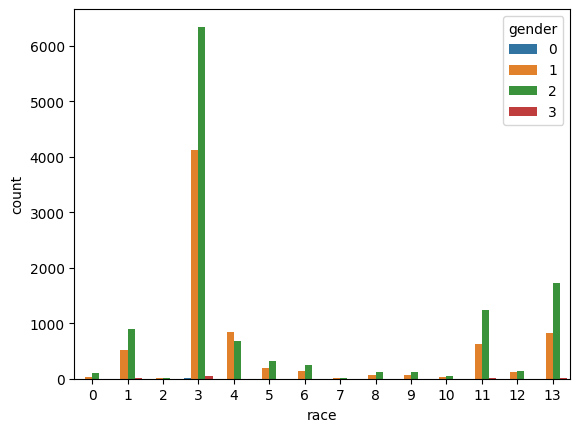
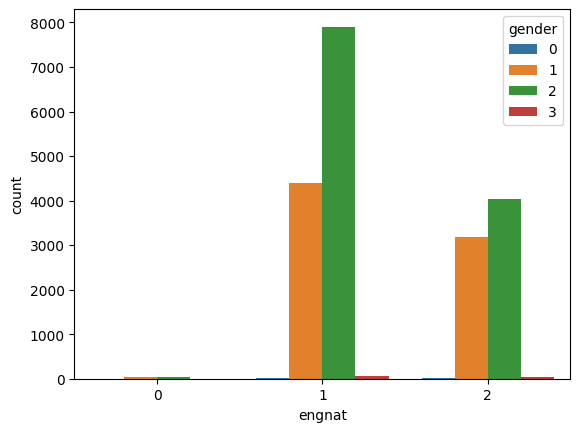
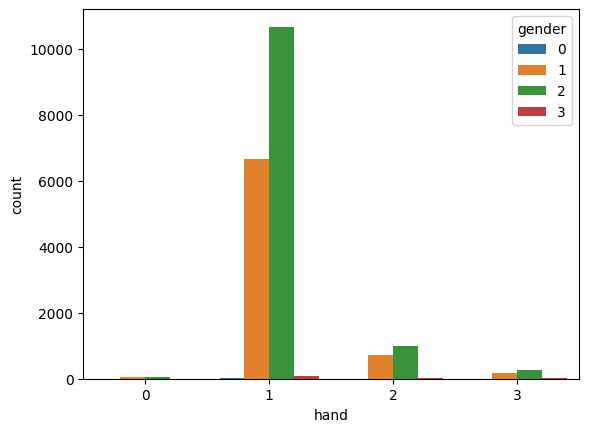
race(=人種)はカテゴリーが多い。今回はこれを予測に使わずに進める。
また、source(=アクセス手段)は性別との因果関係は考えにくいため使わない。
正しく回答されていないレコードが1件だけ存在したため、このレコードは予め削除した。
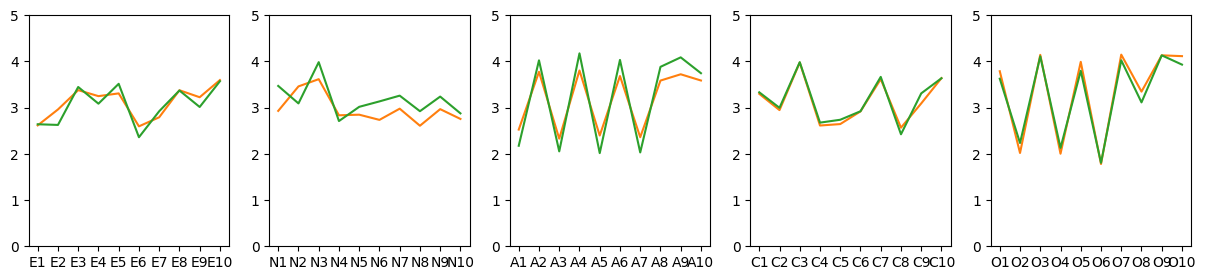
性別差による診断項目の平均値を可視化してみる。
# 診断項目
fig = plt.figure(figsize=(15,3))
for idx, cat in enumerate(["E","N","A","C","O"]):
col_list = [cat+str(idx) for idx in range(1,11)]
ax = fig.add_subplot(1,5,idx+1)
x = [label for label in col_list]
y1 = [(likert_df[likert_df["gender"]==1][label]).mean() for label in col_list]
y2 = [(likert_df[likert_df["gender"]==2][label]).mean() for label in col_list]
ax.plot(x, y1, color="tab:orange")
ax.plot(x, y2, color="tab:green")
ax.set_ylim(0,5)
plt.show()
性別差による診断項目の回答分布を可視化してみる。
# 性別毎の回答の度数分布表(母数が異なるので100%積み上げ棒グラフ)
for cat in ["E","N","A","C","O"]:
col_lists = [cat+str(idx) for idx in range(1,11)]
col_lists.append("gender")
# 診断項目と性別だけ抽出して、性別でグループ化
grp_df = likert_df[col_lists].groupby("gender")
# 各回答毎(1~5)に集計
male_cnts = grp_df.get_group(1).drop("gender", axis=1).apply(lambda x: x.value_counts()).fillna(0).transpose()
female_cnts = grp_df.get_group(2).drop("gender", axis=1).apply(lambda x: x.value_counts()).fillna(0).transpose()
# 100%積み上げのための処理
male_cnts = male_cnts.div(male_cnts.sum(axis=1), axis=0)
female_cnts = female_cnts.div(female_cnts.sum(axis=1), axis=0)
# カテゴリごとにグラフ描画
col_lists.remove("gender")
width = 0.35
ind = np.arange(len(male_cnts))
colors = ["red", "blue", "green", "yellow", "purple"]
fig, ax = plt.subplots(figsize=(8,4))
for pv, value in enumerate(male_cnts.columns):
ax.bar(ind - width/2, male_cnts[value], width, color=colors[pv], bottom=male_cnts.iloc[:, :pv].sum(axis=1), edgecolor="black")
ax.bar(ind + width/2, female_cnts[value], width, color=colors[pv], bottom=female_cnts.iloc[:, :pv].sum(axis=1), edgecolor="black")
ax.set_ylabel("Percentage")
ax.set_title(f"Category-{cat} : 100% stack bar")
ax.set_xticks(ind)
ax.set_xticklabels(male_cnts.index)
plt.tight_layout()
plt.show()
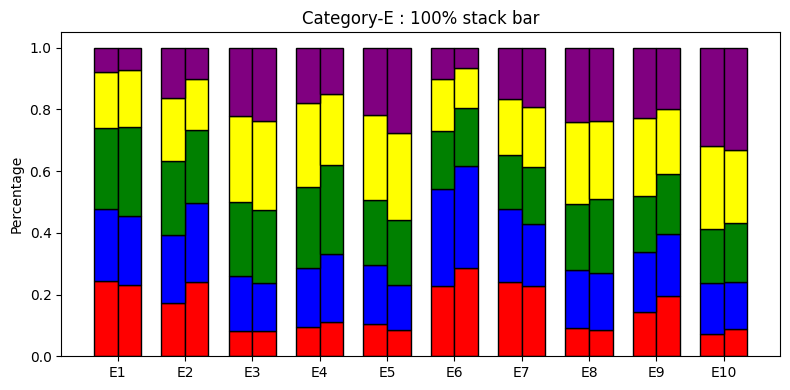
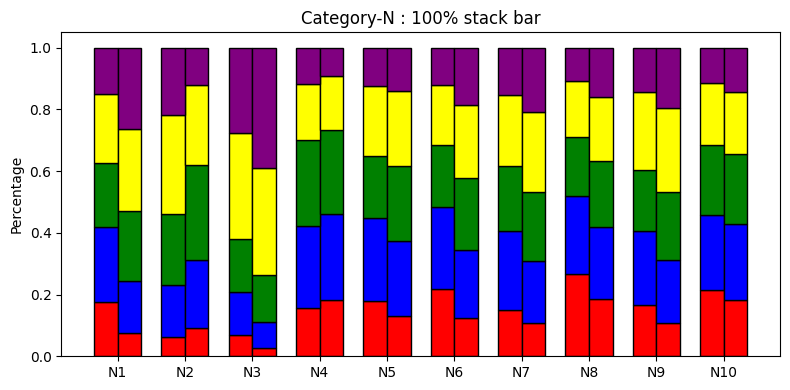
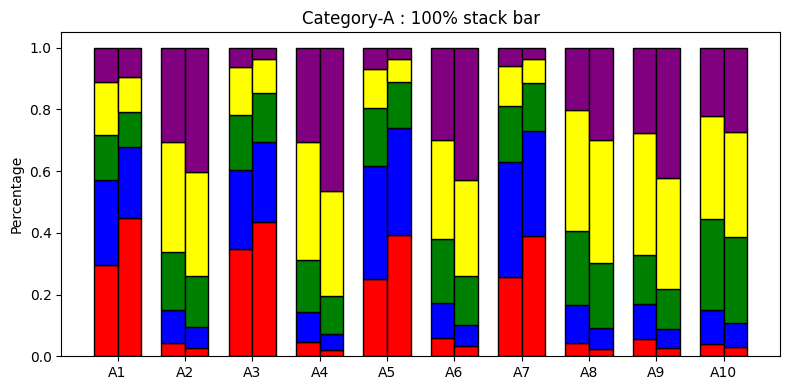
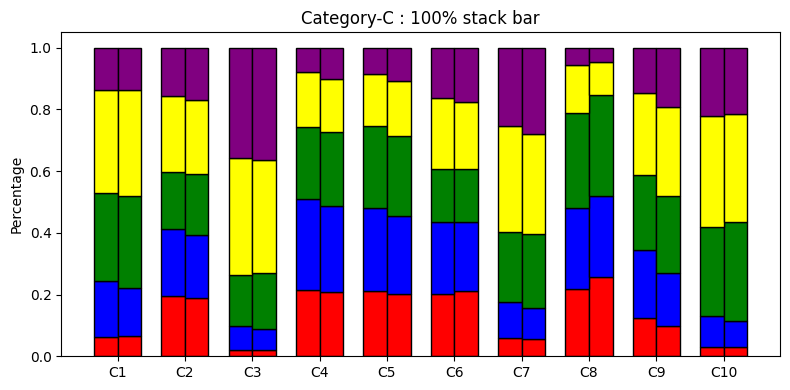
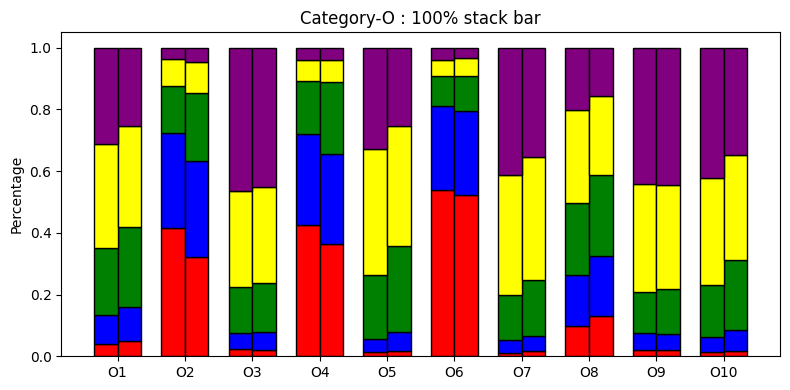
診断項目別に平均値を比較すると、全体的に差はないように見える。
回答分布で見ると、ところどころに特徴があるように見えるので性別の特定は可能かも?
4. データの前処理
データの確認と可視化で得られた知見から、データの前処理を行う。
- country:削除
- race:削除
- gender:入力ミス(=0)とその他(=3)は削除
- engnat:入力ミス(=0)は削除
- hand:入力ミス(=0)は削除
- source:削除
- age:80より大きい回答は平均値に置き換える
- E1~O10:入力ミス(=0)は削除 ※1件のみ
print(raw_df.shape, "元のデータサイズ")
# country, race, sourceの加工
del_columns = ["country","race","source"]
df = raw_df.drop(del_columns, axis=1)
print(df.shape, "country, race, sourceの加工後")
# genderの加工
df = df[(df["gender"] != 0) & (df["gender"] != 3)]
print(df.shape, "genderの加工後")
# engnat,handの加工
df = df[df["engnat"] != 0]
df = df[df["hand"] != 0]
print(df.shape, "engnat,handの加工後")
# 診断項目の加工
df = df[df["E1"] != 0]
print(df.shape, "診断項目の加工後")
# ageの加工
# 80以下の情報で平均値を算出
mean = df["age"].mean()
print(mean, "現時点でのage平均値")
mean = int(df[df["age"] <= 80]["age"].mean())
print(mean, "80以下の平均値")
print()
df['age'].where(df["age"] <= 80, mean, inplace=True)
# indexの振り直し
df.reset_index(drop=True, inplace=True)
print(df.tail(3)) #
print(df.shape, "最終データサイズ")
print()
# 年齢分布の再確認
print(df["age"].describe()) # 統計量の再確認
(19719, 57) 元のデータサイズ
(19719, 54) country, race, sourceの加工後
(19593, 54) genderの加工後
(19432, 54) engnat,handの加工後
(19431, 54) 診断項目の加工後
33.18830734393495 現時点でのage平均値
26 80以下の平均値
age engnat gender hand E1 E2 E3 E4 E5 E6 ... O1 O2 O3 O4 \
19428 16 2 1 1 2 5 4 5 5 5 ... 5 3 1 3
19429 16 1 1 1 1 4 2 3 2 4 ... 3 2 5 3
19430 35 1 1 1 2 3 1 5 3 3 ... 5 1 5 1
O5 O6 O7 O8 O9 O10
19428 4 1 1 5 5 5
19429 4 1 5 3 5 5
19430 4 1 5 5 5 5
[3 rows x 54 columns]
(19431, 54) 最終データサイズ
count 19431.000000
mean 26.235088
std 11.473508
min 13.000000
25% 18.000000
50% 22.000000
75% 31.000000
max 80.000000
Name: age, dtype: float64
5. モデルの作成と予測
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
from sklearn.linear_model import LogisticRegression
from sklearn.neighbors import KNeighborsClassifier
from sklearn.svm import SVC
from sklearn.ensemble import RandomForestClassifier
from sklearn.ensemble import AdaBoostClassifier
# 前処理を施したdfを訓練データとテストデータに分割
train = df.drop("gender",axis=1)
target = df["gender"]
# 訓練データの一部を分割し検証データを作成(検証データは2割)
X_train, X_val, y_train, y_val = train_test_split(train, target, test_size=0.2, shuffle=True, random_state=8)
# モデルを定義
classifiers = {
"ロジスティック回帰":LogisticRegression(max_iter=300), # デフォルト100だとWarning
"K近傍法":KNeighborsClassifier(),
"サポートベクターマシン(Linear)":SVC(kernel="linear",random_state=8),
"サポートベクターマシン(RBF)":SVC(kernel="rbf",random_state=8),
"ランダムフォレスト":RandomForestClassifier(random_state=8),
"AdaBoost":AdaBoostClassifier(random_state=8)
}
for key in classifiers.keys():
# 学習
classifiers[key].fit(X_train, y_train)
# 訓練データと検証データに対しての予測を行う
y_pred = classifiers[key].predict(X_train)
y_val_pred = classifiers[key].predict(X_val)
# 正答率を比較
print(f"訓練={accuracy_score(y_train, y_pred)} / 検証={accuracy_score(y_val, y_val_pred)} : {key}")
訓練=0.6937725167267113 / 検証=0.6827887831232313 : ロジスティック回帰
訓練=0.7570123520329387 / 検証=0.6405968613326473 : K近傍法
訓練=0.6964745239320638 / 検証=0.6812451762284538 : サポートベクターマシン(Linear)
訓練=0.7516083376222337 / 検証=0.6853614612811937 : サポートベクターマシン(RBF)
訓練=0.9998713329902214 / 検証=0.6786724980704913 : ランダムフォレスト
訓練=0.6919068450849202 / 検証=0.6807306405968613 : AdaBoost
サポートベクタマシン(RBF)での予測精度が一番高い結果となった。
しかし、非常に時間がかかることもわかった。
-
予測精度向上にあたっての試行錯誤
"gender"のデータは、カテゴリごとに大きく偏っているわけではないが、
その差を小さくすることで、予測精度を向上させられるのではないか?
という疑問から、ランダムサンプリングによる調整を実施した。Data_analytics_outcome.ipynb# オーバーサンプリングとアンダーサンプリングを試してみる from imblearn.over_sampling import RandomOverSampler from imblearn.under_sampling import RandomUnderSampler female_cnt_train = y_train.value_counts()[2] ros = RandomOverSampler(sampling_strategy={1:np.round(female_cnt_train/1.25).astype(int), 2:female_cnt_train}, random_state=8) X_train_os, y_train_os = ros.fit_resample(X_train, y_train) male_cnt_train = y_train.value_counts()[1] rus = RandomUnderSampler(sampling_strategy={1:male_cnt_train, 2:np.round(male_cnt_train*1.25).astype(int)}, random_state=8) X_train_us, y_train_us = rus.fit_resample(X_train, y_train) print(f"男性:{y_train.value_counts()[1]} / 女性:{y_train.value_counts()[2]} … 元データ") print(f"男性:{y_train_os.value_counts()[1]} / 女性:{y_train_os.value_counts()[2]} … オーバーサンプリング後") print(f"男性:{y_train_us.value_counts()[1]} / 女性:{y_train_us.value_counts()[2]} … アンダーーサンプリング語") print() print("<<オーバーサンプリング後>>") for key in classifiers.keys(): # 学習 classifiers[key].fit(X_train_os, y_train_os) # 訓練データと検証データに対しての予測を行う y_pred = classifiers[key].predict(X_train_os) y_val_pred = classifiers[key].predict(X_val) # 正答率を比較 print(f"訓練={accuracy_score(y_train_os, y_pred)} / 検証={accuracy_score(y_val, y_val_pred)} : {key}") print("<<アンダーサンプリング後>>") for key in classifiers.keys(): # 学習 classifiers[key].fit(X_train_us, y_train_us) # 訓練データと検証データに対しての予測を行う y_pred = classifiers[key].predict(X_train_us) y_val_pred = classifiers[key].predict(X_val) # 正答率を比較 print(f"訓練={accuracy_score(y_train_us, y_pred)} / 検証={accuracy_score(y_val, y_val_pred)} : {key}")Out男性:6049 / 女性:9495 … 元データ 男性:7596 / 女性:9495 … オーバーサンプリング後 男性:6049 / 女性:7561 … アンダーーサンプリング語 <<オーバーサンプリング後>> 訓練=0.6745070504944123 / 検証=0.6701826601492153 : ロジスティック回帰 訓練=0.7709320695102686 / 検証=0.6256753280164652 : K近傍法 訓練=0.6746825814756304 / 検証=0.6670954463596604 : サポートベクターマシン(Linear) 訓練=0.6975601193610672 / 検証=0.6959094417288397 : サポートベクターマシン(RBF) 訓練=1.0 / 検証=0.6789297658862876 : ランダムフォレスト 訓練=0.6769644842314668 / 検証=0.6755852842809364 : AdaBoost <<アンダーサンプリング後>> 訓練=0.6711241734019103 / 検証=0.6688963210702341 : ロジスティック回帰 訓練=0.750991917707568 / 検証=0.6246462567532801 : K近傍法 訓練=0.672226304188097 / 検証=0.6688963210702341 : サポートベクターマシン(Linear) 訓練=0.6939015429831007 / 検証=0.695137638281451 : サポートベクターマシン(RBF) 訓練=1.0 / 検証=0.6753280164651402 : ランダムフォレスト 訓練=0.6753857457751653 / 検証=0.6696681245176228 : AdaBoostいくつか増減を試した中で、25%ほど変化させた際に、
わずかだが1%の精度向上が確認できた。診断項目は合計で50件あることから、項目間に相関があるかもしれない。
もし、なんらかの相関があるのであれば、主成分分析などによる次元削減により、
精度向上が期待できるのではないかと思い試してみた。Data_analytics_outcome.ipynb# 主成分分析を用いた次元削減 from sklearn.decomposition import PCA from sklearn.preprocessing import StandardScaler scaler=StandardScaler() scaler.fit(train) X_scaled=scaler.transform(train) pca=PCA(n_components=0.95) # 95%の寄与率になるまで削減する pca.fit(X_scaled) train_pca=pca.transform(X_scaled) print(train_pca.shape) # 訓練データの一部を分割し検証データを作成(検証データは2割) X_train, X_val, y_train, y_val = train_test_split(train_pca, target, test_size=0.2, shuffle=True, random_state=8) for key in classifiers.keys(): # 学習 classifiers[key].fit(X_train, y_train) # 訓練データと検証データに対しての予測を行う y_pred = classifiers[key].predict(X_train) y_val_pred = classifiers[key].predict(X_val) # 正答率を比較 print(f"訓練={accuracy_score(y_train, y_pred)} / 検証={accuracy_score(y_val, y_val_pred)} : {key}")Out(19431, 45) 訓練=0.6883041688111168 / 検証=0.6722408026755853 : ロジスティック回帰 訓練=0.7563690169840452 / 検証=0.6354515050167224 : K近傍法 訓練=0.6906201749871334 / 検証=0.673012606122974 : サポートベクターマシン(Linear) 訓練=0.7951621204323212 / 検証=0.6827887831232313 : サポートベクターマシン(RBF) 訓練=1.0 / 検証=0.6637509647543093 : ランダムフォレスト 訓練=0.6917138445702522 / 検証=0.6647800360174942 : AdaBoostdf.corr()メソッドにより、下記のことがわかった。
・0.7以上の強い正の相関が1か所(N7とN8)
・0.4以上の弱い正の相関、負の相関がいくつか見られる
しかし、主成分分析による次元削減では精度向上を確認することはできなかった。age(=年齢)の大きさによる影響を小さくするため、年齢範囲を分けてOne-Hot Encodingする。
合わせて、engnat(=英語圏か)とhand(=利き手)も変換する。Data_analytics_outcome.ipynb# カテゴリカル変数への変換 df_catg = df.copy() df_catg["ageBand"] = pd.qcut(df_catg["age"], 4) df_catg = pd.get_dummies(df_catg, columns=["ageBand","engnat","hand"]) df_catg = df_catg.drop("age", axis=1) print(df_catg.columns) # 前処理を施したdfを訓練データとテストデータに分割 train = df_catg.drop("gender",axis=1) target = df_catg["gender"] # 訓練データの一部を分割し検証データを作成(検証データは2割) X_train, X_val, y_train, y_val = train_test_split(train, target, test_size=0.2, shuffle=True, random_state=8) for key in classifiers.keys(): # 学習 classifiers[key].fit(X_train, y_train) # 訓練データと検証データに対しての予測を行う y_pred = classifiers[key].predict(X_train) y_val_pred = classifiers[key].predict(X_val) # 正答率を比較 print(f"訓練={accuracy_score(y_train, y_pred)} / 検証={accuracy_score(y_val, y_val_pred)} : {key}")OutIndex(['gender', 'E1', 'E2', 'E3', 'E4', 'E5', 'E6', 'E7', 'E8', 'E9', 'E10', 'N1', 'N2', 'N3', 'N4', 'N5', 'N6', 'N7', 'N8', 'N9', 'N10', 'A1', 'A2', 'A3', 'A4', 'A5', 'A6', 'A7', 'A8', 'A9', 'A10', 'C1', 'C2', 'C3', 'C4', 'C5', 'C6', 'C7', 'C8', 'C9', 'C10', 'O1', 'O2', 'O3', 'O4', 'O5', 'O6', 'O7', 'O8', 'O9', 'O10', 'ageBand_(12.999, 18.0]', 'ageBand_(18.0, 22.0]', 'ageBand_(22.0, 31.0]', 'ageBand_(31.0, 80.0]', 'engnat_1', 'engnat_2', 'hand_1', 'hand_2', 'hand_3'], dtype='object')Out訓練=0.6946088522902728 / 検証=0.6840751222022124 : ロジスティック回帰 訓練=0.7551466803911477 / 検証=0.6398250578852586 : K近傍法 訓練=0.696152856407617 / 検証=0.6804733727810651 : サポートベクターマシン(Linear) 訓練=0.7420226453937211 / 検証=0.68356058657062 : サポートベクターマシン(RBF) 訓練=0.9998713329902214 / 検証=0.6838178543864163 : ランダムフォレスト 訓練=0.6919068450849202 / 検証=0.6807306405968613 : AdaBoostこちらも精度向上は見られない。
ageだけOne-Hot encodingし、主成分分析+オーバーサンプリングを組み合わせてみた。
一番精度が出ていたSVM(RBF)のみ確認。Data_analytics_outcome.ipynb# ageだけ変換してはどうか? df_Band = df.copy() df_Band["ageBand"] = pd.qcut(df_Band["age"], 4) df_Band = pd.get_dummies(df_Band, columns=["ageBand"]) df_Band = df_Band.drop("age", axis=1) # 前処理を施したdfを訓練データとテストデータに分割 train = df_Band.drop("gender",axis=1) target = df_Band["gender"] # 主成分分析による次元削減 scaler=StandardScaler() scaler.fit(train) X_scaled=scaler.transform(train) pca=PCA(n_components=0.95) # 95%の寄与率になるまで削減する pca.fit(X_scaled) train=pca.transform(X_scaled) # 訓練データの一部を分割し検証データを作成(検証データは2割) X_train, X_val, y_train, y_val = train_test_split(train, target, test_size=0.2, shuffle=True, random_state=8) # オーバーサンプリング female_cnt_train = y_train.value_counts()[2] ros = RandomOverSampler(sampling_strategy={1:np.round(female_cnt_train/1.25).astype(int), 2:female_cnt_train}, random_state=8) X_train, y_train = ros.fit_resample(X_train, y_train) # サポートベクタマシン(RBF)だけ model = SVC(random_state=8, kernel="rbf") # 学習 model.fit(X_train, y_train) # 訓練データと検証データに対しての予測を行う y_pred = model.predict(X_train) y_val_pred = model.predict(X_val) # 正答率を比較 print(f"訓練={accuracy_score(y_train, y_pred)} / 検証={accuracy_score(y_val, y_val_pred)} : サポートベクタマシン(RBF)")Out訓練=0.8094318647241238 / 検証=0.6933367635708773 : サポートベクタマシン(RBF)結果としては、初期状態にオーバーサンプリングを施したSVM(RBF)が最大(0.6959)となった。
データのレコード数は約20000件であるが、ディープラーニングで精度が出るか試す。
全結合層とCNNを適用した。・MLP
エポック数はいくつか試した中で一番精度の高かったものを選定
Data_analytics_outcome.ipynb# MLP # ageだけカテゴリカル変数への変換 df_catg = df.copy() df_catg["ageBand"] = pd.qcut(df_catg["age"], 4) df_catg = pd.get_dummies(df_catg, columns=["ageBand"]) df_catg = df_catg.drop("age", axis=1) # 性別カテゴリをゼロスタートに変換(男性:1→0, 女性:2→1) df_catg["gender"].where(df["gender"] != 1, 0, inplace=True) df_catg["gender"].where(df["gender"] != 2, 1, inplace=True) # 前処理を施したdfを訓練データとテストデータに分割 train = df_catg.drop("gender",axis=1) target = df_catg["gender"] # 訓練データの一部を分割し検証データを作成(検証データは2割) X_train, X_val, y_train, y_val = train_test_split(train, target, test_size=0.2, shuffle=True, random_state=8) model = tf.keras.Sequential([ tf.keras.layers.Input(X_train.shape[1]), tf.keras.layers.Dense(64, activation="relu"), tf.keras.layers.Dense(128, activation="relu"), tf.keras.layers.Dense(32, activation="relu"), tf.keras.layers.Dense(2, activation="softmax") ]) model.compile(optimizer=tf.keras.optimizers.Adam(learning_rate=0.01), loss="categorical_crossentropy", metrics=["accuracy"]) model.fit(X_train, to_categorical(y_train), batch_size=128, epochs=82, verbose=False) y_pred = np.argmax(model.predict(X_train), axis=1) y_val_pred = np.argmax(model.predict(X_val), axis=1) print(f"訓練={accuracy_score(y_train, y_pred)} / 検証={accuracy_score(y_val, y_val_pred)} : MLP")Out486/486 [==============================] - 1s 2ms/step 122/122 [==============================] - 0s 1ms/step 訓練=0.7036155429747812 / 検証=0.6833033187548238 : MLP・CNN
50件の診断項目に10件のデータを合わせ、6×10の画像と見立てて適用することを考えた。
(※診断項目の各カテゴリが10件の項目を持つため、10列としている。)
One-Hot Encodingで列を増やし、足りない分はdummy列として追加した。Data_analytics_outcome.ipynb# CNN # カテゴリカル変数の変換 df_catg = df.copy() df_catg["ageBand"] = pd.qcut(df_catg["age"], 4) df_catg = pd.get_dummies(df_catg, columns=["ageBand","engnat","hand"]) df_catg = df_catg.drop("age", axis=1) print(df_catg.shape) # 1列足りないので、dummy列(=0)を追加する df_catg["dummy"] = 0 # 性別カテゴリをゼロスタートに変換(男性:1→0, 女性:2→1) df_catg["gender"].where(df["gender"] != 1, 0, inplace=True) df_catg["gender"].where(df["gender"] != 2, 1, inplace=True) train = df_catg.drop("gender",axis=1) target = df_catg["gender"] # Conv2Dの入力フォーマットに合わせる(バッチサイズ, 縦サイズ, 横サイズ, チャンネル数) train = train.to_numpy().reshape(-1, 6, 10, 1) # データの正規化(最大値は5なので5で割る) train = train.astype("float32") / 5 # 訓練データの一部を分割し検証データを作成(検証データは2割) X_train, X_val, y_train, y_val = train_test_split(train, target, test_size=0.2, shuffle=True, random_state=8) print(X_train.shape) # Model / data parameters num_classes = 2 input_shape = (6, 10, 1) # convert class vectors to binary class matrices y_train = tf.keras.utils.to_categorical(y_train, num_classes) y_val = tf.keras.utils.to_categorical(y_val, num_classes)モデルの構築にあたっては、
1つ目はkerasのサイトの"mnist convnet"を参考にした。Data_analytics_outcome.ipynbmodels = tf.keras.Sequential( # サイトにあったサンプル [ tf.keras.Input(shape=input_shape), tf.keras.layers.Conv2D(32, kernel_size=(3, 3), padding='same', activation="relu"), tf.keras.layers.MaxPooling2D(pool_size=(2, 2)), tf.keras.layers.Conv2D(64, kernel_size=(3, 3), padding='same', activation="relu"), tf.keras.layers.MaxPooling2D(pool_size=(2, 2)), tf.keras.layers.Flatten(), tf.keras.layers.Dropout(0.5), tf.keras.layers.Dense(num_classes, activation="softmax"), ])2つ目のモデルは、VGGnetを模倣した。
Data_analytics_outcome.ipynbmodels = tf.keras.Sequential( # VGGnetを模倣したモデル [ tf.keras.Input(shape=input_shape), tf.keras.layers.Conv2D(128, kernel_size=(3, 3), padding='same', activation="relu"), tf.keras.layers.Conv2D(128, kernel_size=(3, 3), padding='same', activation="relu"), tf.keras.layers.MaxPooling2D(pool_size=(2, 2)), tf.keras.layers.Conv2D(64, kernel_size=(3, 3), padding='same', activation="relu"), tf.keras.layers.Conv2D(64, kernel_size=(3, 3), padding='same', activation="relu"), tf.keras.layers.MaxPooling2D(pool_size=(2, 2)), tf.keras.layers.Conv2D(32, kernel_size=(3, 3), padding='same', activation="relu"), tf.keras.layers.Conv2D(32, kernel_size=(3, 3), padding='same', activation="relu"), tf.keras.layers.Flatten(), tf.keras.layers.Dropout(0.5), tf.keras.layers.Dense(64, activation="relu"), tf.keras.layers.Dense(64, activation="relu"), tf.keras.layers.Dense(num_classes, activation="softmax"), ])3つ目のモデルは、自分なりに考察した結果を反映したモデル。
・入力サイズが小さいので、Poolingによる圧縮をすると中間サイズが小さぎるのでは?
・診断項目カテゴリ間の特徴を抽出するフィルタがあるとよいのでは?(縦方向)
・診断項目カテゴリ内の特徴を抽出するフィルタがあるとよいのでは?(横方向)
これらを元に、Pooling層では診断項目の特徴抽出するフィルタをかけるまでは圧縮せず、
フィルタサイズを調整しながら検証した。Data_analytics_outcome.ipynbmodels = tf.keras.Sequential( # 入力サイズが小さいのでカーネルサイズを調整してみる [ tf.keras.Input(shape=input_shape), # となりあう診断項目のフィルタ tf.keras.layers.Conv2D(64, kernel_size=(2, 4), padding='same', activation="relu"), tf.keras.layers.Conv2D(64, kernel_size=(2, 4), padding='same', activation="relu"), tf.keras.layers.MaxPooling2D(pool_size=(1, 1)), # 圧縮しない # 診断項目カテゴリごとの横方向フィルタ tf.keras.layers.Conv2D(64, kernel_size=(1, 4), padding='same', activation="relu"), tf.keras.layers.Conv2D(64, kernel_size=(1, 4), padding='same', activation="relu"), tf.keras.layers.MaxPooling2D(pool_size=(1, 2)), # 横方向の圧縮 # 診断項目を縦断するフィルタ tf.keras.layers.Conv2D(32, kernel_size=(5, 1), padding='same', activation="relu"), tf.keras.layers.Conv2D(32, kernel_size=(5, 1), padding='same', activation="relu"), tf.keras.layers.MaxPooling2D(pool_size=(2, 1)), # 縦方向の圧縮 tf.keras.layers.Flatten(), tf.keras.layers.Dropout(0.5), tf.keras.layers.Dense(480, activation="relu"), tf.keras.layers.Dense(480, activation="relu"), tf.keras.layers.Dense(num_classes, activation="softmax"), ])学習と予測にあたっては、GoogleColaboratoryのランタイムタイプを"GPU"にして実行した。
Data_analytics_outcome.ipynb# Train the model batch_size = 128 epochs = 16 for idx in range(len(models)): models[idx].summary() models[idx].compile(loss="categorical_crossentropy", optimizer="adam", metrics=["accuracy"]) models[idx].fit(X_train, y_train, batch_size=batch_size, epochs=epochs, validation_split=0.1) # Evaluate the trained model score = models[idx].evaluate(X_val, y_val, verbose=0) print("Test loss:", score[0]) print("Test accuracy:", score[1])Out# (略) 1つ目のモデル Test loss: 0.5904428958892822 Test accuracy: 0.6815024614334106 # (略) 2つ目のモデル Test loss: 0.6167956590652466 Test accuracy: 0.6856187582015991 # (略) 3つ目のモデル Test loss: 0.6064031720161438 Test accuracy: 0.6935940384864807エポック数はいくつか試した中で、精度の出たものを選定している。
6. おわりに(まとめ)
性格診断の回答から性別の特定は可能か?の結果としては、いまいちと言わざるをえませんでした。納期(期限)もあるため、検証は以上で終了します。予測精度0.7を超えられなかったのは少し残念でした。今回の検証では、SVMで一番よい精度を得られましたが、ディープラーニングでも同等の精度が得られたのは満足です。CNNを適用し、自分で考察したパラメータで成果を得られたのもうれしいことでした。
試行錯誤する中で、SVMは非常に時間がかかり、結果が出るまで正直わずらわしさがありました。が、CNNの検証にGPUを指定したところ、これが非常に高速で快適でした。CPUとGPUとの処理速度の違いを体験することができました。GoogleColaboratoryでは無料でGPUを活用させてもらえる制限があるようですが、手軽に試せるのはとてもありがたいと感じました。
今後に向けては、使用しなかったデータ(人種や国情報)を用いてさらなる精度向上を目指していければと思います。人種やお国柄といったところも性格に影響はあると考えられるので、そこから性別を特定する特徴も見えてくるかもしれません。
さらに、自然言語処理なども活用できればおもしろそうだと考えているので、データ分析を楽しんでいければと思います。
最後まで読んでいただきありがとうございました。