目的
Pythonを利用した解析や分析を行う際に必ず前処理が必要になるが、データ量やデータ格納形式によって処理時間がまちまちで、一度整理をしたかった。
環境
Python==3.6.5
numpy==1.14.3
pandas==0.23.0
検証対象データ
よくある時系列データを想定して以下の形式とした。
| カラム名 | 想定内容 | データ範囲 |
|---|---|---|
| dt | エポック時間(秒) | 15000000000から15100000000 |
| user | ユーザID | 1から100000 |
| data | 何らかのデータ | 1から100000000 |
また、時系列データの場合、元データは時系列で並んでいることが多いので、dtのみでソートをかけている。
コードで書くと以下のようなイメージ。
# データの作成
def create_data(data_size=10**7):
dt = np.random.randint(low=1.5*10**10, high=1.5*10**10+10**8, size=data_size, dtype=np.int64)
user = np.random.randint(low=1, high=10**5, size=data_size, dtype=np.int64)
data = np.random.randint(low=1, high=10**8, size=data_size, dtype=np.int64)
df = pd.DataFrame(
{'dt': dt,
'user': user,
'data': data,
}
)
# 時間のみでソート
df.sort_values('dt', inplace=True)
df.reset_index(drop=True, inplace=True)
return df
まずは1000万行のテストデータを作成。
検証
欠損値の補間とか特徴量の算出とかユーザ単位で処理したいユースケースを考慮して、テストデータとして作成したDataFrameからユーザ単位でデータを抽出するような処理を考える。
実際の処理はやりたいことによりさまざまだと思うので、分割したデータをリストに格納して、それを返すところに留めることにした。
Pandas組み込みのgroupbyを利用
def groupby_pd(_df):
result = []
for k, v in _df.groupby('user'):
result.append(v)
return result
テストデータ生成時にdtでソートがかかっているので、pandasのgroupbyは元のDataFrameの出現順が保持されるので、上記コードだけでuser, dtの2軸ソートがかかっていることになる。
Note that groupby will preserve the order in which observations are sorted within each group
df = create_data()
copy_df = df.copy()
%time res = groupby_pd(copy_df)
Wall time: 10.5 s
処理時間は10.5秒。まあ、普通にやるとこの方法になると思う。
とはいえ、もう少し速くできないか、というのがこの記事の趣旨。
事前にPandasでソートしてスライス(pandas)
ちょっと目線を変えて、事前に元データにuserとdtの2軸ソートをかけておいて、後でuserの切れ目でスライスすれば、実質的なgroupbyになるんじゃないかという案。
def groupby_presort_by_pandas(_df):
# user, dtの順で2軸ソート
_df.sort_values(['user', 'dt'], inplace=True)
# 区切りとなるインデックスを取得
start_index, end_index = split_index(_df['user'].values)
result = []
for s, e in zip(start_index, end_index):
result.append(_df.iloc[s:e])
return result
def split_index(arr):
# arrの区切りがユーザの区切り
uniq_users, split_index = np.unique(arr, return_index=True)
# start_indexはユニークな要素が発生するインデックス
start_index = split_index.tolist()
# のちにインデックスでスライスすることを考慮して、先頭要素を削除して、最後に配列サイズを追加
end_index = split_index[1:].tolist()
end_index.append(len(arr))
return start_index, end_index
copy_df = df.copy()
%time res = groupby_presort_by_pandas(copy_df)
Wall time: 16.2 s
結果は16.2秒。うん、遅くなった。
理由としては、DataFrame#sort_values()の2軸ソートの遅さとDataFrameのilocのアクセスの遅さにあると思われる。
事前にnumpyでソートしてスライス(numpy.lexsort)
というわけで、2軸ソートを高速にするために、numpy#lexsort()を利用する。
この関数の引数設定はかなり変態っぽくて、1st_key, 2nd_keyがあった場合np.lexsort([2nd_key, 1st_key])のように指定する。
戻り値はソートされた状態になるようなインデックスが返されるので、これを全てのnumpy.arrayに適用してあげれば、ソートされた状態となる。
def groupby_presort_by_numpy(_df):
# user, dtの順で2軸ソート
arr_user = _df['user'].values
arr_dt = _df['dt'].values
arr_data = _df['data'].values
sorted_index = np.lexsort([arr_dt, arr_user])
# ソートされたインデックスにより元のnp.arrayを並び替え
sorted_arr_user = arr_user[sorted_index]
sorted_arr_dt = arr_dt[sorted_index]
sorted_arr_data = arr_data[sorted_index]
sorted_df = pd.DataFrame(
{'dt':sorted_arr_dt,
'user':sorted_arr_user,
'data':sorted_arr_data
}
)
# sorted_arr_userの区切りがユーザの区切り
start_index, end_index = split_index(sorted_arr_user)
result = []
for s, e in zip(start_index, end_index):
result.append(sorted_df.iloc[s:e])
return result
copy_df = df.copy()
%time res = groupby_presort_by_numpy(copy_df)
Wall time: 8.94 s
結果は8.94秒。というわけで多少速くなった。
ただ、よくよく考えてみると一度numpy.arrayに変換したデータをなんでわざわざDataFrameに変換しないといけないんだという話になるわけです。
そこで、ユーザごとのデータを辞書形式(key:カラム名、value:numpy.array)にして、それをlistに格納するという方式を考えます。
def groupby_presort_fast_by_numpy(_df):
# user, dtの順で2軸ソート
arr_user = _df['user'].values
arr_dt = _df['dt'].values
arr_data = _df['data'].values
sorted_index = np.lexsort([arr_dt, arr_user])
# ソートされたインデックスにより元のnp.arrayを並び替え
sorted_arr_user = arr_user[sorted_index]
sorted_arr_dt = arr_dt[sorted_index]
sorted_arr_data = arr_data[sorted_index]
# sorted_arr_userの区切りがユーザの区切り
start_index, end_index = split_index(sorted_arr_user)
result = []
for s, e in zip(start_index, end_index):
result.append(
{'dt':sorted_arr_dt[s:e],
'user':sorted_arr_user[s:e],
'data':sorted_arr_data[s:e]
}
)
return result
copy_df = df.copy()
%time res = groupby_presort_fast_by_numpy(copy_df)
Wall time: 2.57 s
2.57秒。というわけで圧倒的に速くなりました。
事前にuser, dtの順でソートされていた場合は?
すでに前処理が済んでいて、user, dtの順で2軸ソートがかかっているという状態でデータをもらえることもあると思います。
Pandas組み込みのgroupbyを利用
copy_df = df.copy()
sorted_copy_df = copy_df.sort_values(['user', 'dt'])
sorted_copy_df.reset_index(drop=True, inplace=True)
%time res = groupby_pd(sorted_copy_df)
Wall time: 9.44 s
9.44秒。そう、実はそんなに速くならない。
numpyを利用した高速化版(ソート除去)
ソート済みという前提なので、ソートに関連するところを省略できる。
def groupby_np_for_sorted_data(_df):
# user, dtの順で2軸ソート
arr_user = _df['user'].values
arr_dt = _df['dt'].values
arr_data = _df['data'].values
# arr_userの区切りがユーザの区切り
start_index, end_index = split_index(arr_user)
result = []
for s, e in zip(start_index, end_index):
result.append(
{'dt':arr_dt[s:e],
'user':arr_user[s:e],
'data':arr_data[s:e]
}
)
return result
%time res = groupby_np_for_sorted_data(sorted_copy_df)
Wall time: 544 ms
おお、0.54秒。1秒を切った。
やっていることは1000万レコードの区切りを探して、スライスするだけなので、だいぶ高速に処理できるようになる。
データ量の呪い
よし、それじゃなんでもnumpy#lexsort()で事前にソートしてスライスするぜ、という方針に倒すと大体事故が起こる。
データ量に依存して、処理時間が変わるのが、この方式の厄介なところなのです。
データ量と条件をいくつか変えてみて、試してみた。先ほどの結果と微妙に処理時間が違うのはスクリプトで一気に回しなおしたため。
単位は全て秒で、カッコ内は事前にソートされたカラム。
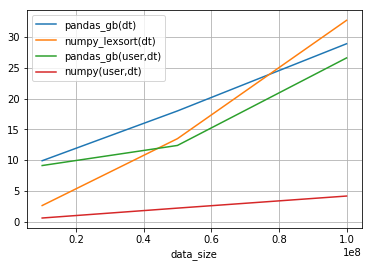
| data_size | pandas_gb(dt) | numpy_lexsort(dt) | pandas_gb(user,dt) | numpy(user,dt) |
|---|---|---|---|---|
| 10,000,000 | 9.92 | 2.65 | 9.14 | 0.626 |
| 50,000,000 | 18.00 | 13.50 | 12.40 | 2.230 |
| 100,000,000 | 28.90 | 32.70 | 26.60 | 4.190 |
x軸がデータサイズ(1000万、5000万、1億)、y軸は処理時間(秒)、グラフ凡例のカッコ内は事前にソートされているカラム。
1億レコードを処理した場合、dtのみがソートされた状態だとpandas.groupbyに比較してnumpy+lexsortによる処理のほうが遅くなる。
事前にuser, dtでソートされた形式のgroupbyはpandas.groupbyはレコード数に応じて、急激に性能が劣化する。
結論
- 前処理の方法はいくつもあるが、保守性と性能とデータフォーマットによって選択する必要がある。
- 時系列データの前処理をする際には事前にデータの格納仕様を決めておくと、処理が高速化することもある。