ChemTHEATREに収録されたデータを活用して有機フッ素化合物の濃度を可視化してみる
有機フッ素化合物とは
親水性と親油性の両方の性質を持ち,撥水剤や消化剤,油剤,エッチング剤など幅広い用途で使用されています。
有機フッ素化合物に関するわかりやすい解説は,NHKのクローズアップ現代のHPを参照。
https://www.nhk.or.jp/gendai/articles/4280/index.html
その有害性から,有機フッ素化合物のうちペルフルオロオクタン酸(Perfluorooctanoic acid: PFOA)は2019年からその製造・使用・輸出入が国際的に禁止され,ペルフルオロオクタンスルホン酸(Perfluorooctanesulfonate: PFOS)は2009年から製造・使用・輸出入が制限されています。
有機フッ素化合物の基準値等
近年,両物質が井戸水から検出されたり,河川に流出するなど,問題となっています。
これを受け,厚生労働省は水質管理目標設定項目にPFOSとPFOAを追加しました(令和2年4月1日施行)。水道水質基準の目標値をPFOSとPFOAの合算値で50 ng/Lとしています。
https://www.mhlw.go.jp/stf/seisakunitsuite/bunya/topics/bukyoku/kenkou/suido/kijun/index.html
また,環境省は水環境におけるPFOSとPFOAの全国存在量調査を実施し,「水質汚濁に係る人の健康の保護に関する環境基準等の施行について(通知)」(令和2年5月28日付け)で,水環境に係る暫定的な目標値としてPFOSとPFOAの合算値で50 ng/Lを設定しました。
令和元年度PFOA及びPFOA全国存在量調査結果
https://www.env.go.jp/press/108091.html
ちなみに,令和2年6月に厚生労働省が公開した浄水場を対象とした調査結果では,水道水の暫定目標値であるPFOS+PFOA 50 ng/Lを超過したところはありませんでした。
https://www.mhlw.go.jp/content/10900000/000638290.pdf
ChemTHEATREから有機フッ素化合物の環境中濃度データを取得する
ChemTHEATREでは環境中の化学物質濃度のモニタリングデータを公開しており,有機フッ素化合物のデータも収録しています。実際に,PFOSやPFOAがどのくらいの濃度で検出されたのか,また,その合算値は基準値と比較してどの程度高い,あるいは低いのか,実際に見てみましょう。
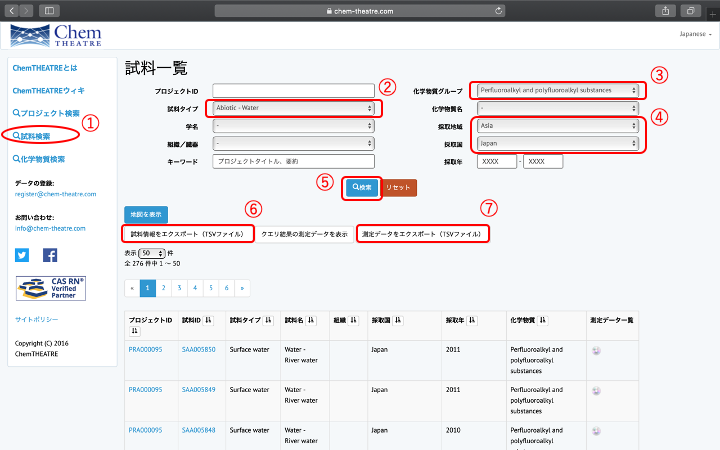
- ChemTHEATREのメニューバーから「試料検索」を選択する。
- 「試料タイプ」から「Abiotic - Water」(非生物 - 水)を選択する。
- 「化学物質グループ」から「Perfluoroalkyl and polyfluoroalkyl substances」を選択する。
- 「採取地域」から「Asia」を選択し,採取国を「Japan」にする。
- 「Search」ボタンをクリックすると,条件に合致する試料の一覧が出力される。
- 「Export samples」で試料の情報が,「Export measured data」で目的の化学物質の測定値が,タブ区切りのテキストファイルとして出力する。
エクスポートしたファイルを任意のディレクトリに保存して,解析に使用してください。
エクスポートしたデータを整形する
必要なライブラリを読み込む。
import pandas as pd
化学物質の測定値の情報を読み込む。
data_file = "measureddata_20200521044415.tsv"
data = pd.read_csv(data_file, delimiter="\t")
data
| MeasuredID | ProjectID | SampleID | ScientificName | ChemicalID | ChemicalName | ExperimentID | MeasuredValue | AlternativeData | Unit | Remarks | RegisterDate | UpdateDate | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 81245 | PRA000095 | SAA005816 | Water | CH0000362 | PFBS | EXA000001 | 0.00100 | <1.00E-3 | μg/L | NaN | 2019/7/26 | 2019/7/26 |
| 1 | 81246 | PRA000095 | SAA005817 | Water | CH0000362 | PFBS | EXA000001 | 0.00100 | <1.00E-3 | μg/L | NaN | 2019/7/26 | 2019/7/26 |
| 2 | 81247 | PRA000095 | SAA005818 | Water | CH0000362 | PFBS | EXA000001 | 0.00100 | NaN | μg/L | NaN | 2019/7/26 | 2019/7/26 |
| 3 | 81248 | PRA000095 | SAA005819 | Water | CH0000362 | PFBS | EXA000001 | 0.00100 | <1.00E-3 | μg/L | NaN | 2019/7/26 | 2019/7/26 |
| 4 | 81249 | PRA000095 | SAA005820 | Water | CH0000362 | PFBS | EXA000001 | 0.00100 | <1.00E-3 | μg/L | NaN | 2019/7/26 | 2019/7/26 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 3087 | 48619 | PRA000060 | SAA003645 | Water | CH0000793 | THPFOS | EXA000001 | 0.00030 | NaN | μg/L | NaN | 2018/2/9 | 2018/6/8 |
| 3088 | 48620 | PRA000060 | SAA003646 | Water | CH0000793 | THPFOS | EXA000001 | 0.00008 | NaN | μg/L | NaN | 2018/2/9 | 2018/6/8 |
| 3089 | 48621 | PRA000060 | SAA003647 | Water | CH0000793 | THPFOS | EXA000001 | 0.00159 | NaN | μg/L | NaN | 2018/2/9 | 2018/6/8 |
| 3090 | 48622 | PRA000060 | SAA003648 | Water | CH0000793 | THPFOS | EXA000001 | 0.00188 | NaN | μg/L | NaN | 2018/2/9 | 2018/6/8 |
| 3091 | 48623 | PRA000060 | SAA003649 | Water | CH0000793 | THPFOS | EXA000001 | 0.00070 | NaN | μg/L | NaN | 2018/2/9 | 2018/6/8 |
3092 rows × 13 columns
続いて,試料の情報を読み込む。
sample_file = "samples_20200521044410.tsv"
sample = pd.read_csv(sample_file, delimiter="\t")
sample
| ProjectID | SampleID | SampleType | TaxonomyID | UniqCodeType | UniqCode | SampleName | ScientificName | CommonName | CollectionYear | ... | FlowRate | MeanPM10 | MeanTotalSuspendedParticles | HumidityStartEnd | WindDirectionStartEnd | WindSpeedMSStartEnd | AmountOfCollectedAirStartEnd | Remarks | RegisterDate | UpdateDate | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | PRA000048 | SAA002867 | ST014 | NaN | NaN | NaN | SW-St.1 | Water | Surface water | 2004 | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 2017/10/25 | 2019/7/18 |
| 1 | PRA000048 | SAA002868 | ST014 | NaN | NaN | NaN | SW-St.3 | Water | Surface water | 2004 | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 2017/10/25 | 2019/7/18 |
| 2 | PRA000048 | SAA002869 | ST014 | NaN | NaN | NaN | SW-St.4 | Water | Surface water | 2004 | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 2017/10/25 | 2019/7/18 |
| 3 | PRA000048 | SAA002870 | ST014 | NaN | NaN | NaN | SW-St.5 | Water | Surface water | 2004 | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 2017/10/25 | 2019/7/18 |
| 4 | PRA000048 | SAA002871 | ST014 | NaN | NaN | NaN | SW-St.7 | Water | Surface water | 2004 | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 2017/10/25 | 2019/7/18 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 271 | PRA000095 | SAA005846 | ST015 | NaN | NaN | NaN | W_1xyz24_20100821 | Water | River water | 2010 | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | Around Kushiro Airport | 2019/7/26 | 2019/7/26 |
| 272 | PRA000095 | SAA005847 | ST015 | NaN | NaN | NaN | W_1xyz25_20100821 | Water | River water | 2010 | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | Around Kushiro Airport | 2019/7/26 | 2019/7/26 |
| 273 | PRA000095 | SAA005848 | ST015 | NaN | NaN | NaN | W_1xyz26_20100821 | Water | River water | 2010 | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | Downstream from the inflow of wastewater from ... | 2019/7/26 | 2019/7/26 |
| 274 | PRA000095 | SAA005849 | ST015 | NaN | ZETTAICODE_FY2011_W | 113680.0 | W_113680_20110702 | Water | River water | 2011 | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | Upstream from the inflow of wastewater from Ch... | 2019/7/26 | 2019/7/26 |
| 275 | PRA000095 | SAA005850 | ST015 | NaN | ZETTAICODE_FY2011_W | 118873.0 | W_118873_20110702 | Water | River water | 2011 | ... | NaN | NaN | NaN | NaN | NaN | NaN | NaN | Downstream from the inflow of wastewater from ... | 2019/7/26 | 2019/7/26 |
276 rows × 66 columns
pfos = data[data["ChemicalName"] == "PFOS"] #ChemicalNameがPFOSのデータだけを抽出
pfoa = data[data["ChemicalName"] == "PFOA"] #ChemicalNameがPFOAのデータだけを抽出
それぞれの中身を確認すると,下記のように見える。
pfos
| MeasuredID | ProjectID | SampleID | ScientificName | ChemicalID | ChemicalName | ExperimentID | MeasuredValue | AlternativeData | Unit | Remarks | RegisterDate | UpdateDate | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 269 | 35646 | PRA000048 | SAA002867 | Water | CH0000365 | PFOS | EXA000001 | 0.0073 | NaN | μg/L | NaN | 2017/10/25 | 2018/6/8 |
| 270 | 35647 | PRA000048 | SAA002868 | Water | CH0000365 | PFOS | EXA000001 | 0.0030 | NaN | μg/L | NaN | 2017/10/25 | 2018/6/8 |
| 271 | 35648 | PRA000048 | SAA002869 | Water | CH0000365 | PFOS | EXA000001 | 0.0034 | NaN | μg/L | NaN | 2017/10/25 | 2018/6/8 |
| 272 | 35649 | PRA000048 | SAA002870 | Water | CH0000365 | PFOS | EXA000001 | 0.0038 | NaN | μg/L | NaN | 2017/10/25 | 2018/6/8 |
| 273 | 35650 | PRA000048 | SAA002871 | Water | CH0000365 | PFOS | EXA000001 | 0.0020 | NaN | μg/L | NaN | 2017/10/25 | 2018/6/8 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 540 | 81380 | PRA000095 | SAA005846 | Water | CH0000365 | PFOS | EXA000001 | 0.0010 | <1.00E-3 | μg/L | NaN | 2019/7/26 | 2019/7/26 |
| 541 | 81381 | PRA000095 | SAA005847 | Water | CH0000365 | PFOS | EXA000001 | 0.0010 | <1.00E-3 | μg/L | NaN | 2019/7/26 | 2019/7/26 |
| 542 | 81382 | PRA000095 | SAA005848 | Water | CH0000365 | PFOS | EXA000001 | 0.0076 | NaN | μg/L | NaN | 2019/7/26 | 2019/7/26 |
| 543 | 81383 | PRA000095 | SAA005849 | Water | CH0000365 | PFOS | EXA000001 | 0.0028 | NaN | μg/L | NaN | 2019/7/26 | 2019/7/26 |
| 544 | 81384 | PRA000095 | SAA005850 | Water | CH0000365 | PFOS | EXA000001 | 0.0120 | NaN | μg/L | NaN | 2019/7/26 | 2019/7/26 |
276 rows × 13 columns
pfoa
| MeasuredID | ProjectID | SampleID | ScientificName | ChemicalID | ChemicalName | ExperimentID | MeasuredValue | AlternativeData | Unit | Remarks | RegisterDate | UpdateDate | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 896 | 46410 | PRA000060 | SAA003568 | Water | CH0000372 | PFOA | EXA000001 | 0.00436 | NaN | μg/L | NaN | 2018/2/9 | 2018/6/8 |
| 897 | 46411 | PRA000060 | SAA003569 | Water | CH0000372 | PFOA | EXA000001 | 0.01166 | NaN | μg/L | NaN | 2018/2/9 | 2018/6/8 |
| 898 | 46412 | PRA000060 | SAA003570 | Water | CH0000372 | PFOA | EXA000001 | 0.01180 | NaN | μg/L | NaN | 2018/2/9 | 2018/6/8 |
| 899 | 46413 | PRA000060 | SAA003571 | Water | CH0000372 | PFOA | EXA000001 | 0.00430 | NaN | μg/L | NaN | 2018/2/9 | 2018/6/8 |
| 900 | 46414 | PRA000060 | SAA003572 | Water | CH0000372 | PFOA | EXA000001 | 0.00439 | NaN | μg/L | NaN | 2018/2/9 | 2018/6/8 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 1143 | 81135 | PRA000095 | SAA005846 | Water | CH0000372 | PFOA | EXA000001 | 0.00100 | <1.00E-3 | μg/L | NaN | 2019/7/26 | 2019/7/26 |
| 1144 | 81136 | PRA000095 | SAA005847 | Water | CH0000372 | PFOA | EXA000001 | 0.00200 | NaN | μg/L | NaN | 2019/7/26 | 2019/7/26 |
| 1145 | 81137 | PRA000095 | SAA005848 | Water | CH0000372 | PFOA | EXA000001 | 0.00140 | tr(1.40E-3) | μg/L | NaN | 2019/7/26 | 2019/7/26 |
| 1146 | 81138 | PRA000095 | SAA005849 | Water | CH0000372 | PFOA | EXA000001 | 0.03900 | NaN | μg/L | NaN | 2019/7/26 | 2019/7/26 |
| 1147 | 81139 | PRA000095 | SAA005850 | Water | CH0000372 | PFOA | EXA000001 | 0.02400 | NaN | μg/L | NaN | 2019/7/26 | 2019/7/26 |
252 rows × 13 columns
PFOSとPFOAのデータだけを抜き出したものから,SampleIDとMeausredValueの列だけを抜き出し,測定値のカラム名がどちらもMeasuredValueになっているので,これをそれぞれPFOS,PFOAに変更する。
pfos = pfos[["SampleID","MeasuredValue"]].rename(columns={'MeasuredValue': 'PFOS'})
pfoa = pfoa[["SampleID","MeasuredValue"]].rename(columns={'MeasuredValue': 'PFOA'})
PFOSとPFOAのデータフレームをSampleIDでマージする。
df = pd.merge(pfos, pfoa, on="SampleID").astype({"PFOS": float}, {"PFOA": float})
df
| SampleID | PFOS | PFOA | |
|---|---|---|---|
| 0 | SAA003568 | 0.00551 | 0.00436 |
| 1 | SAA003569 | 0.01877 | 0.01166 |
| 2 | SAA003570 | 0.01546 | 0.01180 |
| 3 | SAA003571 | 0.00356 | 0.00430 |
| 4 | SAA003572 | 0.00682 | 0.00439 |
| ... | ... | ... | ... |
| 247 | SAA005846 | 0.00100 | 0.00100 |
| 248 | SAA005847 | 0.00100 | 0.00200 |
| 249 | SAA005848 | 0.00760 | 0.00140 |
| 250 | SAA005849 | 0.00280 | 0.03900 |
| 251 | SAA005850 | 0.01200 | 0.02400 |
252 rows × 3 columns
今回知りたいのはPFOSとPFOAの濃度の合計値なので,TOTALという列を作って,そこに合計値を入れる。
df['TOTAL'] = df.sum(axis=1, numeric_only=True)
df
| SampleID | PFOS | PFOA | TOTAL | |
|---|---|---|---|---|
| 0 | SAA003568 | 0.00551 | 0.00436 | 0.00987 |
| 1 | SAA003569 | 0.01877 | 0.01166 | 0.03043 |
| 2 | SAA003570 | 0.01546 | 0.01180 | 0.02726 |
| 3 | SAA003571 | 0.00356 | 0.00430 | 0.00786 |
| 4 | SAA003572 | 0.00682 | 0.00439 | 0.01121 |
| ... | ... | ... | ... | ... |
| 247 | SAA005846 | 0.00100 | 0.00100 | 0.00200 |
| 248 | SAA005847 | 0.00100 | 0.00200 | 0.00300 |
| 249 | SAA005848 | 0.00760 | 0.00140 | 0.00900 |
| 250 | SAA005849 | 0.00280 | 0.03900 | 0.04180 |
| 251 | SAA005850 | 0.01200 | 0.02400 | 0.03600 |
252 rows × 4 columns
試料のテーブルから,SampleIDと緯度経度のデータだけを抜き出す。
sample = sample[["SampleID", "CollectionLongitudeFrom", "CollectionLatitudeFrom"]]
sample
| SampleID | CollectionLongitudeFrom | CollectionLatitudeFrom | |
|---|---|---|---|
| 0 | SAA002867 | 139.850000 | 35.599333 |
| 1 | SAA002868 | 140.000000 | 35.583000 |
| 2 | SAA002869 | 139.834500 | 35.515833 |
| 3 | SAA002870 | 139.900333 | 35.532500 |
| 4 | SAA002871 | 139.833667 | 35.433000 |
| ... | ... | ... | ... |
| 271 | SAA005846 | 144.192783 | 43.062302 |
| 272 | SAA005847 | 144.232365 | 43.041624 |
| 273 | SAA005848 | 144.155650 | 42.997641 |
| 274 | SAA005849 | 141.719167 | 42.765833 |
| 275 | SAA005850 | 141.719167 | 42.782500 |
276 rows × 3 columns
これを,先に作成した濃度のテーブルとSampleIDでマージする。
df = pd.merge(df, sample, on="SampleID")
df
| SampleID | PFOS | PFOA | TOTAL | CollectionLongitudeFrom | CollectionLatitudeFrom | |
|---|---|---|---|---|---|---|
| 0 | SAA003568 | 0.00551 | 0.00436 | 0.00987 | 139.607158 | 35.453746 |
| 1 | SAA003569 | 0.01877 | 0.01166 | 0.03043 | 139.677734 | 35.501549 |
| 2 | SAA003570 | 0.01546 | 0.01180 | 0.02726 | 139.617230 | 35.528481 |
| 3 | SAA003571 | 0.00356 | 0.00430 | 0.00786 | 139.498684 | 35.578287 |
| 4 | SAA003572 | 0.00682 | 0.00439 | 0.01121 | 139.480358 | 35.536396 |
| ... | ... | ... | ... | ... | ... | ... |
| 247 | SAA005846 | 0.00100 | 0.00100 | 0.00200 | 144.192783 | 43.062302 |
| 248 | SAA005847 | 0.00100 | 0.00200 | 0.00300 | 144.232365 | 43.041624 |
| 249 | SAA005848 | 0.00760 | 0.00140 | 0.00900 | 144.155650 | 42.997641 |
| 250 | SAA005849 | 0.00280 | 0.03900 | 0.04180 | 141.719167 | 42.765833 |
| 251 | SAA005850 | 0.01200 | 0.02400 | 0.03600 | 141.719167 | 42.782500 |
252 rows × 6 columns
出来上がったファイルをcsv形式で保存する。
df.to_csv("sum_pfcs.csv")
これを,QGISに読み込ませます。
QGISを使って濃度データを地図上に表示させる
QGISは下記からダウンロードする。
https://www.qgis.org/ja/site/
QGISの使用法はいろんなサイトで説明されているので,そちらを参照してください。
地図情報(GMLシェープファイル)は,国土交通省のGISホームページからダウンロードする。
https://nlftp.mlit.go.jp/index.html
とりあえず今回は,国土数値情報ダウンロードから,2. 政策区域のうち行政区域を利用する。
下記のページから「全国」のデータを選択してダウンロードする。年度は目的に応じて選んでください。
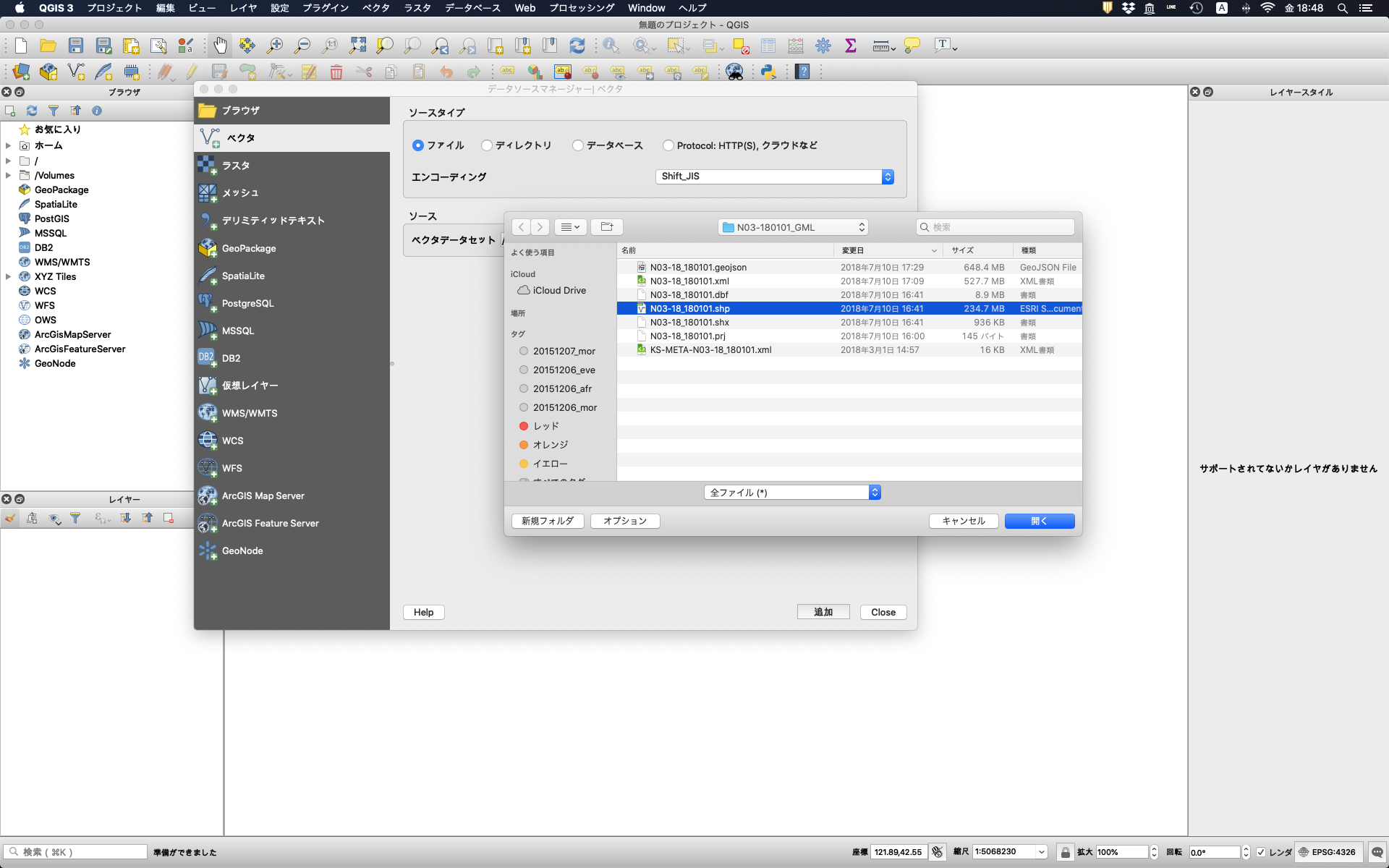
QGISを立ち上げ,データソースマネージャーからベクタのタブをクリックし,上でダウンロードした全国行政区域のシェープファイルを選択して「追加」をクリックする。
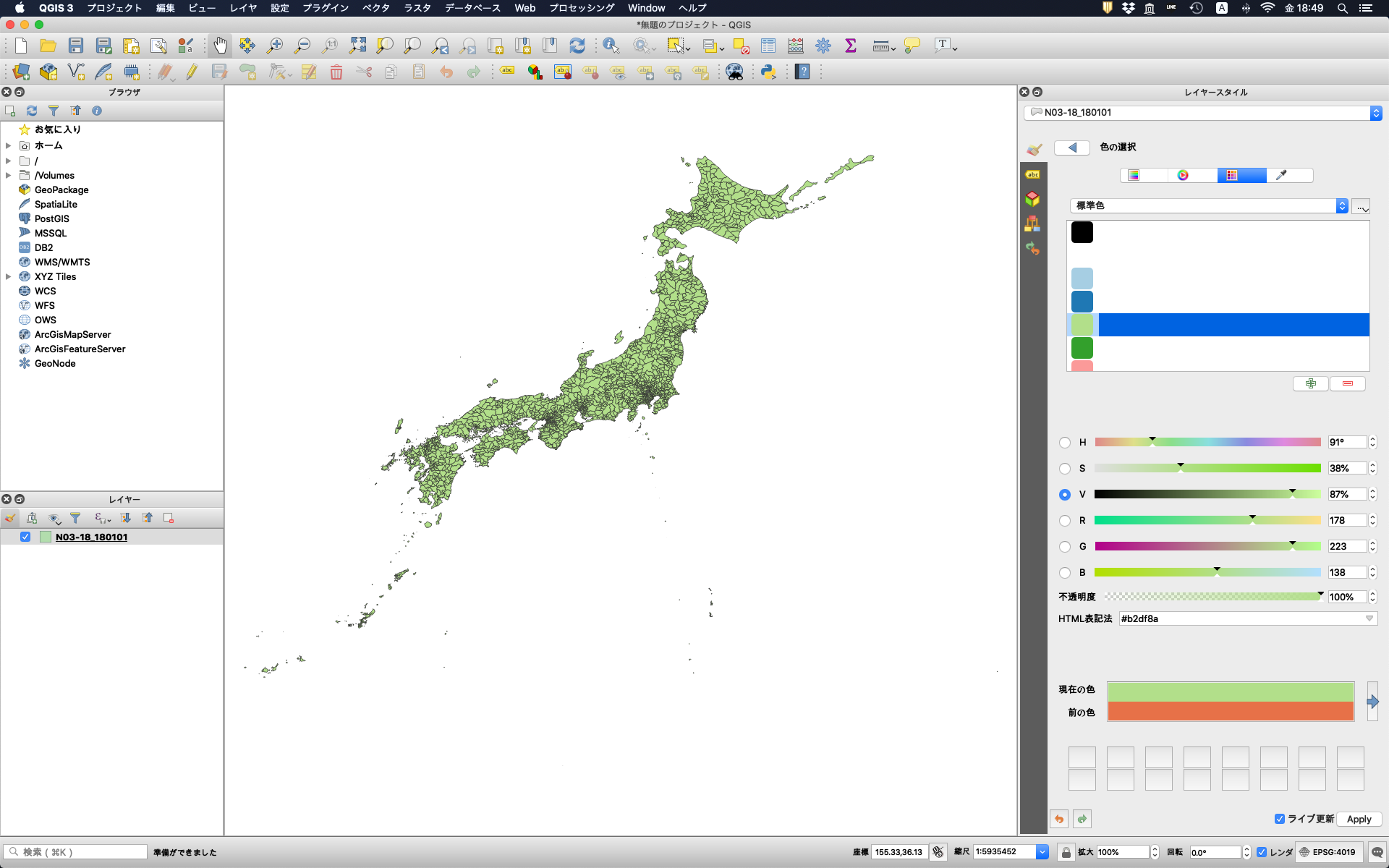
以下のように日本地図が読み込まれるので,好みの色に変更する。
再び,データソースマネージャーからデリミティッドテキストのタブをクリックし,先のセクションで作成したCSVファイル(sum_pfcs.csv)を選択する。その後に,ジオメトリ定義のXフィールドに経度(CollectionLongitudeFrom)を,Yフィールドに緯度(CollectionLatitudeFrom)を選択し,「追加」をクリックする。
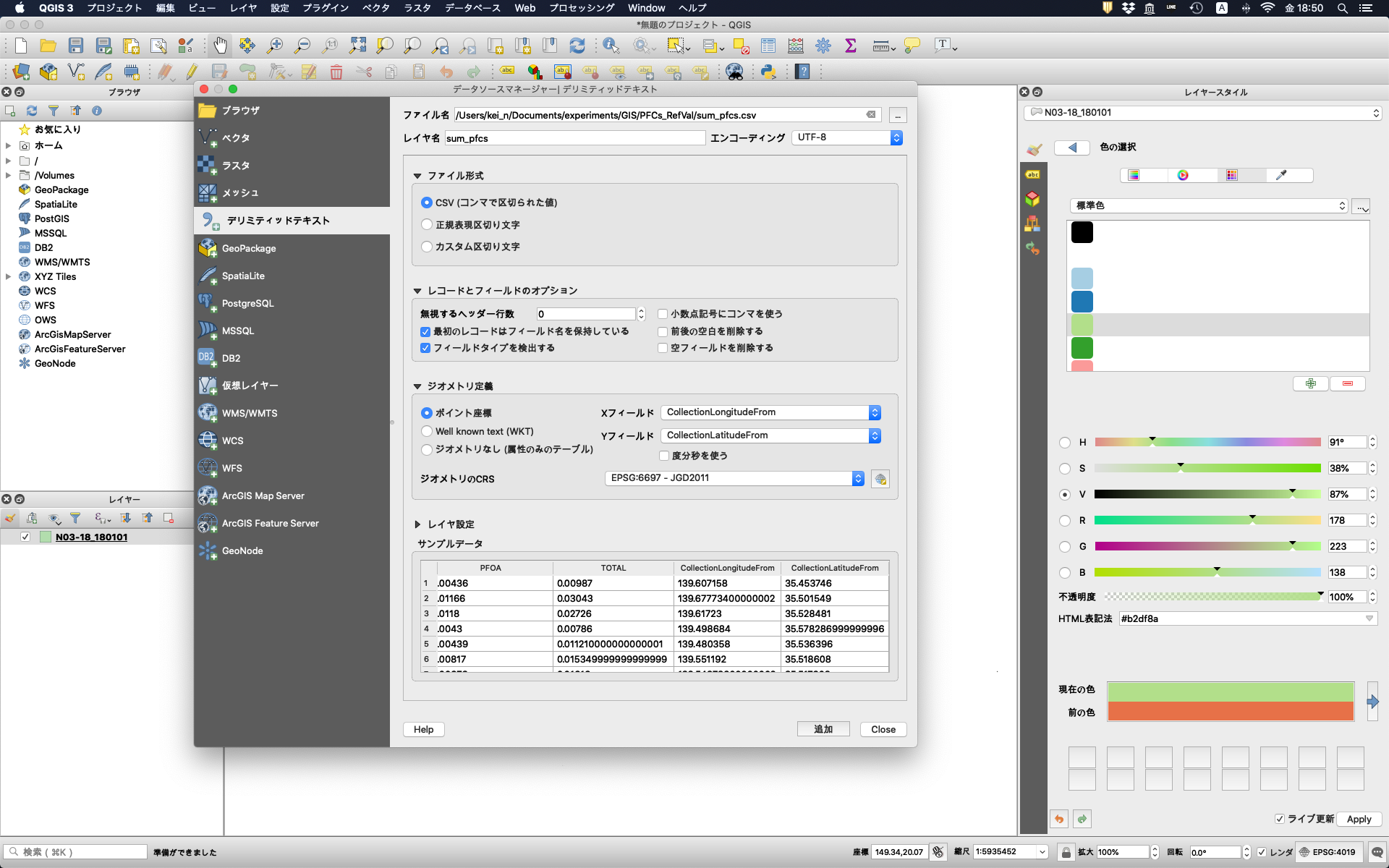
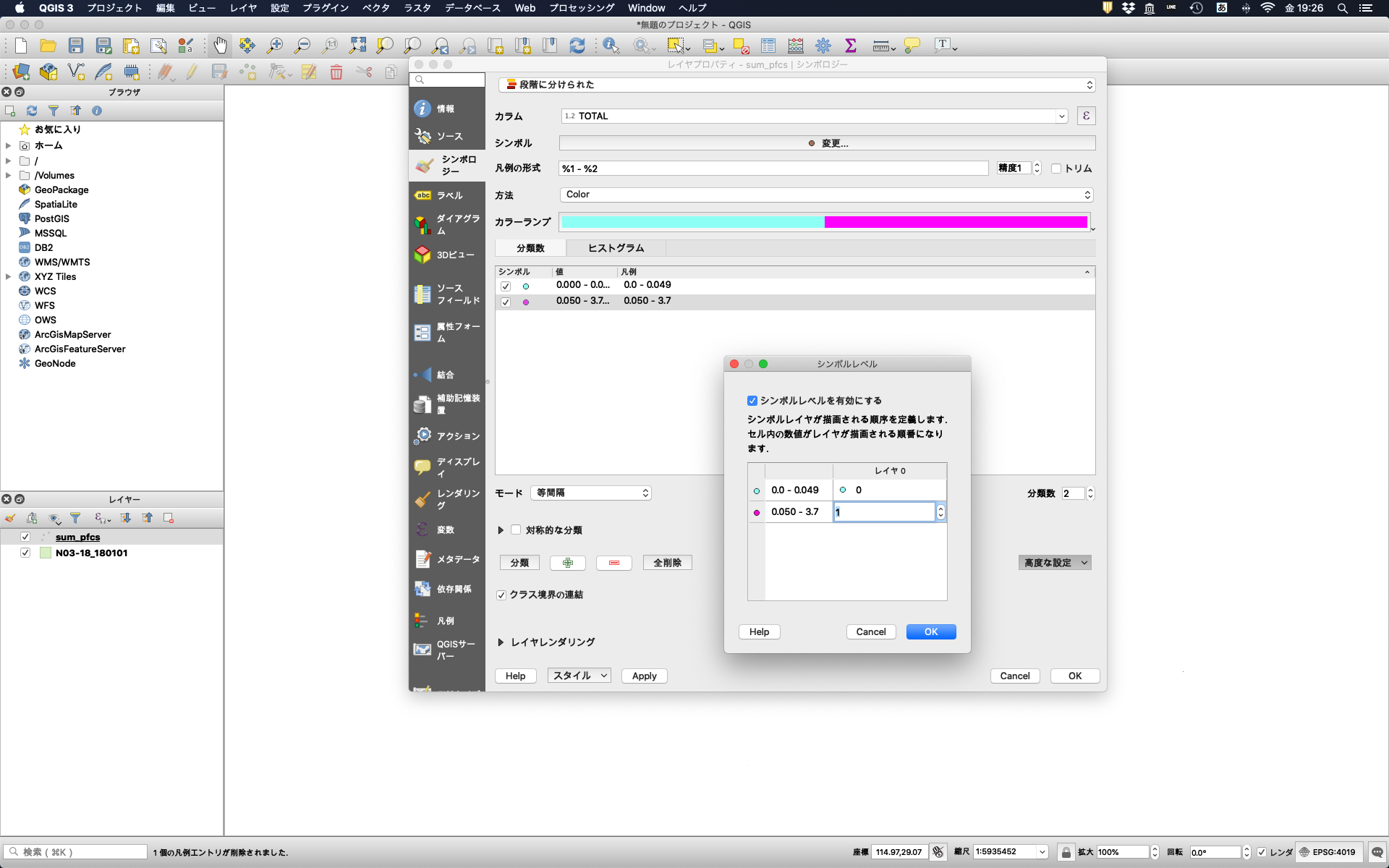
すると,サンプリング地点が地図上にプロットされる。次に,濃度で色分けをする。レイヤーから「sum_pfcs」をダブルクリックするとレイヤプロパティが開くので,シンボロジーのタブを選択する。シンボルは「段階に分けられた」を選び,PFOS+PFOAの濃度で色分けしたいので,カラムには「TOTAL」を選択する。シンボルはお好みで変更する。モードを等間隔のまま,分類数を変更すると,自動で等間隔の分類が出力される。ここでは,分類数を2にして,小さい数値のグループの値を0 - 0.05とする。(ChemTHEATREでは水中の化学物質濃度はµg/Lで統一されているので,50 ng/Lの基準値を超えるか超えないかで色分けしたい場合は,0.05とする必要がある。)デフォルトだと,高濃度のプロットが下層に表示されてしまうので,「高度な設定」から「シンボルレベル」を選択し,シンボルレベルを有効にするのチェックボックスにチェックを入れ,高濃度グループのレイヤを1に設定する。
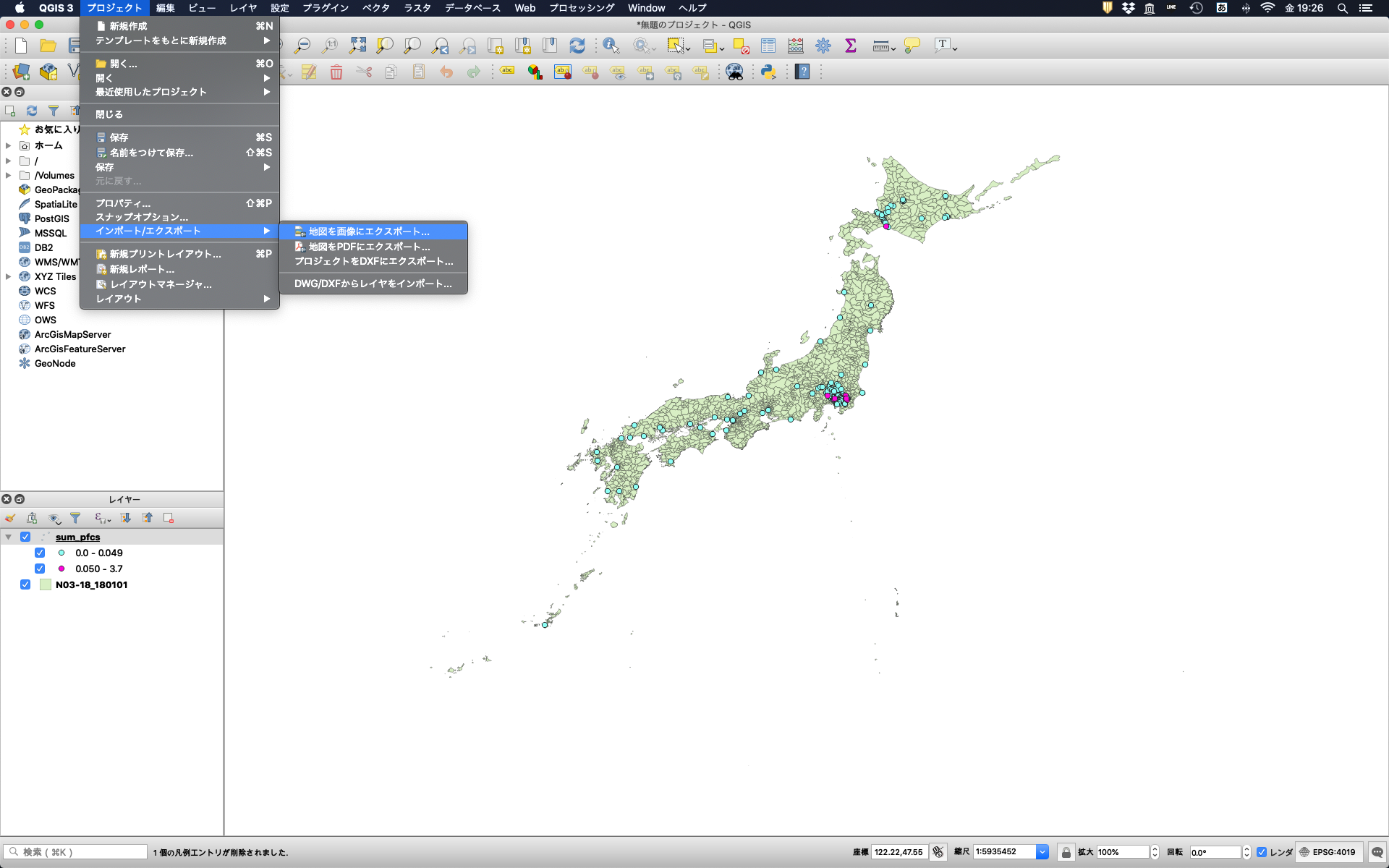
その結果,PFOS+PFOAが50 ng/Lを超過する地点と超過しない地点が色分けされて表示される。この図を出力したい場合は,「プロジェクト」から「インポート/エクスポート」を選び,「地図を画像にエクスポート」あるいは「地図をPDFにエクスポート」を選択し,クリップボードにコピーするか,任意のディレクトリにファイルとして保存する。
いかがでしょう?出来ましたでしょうか?
このようにして,ChemTHEATREに収録されたデータを地図上に表現するなどして活用していただければ幸いです。
「ChemTHEATREでPython学習」に戻る