はじめに
データの前処理とは、データをモデルに学習させる前に行う処理のことです。
料理でいうところの下ごしらえです。
下ごしらえの良し悪しで料理の味(分析結果)も劇的に変わります。
個人的にはデータの前処理はモデルの構築と同じくらい大切な過程だと思っています。
この記事では「前処理の理論」に加えて、「主にpandas, scikit-learnを用いた実装方法」について解説していきたいと思います。
TL;DR
以下概要
1.欠損値の処理
欠損値とは何らかの理由で値が欠損している場合です。
理由は様々ですが、例えばアンケート調査で空欄のままになっている項目があったりするときなどに起こり得ます。
残念ながら、ほとんどの計算ツールは欠損値に対処できないか、欠損値を無視した場合に予期せぬ結果を生み出します。
よって欠損値に適切な対処を施すことが重要になります。
2.特徴量のスケーリング
簡単にいうと特徴量の尺度を揃えること。
忘れがちだが、重要なステップ。
一般的な手法として、正規化と標準化の2つの手法がある。
3.トレーニングデータとテストデータの分割
データセットをトレーニングデータとテストデータに分ける手法について説明します。
4.ラベル、特徴量のエンコーディング
特徴量については、名義特徴量と順序特徴量を区別する必要があります。
これらを数値にマッピング(エンコーディング)する手法について説明していきます。
欠損値の処理
まず欠損値のあるデータを作ってみます。
以下のようなcsvファイルを作成します。
A,B,C,D
1.0,2.0,3.0,4.0
5.0,6.0,,7.0
8.0,9.0,10.0,
そしてこれをpandasで読み込みます。
>>> import pandas as pd
>>> # sample_data.csvを読み込み
>>> df = pd.read_csv(./sample_data.csv)
>>> df
A B C D
0 1.0 2.0 3.0 4.0
1 5.0 6.0 NaN 7.0
2 8.0 9.0 10.0 NaN
このように欠損値の入ったデータができあがりました。
これを処理していきます。
欠損値の特定
isnullメソッドを使って、論理値が含まれたDataFrameオブジェクトを取得する。
>>> df.isnull()
A B C D
0 False False False False
1 False False True False
2 False False False True
さらにsumメソッドを使って欠損値の個数を列ごとに見ることができます。
>>> df.isnull().sum()
A 0
B 0
C 1
D 1
dtype: int64
欠損データの削除
欠損値の処理でもっとも簡単な方法の一つは、欠損値を含む特徴量(列)またはサンプル(行)を完全に削除してしまうことである。
これを行うにはDataFrameオブジェクトのdropnaメソッドを使う。
>>> df.dropna()
A B C D
0 1.0 2.0 3.0 4.0
同様に、axis=1を設定すればNaNを含んでいる特徴量(列)を削除できる。
>>> df.dropna(axis=1)
A B
0 1.0 2.0
1 5.0 6.0
2 8.0 9.0
dropnaメソッドには、他にも役に立つ引数がたくさんある。いくつか紹介する。
# すべてNaNである行を削除
# この場合は全行残る
>>> df.dropna(how='all')
A B C D
0 1.0 2.0 3.0 4.0
1 5.0 6.0 NaN 7.0
2 8.0 9.0 10.0 NaN
# NaNでない値が4つ未満の行を削除
>>> df.dropna(thresh=4)
A B C D
0 1.0 2.0 3.0 4.0
# 特定の列にNaNが含まれている行だけを削除
>>> df.dropna(subset=['C'])
A B C D
0 1.0 2.0 3.0 4.0
2 8.0 9.0 10.0 NaN
補完法
欠損データの削除は有益な情報が失われてしまう恐れがある。そこでよく用いられるのが平均値補完である。平均値補完では欠損値を特徴量の列全体と置き換える。
補完法ではsklearn.preprocessingのImputerクラスを使うのが便利である。
>>> from sklearn.preprocessing import Imputer
>>> # インスタンス作成
>>> imr = Imputer(missing_values='NaN', strategy='mean', axis=0)
>>> # fit(numpyの配列にして渡す)
>>> imr.fit(df.values)
>>> # 補完を実行
>>> imputed_data = imr.transform(df.values)
>>> imputed_data
array([[ 1. , 2. , 3. , 4. ],
[ 5. , 6. , 6.5, 7. ],
[ 8. , 9. , 10. , 5.5]])
axis=1とすると行の平均値がNaNと置き換えられます。また、strategy引数には'median'(中央値)や'most_frequent'(最頻値)(エクセルではMODE関数で求められるやつ)も指定できます。
特徴量のスケーリング
正規化(normalization)
特徴量を[0, 1]の範囲にスケーリングし直すことを意味する。
サンプル$x^{(i)}$の新しい値$x_{norm}^{(i)}$は、次のように計算できます。
$$
x_{norm}^{(i)} = \frac{x^{(i)} - x_{min}}{x_{max} - x_{min}}
$$
min-maxスケーリングはsklearnで実装されていて、次のように使用できる。
>>> from sklearn.preprocessing imoprt MinMaxScaler
>>> # インスタンスを生成
>>> mms = MinMaxScaler()
>>> # データをスケーリング
>>> data_norm = mms.fit_transform(imputed_data)
>>> data_norm
array([[0. , 0. , 0. , 0. ],
[0.57142857, 0.57142857, 0.5 , 1. ],
[1. , 1. , 1. , 0.5 ]])
実際に[0, 1]にスケーリングされた。
標準化(standardization)
標準化は平均値0、標準偏差1になるように変換する。
変換式は以下
$$
x_{std}^{(i)} = \frac{x^{(i)} - \mu_x}{\sigma_x}
$$
ここで$\mu_x$は特徴量の列の平均値、$\sigma_x$は対応する標準偏差を表します。
sklearnには標準化のクラスも実装されており、以下のように標準化できる。
>>> from sklearn.preprocessing import StandardScaler
>>> # インスタンス作成
>>> stdsc = StandardScaler()
>>> data_std = stdsc.fit_transform(imputed_data)
>>> data_std
array([[-1.27872403, -1.27872403, -1.22474487, -1.22474487],
[ 0.11624764, 0.11624764, 0. , 1.22474487],
[ 1.16247639, 1.16247639, 1.22474487, 0. ]])
トレーニングデータとテストデータの分割
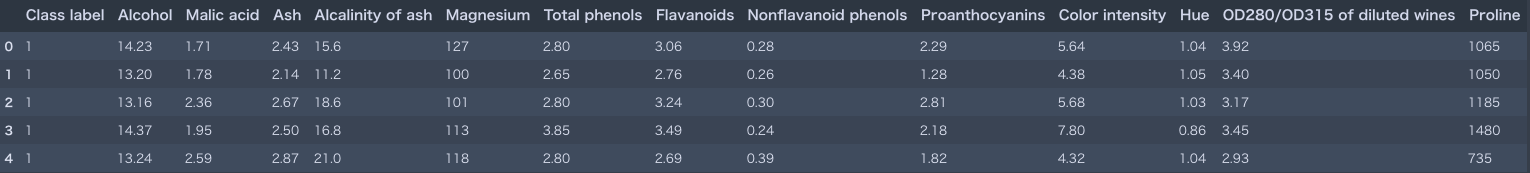
ここではWineというオープンソースのデータセットを使ってみる。これはUCI Machine Learning Repositoryからダウンロードできる。
このデータセットは、178行のワインサンプルとそれらの科学的性質を表す13列の特徴量で構成されています。
>>> url = "https://archive.ics.uci.edu/ml/machine-learning-databases/wine/wine.data"
>>> df_wine = pd.read_csv(url, header=None)
>>> # 列名を設定する
>>> df_wine.columns = ['Class label', 'Alcohol', 'Malic acid', 'Ash',
'Alcalinity of ash', 'Magnesium', 'Total phenols',
'Flavanoids', 'Nonflavanoid phenols', 'Proanthocyanins',
'Color intensity', 'Hue', 'OD280/OD315 of diluted wines',
'Proline']
>>> df_wine.head()
jupyterでみると上の表のように表示されます。ちょっと小さいですがクリックすると拡大できますm(__)m
これらのサンプルはクラス1, 2 ,3のいずれかに属していて、これら3つのクラスはイタリアの同じ地域で栽培されている異なる品種のぶどうを表しています。
このデータセットをテストデータセットとトレーニングデータセットにランダムに分割するにはsklearnのmodel_selectionサブモジュールで定義されているtrain_test_split関数を使います。
>>> from sklearn.model_selection import train_test_split
>>> # 特徴量とクラスラベルに分ける
>>> X, y = df_wine.iloc[:, 1:].values, df_wine.iloc[:, 0].values
>>> # トレーニングデータとテストデータに分割(全体の30%をテストデータにする)
>>> X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=0, stratify=y)
stratifyバラメータに引数としてクラスラベルの配列yを渡すことで、トレーニングセットとテストセットのクラスの比率が元のデータセットと同じになるようにしている。
テストデータの割合ですが、ここでは30%にしていますが、最もよく使われるのは20%, 30%, 40%のいずれかで、データセットが莫大なときはトレーニングデータセットとテストデータセットを90:10または99:1の比率で分割するのも一般的であり、妥当であります。
層化サンプリング(stratified sampling)
層化サンプリングとは簡単に言うと母集団の分布と同じになるようにサンプリングする方法です。
具体的には、どのクラス(層)からも母集団分布に近いサンプリングを行います。
層化サンプリングは上記のtrain_test_split、また同じくscikit-learnのStratifiedShuffleSplitで実装されています。
ラベル、特徴量のエンコーディング
カテゴリデータ(名義特徴量と順序特徴量)と数値特徴量
数値特徴量は数値で表されている特徴量である。(身長、体重など)
カテゴリデータは文字列で表されたデータのこと。
カテゴリデータに関して、名義特徴量と順序特徴量を区別する必要があります。
順序特徴量に関しては、並び替えや順序付けが可能なカテゴリ値とみなすことができます。(例えば、服のサイズS,M,Lなど)
これに対し、名義特徴量には順序がありません。(例えば、服の色・種類など)(補足:色については色味が近い色とかを考えると順序はあるかもしれないがここでは順序のないものとして扱う)
ここでサンプルデータセットを作成します。
>>> import pandas as pd
>>> df = pd.DataFrame([['green', 'M', 10.1, 'class2'],
['red', 'L', 13.5, 'class1'],
['blue', 'XL', 15.3, 'class2']])
>>> df.columns = ['color', 'size', 'price', 'classlabel']
>>> df
color size price classlabel
0 green M 10.1 class2
1 red L 13.5 class1
2 blue XL 15.3 class2
このDataFrameオブジェクトには、名義特徴量(color)、順序特徴量(size)、数値特徴量(price)の列が含まれています。
順序特徴量のエンコーディング
学習アルゴリズムに順序特徴量を正しく解釈させるには、カテゴリ文字列の値を整数に変換する必要がある。
たとえば、以下のようにマッピングする。(この場合便利な関数は用意されていない)
>>> size_mapping = {'XL': 3, 'L': 2, 'M': 1}
>>> # Tシャツのサイズを整数に変換
>>> df['size'] = df['size'].map(size_mapping)
>>> df
color size price classlabel
0 green 1 10.1 class2
1 red 2 13.5 class1
2 blue 3 15.3 class2
クラスラベルのエンコーディング
多くの機械学習ライブラリは、クラスラベルが整数値としてエンコードされていることを要求します。
クラスラベルは順序特徴量ではなく、文字列のラベルにどの整数を割り当てるのかは重要でないことを考えると、クラスラベルを0から順番に番号付けすればよい。
これには順序特徴量のようにディクショナリを定義してやってもよいが、sklearnでLabelEncoderという便利なクラスが定義されている。
>>> from sklearn.preprocessing import LabelEncoder
>>> # インスタンスを生成
>>> class_le = LabelEncoder()
>>> # クラスラベルから整数に変換
>>> y = class_le.fit_transform(df['classlabel'].values)
>>> y
array([1, 0, 1])
元に戻すにはinverse_transformメソッドを利用できる!
名義特徴量でのone-hotエンコーディング
例えば次のようにエンコードすると考える。
- blue → 0
- green → 1
- red → 2
するとredがgreenより大きいなど、順序がついてしまう問題がある。
この問題を回避する一般的な方法はone-hotエンコーディングという手法を使うことである。
考え方は簡単で、名義特徴量の列の一意な値ごとにダミー特徴量を新たに作成する。
この場合は、color特徴量のblue, green, redを3つの新しい特徴量に変換します。
そうすると、たとえば青のサンプルはblue=1, green=0, red=0としてエンコーディングできる。
この変換には、sklearn.preprocessingモジュールで実装されているOneHotEncoderクラスを使用できる。
>>> from sklearn.preprocessing import OneHotEncoder
>>> X = df[['color', 'size', 'price']].values
>>> color_le = LabelEncoder()
>>> X[:, 0] = color_le.fit_transform(X[:, 0])
>>> # one-hotエンコーダの生成
>>> ohe = OneHotEncoder(categorical_features=[0])
>>> # 実行
>>> ohe.fit_transform(X).toarray()
array([[ 0. , 1. , 0. , 1. , 10.1],
[ 0. , 0. , 1. , 2. , 13.5],
[ 1. , 0. , 0. , 3. , 15.3]])
OneHotEncoderで初期化するときにcategorical_features引数を使って、変換したい変数の列位置をリストで定義しています。
one-hotエンコーディングを使ってダミー特徴量を作成する場合は、pandasのget_dummies関数を使用するとさらに便利であります。
get_dummies関数をDataFrameオブジェクトに適用すると、文字列値を持つ列だけが変換されます。
>>> pd.get_dummies(df[['price', 'color', 'size']])
price size color_blue color_green color_red
0 10.1 1 0 1 0
1 13.5 2 0 0 1
2 15.3 3 1 0 0
おわりに
以上でデータの前処理に関する解説は終わりです。
いろんな知見が欲しいので、何か補足等ありましたらコメントで教えてくださると嬉しいです!