Chainer Advent Calendar 2017 4日目の記事です。
はじめに
最近、猫も杓子も機械学習って感じですね。「私も機械学習やってみたい!でもどうやっていいかわからない」って人、多いと思います。で、TensorFlowでもChainerでもセットアップして、MNISTとかの学習やってみて「あーなんか動いてるな」となった後、どうして良いかわからなくなる人、多いんじゃないでしょうか。画像の分類とかやってみたくても、スクレイピングとかやらなきゃいけないし、タグ付けとかどうすればいいのかわからないし、そもそもたくさんの画像をどうやってニューラルネットに食わせればいいのかわからない・・・。機械学習ビギナー、あるあるですね。
というわけで、本稿では機械学習ビギナー(=僕)が、とりあえず簡単に何か機械学習を試すために必要な最低限のプログラム群を作ってみたりして、初心者の敷居を下げられたらなぁ、とか思って本稿を書いたのですが、前日のChainer v3 ビギナー向けチュートリアルが素晴らしい記事だったので、あまり本稿の意味は無い気もしてきました。まぁせっかく書いたので公開します。
とりあえず目標は「とにかく数字の羅列が書かれたデータファイルさえ用意できれば機械学習させることができて、その学習結果を何かに使うことができるようになる」ことです。
そのために、
- 二値分類問題にする
- データはファイルから与えるようにする
- データ形式は単純な形にする(一行一レコードの数字の羅列)
- 学習したモデルを、別の用途に使うサンプルとして、C++から読み込んで動作確認をする
といったところまでやります。
ソースは以下に置いてあります。
Chainerのセットアップ
まずはChainerのセットアップをします。Mac+Homebrewだとかなり簡単で、
- pyenvをインストール
- pyenvからanacondaをインストール
- パスの修正
- pipからChainerをインストール
これだけでいけます。
$ brew update
$ brew install pyenv
$ pyenv install anaconda2-4.4.0
$ pyenv install anaconda3-4.4.0
$ pyenv global anaconda2-4.4.0
$ export PATH=$HOME/.pyenv/shims:$PATH
$ pip install chainer
別途pipをインストールしてある人は、pyenvが入れたpipが先に見つかるようにパスを修正しておきましょう。今後も使う場合は.zshrcに入れておくことを忘れずに。もしかしたら途中でXcodeをインストールしろとか言われるかもしれませんが、その場合は入れてください。
Chainerが入ったかどうか確認しましょう。
$ ipython
In [1]: import chainer
In [2]: print chainer.__version__
3.1.0
無事に3.1.0が入ったようですね。
モデルの定義
まず、Chainerのモデルを定義します。Chainerはその名の通り、ユニットのリンクをどうつなげるかを定義することでニューラルネットを構築します。とりあえず、三層のニューラルネットによる二値分類をやることにしましょう。どんな入力をするかは後で決めますが、二値分類なので、出力は二つです。また、中間層の数もいろいろ変えられますが、ここでは入力の数と同じとしましょう。
つまり、入力をn_unitsとすると、n_units→n_units→n_units→2、というような三層のニューラルネットを組むことにします。以下のように、モデルを定義するスクリプトを書いてみましょう。
import chainer
import chainer.functions as F
import chainer.links as L
from chainer import training
from chainer.training import extensions
class MLP(chainer.Chain):
def __init__(self, n_units, n_out):
super(MLP, self).__init__(
l1 = L.Linear(None, n_units),
l2 = L.Linear(None, n_units),
l3 = L.Linear(None, n_out)
)
def __call__(self, x):
h1 = F.relu(self.l1(x))
h2 = F.relu(self.l2(h1))
return self.l3(h2)
class Model:
def __init__(self,n_in):
self.model = L.Classifier(MLP(n_in, 2))
def load(self,filename):
chainer.serializers.load_npz(filename, self.model)
def save(self,filename):
chainer.serializers.save_npz(filename, self.model)
def predictor(self, x):
return self.model.predictor(x)
def get_model(self):
return self.model
ほとんどChainerのMNISTサンプルと同じですが、後でデータを保存/読込をするために、シリアライズ/デシリアライズのラッパーとしてsave、loadメソッドを作っています。
データの読み込み
何かファイルでデータが与えられた時、それをChainerが扱える形式に変換する必要があります。Chainerで学習に使うデータはchainer.datasets.TupleDatasetにする必要があります。TupleDatasetの第一引数にはデータのリスト、第二引数にはラベルのリストを与えます。それぞれnumpyのfloat32、int32型にする必要があります。
CSV形式で与えられたファイルを読み込んでTupleDatasetにするスクリプトはこんな感じになるでしょう。
import chainer
import numpy as np
class Data:
@staticmethod
def load_data(f_on, f_off):
data = []
for line in open(f_on,'r'):
s = line.split(",")
n = [float(t) for t in s]
data.append([n,1])
for line in open(f_off,'r'):
s = line.split(",")
n = [float(t) for t in s]
data.append([n,0])
return data
@staticmethod
def make_dataset(data):
n = len(data)
xn = len(data[0][0])
x = np.empty((n,xn),dtype=np.float32)
y = np.empty(n,dtype=np.int32)
for i in range(n):
x[i] = np.asarray(data[i][0])
y[i] = data[i][1]
return chainer.datasets.TupleDataset(x,y)
まず、二値分類を扱うので、「ON」のデータファイル群と「OFF」のデータファイル群が分かれているとします1。それぞれを読み込むのがload_dataです。これは、「データの配列, ラベル」という二次元配列を作ります。
その二次元配列からTupleDatasetを作るのがmake_datasetです。もっと賢い方法がありそうな気がしますが、Python慣れしていないので勘弁してください。使い方としては
data = Data.load_data("on.txt","off_txt")
dataset = Data.make_dataset(data)
などとすれば、Chainerに食わせることができるdatasetができます。データの読み込みとデータセットの作成を分けているのは後でdataだけを使いたいことがあるためです。
学習
学習はサンプルそのままなので簡単でしょう。
import chainer
import numpy as np
from chainer import training
from chainer.training import extensions
import random
import collections
from data import Data
from model import Model
def main(f_on, f_off):
data = Data.load_data(f_on,f_off)
random.seed(1)
np.random.seed(1)
random.shuffle(data)
dataset = Data.make_dataset(data)
epoch = 200
batchsize = 100
units = len(data[0][0])
m = Model(units)
model = m.get_model()
gpu = -1
# for GPU
if gpu >= 0:
chainer.cuda.get_device(0).use()
model.to_gpu()
optimizer = chainer.optimizers.Adam()
#optimizer = chainer.optimizers.SGD()
optimizer.setup(model)
test_ratio = 0.05
nt = int(len(data)*test_ratio)
test = dataset[:nt]
train = dataset[nt:]
train_iter = chainer.iterators.SerialIterator(train, batchsize)
test_iter = chainer.iterators.SerialIterator(test, batchsize, repeat=False, shuffle=False)
updater = training.StandardUpdater(train_iter, optimizer, device=gpu)
trainer = training.Trainer(updater, (epoch, 'epoch'), out='result')
trainer.extend(extensions.Evaluator(test_iter, model,device=gpu))
trainer.extend(extensions.dump_graph('main/loss'))
trainer.extend(extensions.snapshot(), trigger=(epoch, 'epoch'))
trainer.extend(extensions.LogReport())
trainer.extend(extensions.PrintReport(
['epoch', 'main/loss', 'validation/main/loss',
'main/accuracy', 'validation/main/accuracy']))
trainer.extend(extensions.ProgressBar())
# Training
trainer.run()
if gpu >= 0:
model.to_cpu()
m.save('test.model')
if __name__ == '__main__':
main("on.txt","off.txt")
難しいところはないと思います。サンプルと違うところは
- 最初に一括してデータを読み込んでいる
- ラベル1のデータファイルが
on.txt、ラベル0のデータがoff.txtで与えられる - データのうち、5%用をテスト用(
train = dataset[nt:])、95%を学習用(train = dataset[nt:])に振り分けている - 最後に学習済みデータをシリアライズして保存している(
m.save('test.model'))
といったところです。モデル定義を外に出したのですっきりしたのと、シリアライズ・デシリアライズメソッドをつけているのが地味に便利です。
また、データセットは最初の半分が1、残りが0のラベルがついているので、念のため最初にシャッフルしています。エポックやバッチサイズは適当です。最適化手法はAdamにしてありますが、たまにこけるので、その時にはSGDに変えたりします。たくさんあるので好きなのを選んでください。
データ作成
データは、ラベル1のデータ(ON)と、ラベル0のデータ(OFF)をそれぞれ別ファイルで与えることにします。また、後でちゃんと学習できたかテストするためのデータも作成しましょう。どんなデータでもいいですが、とりあえず二次曲線を与えて、下に凸ならON(ラベル1)、上に凸ならOFF(ラベル0)というデータを作ってみましょう。Pythonでやるべきなのでしょうが、筆者の母国語がRubyなのでRubyで失礼します。
def makedata(s,n,l,filename)
puts filename
f = open(filename,"w")
n.times do
a = s*rand()/l
b = rand()*l
c = rand()
data = []
l.times do |i|
data.push a*(i-b)**2 + c
end
ave = data.inject{|sum,v| sum+v}/data.size
data.map{|v| v - ave}
f.puts data.join(",")
end
end
TRAIN_DATA = 1000
TEST_DATA = 100
L = 10
makedata(1,TRAIN_DATA,L,"on.txt")
makedata(-1,TRAIN_DATA,L,"off.txt")
makedata(1,TEST_DATA,L,"on_test.txt")
makedata(-1,TEST_DATA,L,"off_test.txt")
実行すると4つのファイルを作ります。
$ ruby makedata.rb
on.txt
off.txt
on_test.txt
off_test.txt
それぞれ、
-
on.txtラベル1の学習用データ -
off.txtラベル0の学習用データ -
on_test.txtラベル1のテスト用データ -
off_test.txtラベル1のテスト用データ
ここで、「テスト用データ」というのは、学習が終わったモデルに食わせて結果を確認するためのもので、学習中に使うテストデータではありません。学習中に使うテストデータは、学習用データの5%を使うことにします。
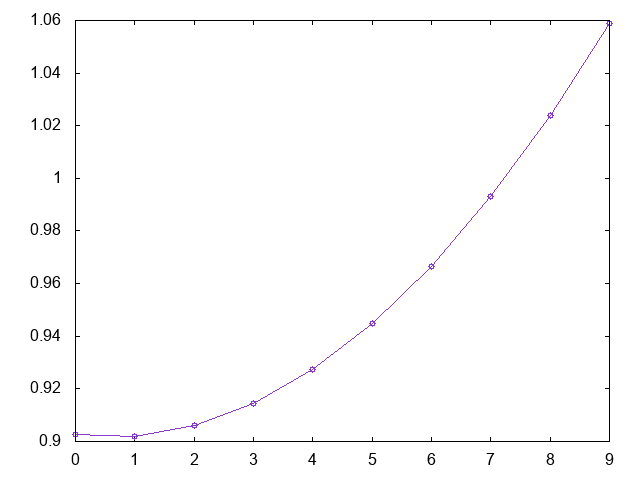
たとえばon.txtの一行目はこんな感じになります。
0.9025886354206973,0.9020880910609956,0.9060572974424307,0.9144962545650025,0.9274049624287111,0.9447834210335564,0.9666316303795386,0.9929495904666575,1.0237373012949131,1.0589947628643057
これをプロットするとこんな感じになります。
下に凸なので、これはラベル1に分類すべきデータです。
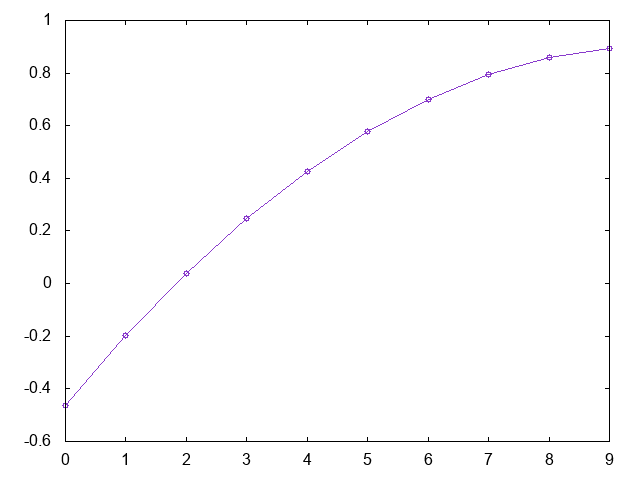
同様にoff.txtの一行目は
-0.46338239086091526,-0.1975468181134049,0.03954800953069304,0.24790209207137892,0.4275154295086525,0.5783880218425139,0.7005198690729633,0.7939109712000002,0.8585613282236251,0.8944709401438378
プロットするとこんな感じです。
これは上に凸なのでラベル0に分類されるべきデータです。
学習
さて、ファイルができた段階で学習させてみましょう。train.pyを実行するだけです。すぐに終わります。
$ python train.py
epoch main/loss validation/main/loss main/accuracy validation/main/accuracy
1 0.514519 0.51211 0.815263 0.83
2 0.431453 0.451503 0.87 0.86
(snip)
199 0.00472971 0.00693513 0.998947 1
200 0.004677 0.00922504 0.999474 1
楽勝で学習できているみたいですね。
学習モデルのテスト
普通、何かを学習させるのは、その後分類器として使いたいからですよね。というわけで、学習済みのモデルを読み込み、学習に使ったのとは異なるデータセットを与えて、ちゃんと分類できているかを確認してみましょう。そのために先程on_test.txtとoff_test.txtを作ったので、それらを読み込ませて、成績を調べます。
学習後にはtest.modelというファイルができてるはずです。これはこれはChainerがシリアライズしたモデルで、Chainerから読み込む分には中身を気にする必要はありません。単にデシリアライズするだけでモデル読み込み完了です。データのデシリアライズは、先程作ったModelクラスのloadメソッドとしてラップしてあります。
from __future__ import print_function
import sys
import numpy as np
from model import Model
from data import Data
def main(f_model, f_on,f_off):
data = Data.load_data(f_on,f_off)
n = len(data)
units = len(data[0][0])
model = Model(units)
model.load(f_model)
s = 0
f = open('failed.txt','w')
for i in range(n):
x = np.array([data[i][0]],dtype=np.float32)
y = model.predictor(x).data[0]
if np.argmax(y) == data[i][1]:
s = s + 1
else:
f.write(str(data[i][0])+'\n')
c = float(s)/float(n)
print('Success/Total = %d/%d' % (s,n))
print('Ratio = %f' % c)
if __name__ == '__main__':
main("test.model","on_test.txt","off_test.txt")
データセットを読み込み、分類させてみて、その結果がデータのラベルと一致していたら成功とします。また、失敗した場合はfailed.txtにそのデータを保存するようにしています。実行するとこうなります。
$ python test.py
Success/Total = 200/200
Ratio = 1.000000
今回は100%分類に成功しましたが、たまに曲率が極端に小さいデータを与えると失敗したりします。
データのエクスポート
学習はPythonで行いましたが、そのモデルをC++のプログラムに組み込みたい、ということもあるでしょう。その場合はChainerがシリアライズしたデータをC++から読み込ませる必要があります。Chainerがシリアライズしたデータの中身は、numpyのデータをzipしたものになっています。
$ zipinfo test.model
Archive: test.model
Zip file size: 2116 bytes, number of entries: 6
-rw------- 2.0 unx 480 b- defN 17-Nov-28 13:34 predictor/l2/W.npy
-rw------- 2.0 unx 120 b- defN 17-Nov-28 13:34 predictor/l1/b.npy
-rw------- 2.0 unx 88 b- defN 17-Nov-28 13:34 predictor/l3/b.npy
-rw------- 2.0 unx 120 b- defN 17-Nov-28 13:34 predictor/l2/b.npy
-rw------- 2.0 unx 480 b- defN 17-Nov-28 13:34 predictor/l1/W.npy
-rw------- 2.0 unx 160 b- defN 17-Nov-28 13:34 predictor/l3/W.npy
6 files, 1448 bytes uncompressed, 1422 bytes compressed: 1.8%
行列Wとベクトルbが3つずつ保存されていることがわかります。zipファイルをC++から読み込むのは面倒なので、C++に読み込みやすい形にPython側でエクスポートしてしまいましょう2。
from __future__ import print_function
import struct
import numpy as np
from model import Model
from data import Data
def main(f_model, f_on, f_off):
d = Data()
data = d.load_data(f_on, f_off)
n = len(data)
units = len(data[0][0])
model = Model(units)
model.load(f_model)
p = model.model.predictor
d = bytearray()
for v in p.l1.W.data.reshape(units*units):
d += struct.pack('f',v)
for v in p.l1.b.data:
d += struct.pack('f',v)
for v in p.l2.W.data.reshape(units*units):
d += struct.pack('f',v)
for v in p.l2.b.data:
d += struct.pack('f',v)
for v in p.l3.W.data.reshape(2*units):
d += struct.pack('f',v)
for v in p.l3.b.data:
d += struct.pack('f',v)
open("test.dat",'w').write(d);
print("Exported to test.dat")
a = [0.5]*units
x = np.array([a],dtype=np.float32)
y = model.predictor(x).data[0]
print(y)
if __name__ == '__main__':
main("test.model","on_test.txt","off_test.txt")
モデルをロードして、L1,L2,L3の順番でWとbをそのまま生のfloatデータとしてpackしてからファイルに保存しているだけです。ただし、最後にC++の読み込みテスト用に「全て0.5の値を持つベクトルを食わせた時のモデルの出力」を表示しています。実行すると以下のような出力となるでしょう。
$ python export.py
Exported to test.dat
[ 0.69980705 1.07327592]
データがtest.datに保存され、かつ0.5,0.5,...0.5を食わせた時の出力が[ 0.69980705 1.07327592]であることがわかります。この「生の重み」はデバッグに有効です。
C++からの読み込み
さて、保存したtest.datをC++から読み込みましょう。適当なヘッダファイルを書いてやります。
# pragma once
//------------------------------------------------------------------------
# include <iostream>
# include <fstream>
# include <vector>
# include <math.h>
# include <algorithm>
//------------------------------------------------------------------------
typedef std::vector<float> vf;
//------------------------------------------------------------------------
class Link {
private:
vf W;
vf b;
float relu(float x) {
return (x > 0) ? x : 0;
}
public:
const int n_in, n_out;
Link(int in, int out) : n_in(in), n_out(out) {
W.resize(n_in * n_out);
b.resize(n_out);
}
void read(std::ifstream &ifs) {
ifs.read((char*)W.data(), sizeof(float)*n_in * n_out);
ifs.read((char*)b.data(), sizeof(float)*n_out);
}
vf get(vf x) {
vf y(n_out);
for (int i = 0; i < n_out; i++) {
y[i] = 0.0;
for (int j = 0; j < n_in; j++) {
y[i] += W[i * n_in + j] * x[j];
}
y[i] += b[i];
}
return y;
}
vf get_relu(vf x) {
vf y = get(x);
for (int i = 0; i < n_out; i++) {
y[i] = relu(y[i]);
}
return y;
}
};
//------------------------------------------------------------------------
class Model {
private:
Link l1, l2, l3;
public:
const int n_in, n_out;
Model(int in, int n_units, int out):
n_in(in), n_out(out),
l1(in, n_units), l2(n_units, n_units), l3(n_units, out) {
}
void load(const char* filename) {
std::ifstream ifs(filename);
if (ifs.fail()) {
std::cerr << "Could not read file " << filename << std::endl;
}
l1.read(ifs);
l2.read(ifs);
l3.read(ifs);
}
vf predict(vf &x) {
return l3.get(l2.get_relu(l1.get_relu(x)));
}
int argmax(vf &x) {
vf y = predict(x);
auto it = std::max_element(y.begin(), y.end());
auto index = std::distance(y.begin(), it);
return index;
}
};
//------------------------------------------------------------------------
ChainerのLinkにあたるクラスと、それをまとめたModelクラスを作っています。いちいちstd::vector<float>と書くのが面倒だったのでvfにtypedefしてあります。モデルのユニット数をちゃんとファイルから判断するようにすれば麗しいのでしょうが、ここでは手抜きでハードコーディングすることにします。
さて、モデルデータを読み込んで、0.5,0.5,...0.5を食わせてみましょう。
# include <cstdio>
# include "model.hpp"
int
main(void) {
const int n_in = 10;
const int n_units = 10;
const int n_out = 2;
Model model(n_in, n_units, n_out);
model.load("test.dat");
vf x(model.n_in, 0.5);
vf y = model.predict(x);
printf("[%.8f,%.8f]\n", y[0], y[1]);
}
実行するとこんな感じになります。
$ g++ test.cpp
$ ./a.out
[0.69980741,1.07327533]
さきほどのexport.pyとほぼ同じ結果となり、モデルの読み込みがうまくいっていることがわかります3。
さらに、テスト用のデータを読み込んで分類するタスクもやらせてみましょう。それらも含めたソースはこんな感じになるでしょう。
//------------------------------------------------------------------------
# include <cstdio>
# include <fstream>
# include <string>
# include <sstream>
# include "model.hpp"
//------------------------------------------------------------------------
void
import_test(Model &model) {
vf x(model.n_in, 0.5);
vf y = model.predict(x);
printf("[%.8f,%.8f]\n", y[0], y[1]);
}
//------------------------------------------------------------------------
std::vector<vf>
read_file(const std::string filename) {
std::ifstream ifs(filename);
std::string str;
int sum = 0;
std::vector<vf> vvf;
while (getline(ifs, str)) {
std::string token;
std::istringstream ss(str);
vf x;
while (getline(ss, token, ',')) {
float v = stof(token);
x.push_back(v);
}
vvf.push_back(x);
}
return vvf;
}
//------------------------------------------------------------------------
void
file_test(Model &model) {
std::vector<vf> vvf = read_file("on_test.txt");
int sum = vvf.size();
int s = 0;
for (auto &x : vvf) {
if (model.argmax(x) == 1)s++;
}
vvf = read_file("off_test.txt");
sum += vvf.size();
for (auto &x : vvf) {
if (model.argmax(x) == 0)s++;
}
printf("Success/Total = %d/%d\n", s, sum);
printf("Ratio = %f\n", static_cast<double>(s) / static_cast<double>(sum));
}
//------------------------------------------------------------------------
int
main(void) {
const int n_in = 10;
const int n_units = 10;
const int n_out = 2;
Model model(n_in, n_units, n_out);
model.load("test.dat");
import_test(model);
file_test(model);
}
//------------------------------------------------------------------------
以下は実行結果です。
$ g++ -O3 model.cpp
$ time ./a.out
[0.69980741,1.07327533]
Success/Total = 200/200
Ratio = 1.000000
./a.out 0.00s user 0.00s system 53% cpu 0.011 total
ちゃんと分類に成功しているようですね。ちなみにPythonによる分類にかかる時間はこんな感じです。
$ time python test.py
Success/Total = 200/200
Ratio = 1.000000
python test.py 0.51s user 0.23s system 68% cpu 1.099 total
こういうのの比較に意味があるのかわかりませんし、そもそもPython版もC++版は全く高速化とか考えていないコードなのでどちらも高速化の余地はあるでしょうが、普通に組んだ範囲ではC++の方が早いです4。
別のデータを食わせてみる
上記までで、データファイルさえ作ってしまえば後は学習からC++の分類器までブラックボックスで作ることができるようになりました。というわけで別のデータセットを食わせてみましょう。以下は10個の0か1の数字列のうち、1の数が奇数個のものをON(ラベル1)、偶数個のものをOFF(ラベル0)とするデータセットを作るスクリプトです。
def makedata(s,n,l,filename)
puts filename
f = open(filename,"w")
num = 0
while num < n
str = Array.new(10){ rand<0.5? 1:0}.join(",")
if (str.count("1") % 2 == s)
f.puts str
num = num + 1
end
end
end
TRAIN_DATA = 1000
TEST_DATA = 100
L = 10
makedata(1,TRAIN_DATA,L,"on.txt")
makedata(0,TRAIN_DATA,L,"off.txt")
makedata(1,TEST_DATA,L,"on_test.txt")
makedata(0,TEST_DATA,L,"off_test.txt")
あとは先程の一連の手順をそのまま実行できます。
$ ruby evenodd.rb # データファイルの作成
on.txt
off.txt
on_test.txt
off_test.txt
$ python train.py # 学習
epoch main/loss validation/main/loss main/accuracy validation/main/accuracy
1 0.706044 0.692044 0.491579 0.55
2 0.700221 0.691917 0.504211 0.56
(snip)
198 0.245688 0.291011 0.901579 0.85
199 0.244825 0.292672 0.901579 0.85
200 0.240869 0.301571 0.900526 0.85
$ python test.py # 学習モデルのテスト
Success/Total = 173/200
Ratio = 0.865000
$ python export.py # モデルのエクスポート
Exported to test.dat
[ 1.68379354 1.78699565]
$ ./a.out # ユニット数が同じなら再コンパイル不要
[1.68379283,1.78699589]
Success/Total = 173/200
Ratio = 0.865000
「データが上に凸」よりも、より細かい情報が必要になったため学習に苦戦したようですが、それでも87%程度は正答しているようです。
まとめ
Chainerを使って、二値分類をやってみました。Chainerのサンプルというとすごくかっこいい何かが多数見つかりますが、機械学習初心者は「そもそもデータをどうやってChainerに食わせれば良いのかも、学習したモデルをどうやって使えばいいのかもわからない」状態で、最初のハードルを超えるのが困難です(僕はそうでした)。この記事のソースを使えば、「とにかくデータをCSV形式で吐ければChainerで二値分類の学習ができる」ところまでは行きます。その後、二値分類を多値分類にしたり、食わせるデータをテキストから画像や音声にしたりとステップアップするのは難しくないでしょう。
本稿がChainerを使った機械学習の最初のステップの助けになれば幸いです。
参考
- MacでのChainerインストールに必要なものの説明とまとめ
- 人工知能(Chainer/TensorFlow/CSLAIER)のためのpython環境構築
- Chainerで学習したモデルをC++で読み込む