モチベーション
研究者のみなさま、今年も科研費執筆お疲れ様です。科研費申請書の「関連する国内外の研究動向と本研究の位置づけ」欄を書く(助けにする)ために、PubMedの論文情報を自然言語処理で簡単に解析したいです。語の頻度の可視化と、キーワード抽出を試してみます。
環境
- MacOS Mojave 10.14.5
- Python 3.7.10
- Jupyter Notebook 6.4.3
準備
pip install Bio
pip install nltk
pip install rake-nltk
pip install yake
pip install spacy
python3 -m spacy download en
BiopythonでPubMedから論文情報をダウンロードする
まず検索して件数を出します。Chlamydomonasで検索してみましょう。ここの9.14を参考にやっていきます。
from Bio import Entrez
Entrez.email = "sample@email" #メールアドレスを入力
handle = Entrez.egquery(term="Chlamydomonas")
record = Entrez.read(handle)
for row in record["eGQueryResult"]:
if row["DbName"]=="pubmed":
number = row["Count"]
print(number) #9335
次にesearchを使ってIDリストを取得します。
handle = Entrez.esearch(db="pubmed", term="Chlamydomonas", retmax=number, usehistory="y") #取得上限をさっき検索した数に設定
record = Entrez.read(handle)
idlist = record["IdList"]
print(idlist)
efetchで論文情報を取得します。
from Bio import Medline
handle = Entrez.efetch(db="pubmed", id=idlist, rettype="medline",
retmode="text")
records = Medline.parse(handle)
records = list(records)

論文情報が取得できました。タイトル(TI)とアブストラクト(AB)を抽出して操作します。
titles = []
abstracts = []
for record in records:
title = record.get("TI", "?")
abstract = record.get("AB", "?")
titles.append(title)
abstracts.append(abstract)
texts = titles + abstracts #リストを結合
キーワードの抽出と可視化
日本語で前にやったのとほぼ同じです。前処理として形態素解析をしないだけ日本語より楽です。
単純に語の出現頻度を数える
textsをバラバラにしてみます。まずはテストから。
import re
print(re.split('\.|,| |:|;|\(|\)|\[|\]|\?|\!', abstracts[0]))
いい感じですが、空の要素が含まれるので削除します。
words = re.split('\.|,| |:|;|\(|\)|\[|\]|\?|\!', abstracts[0])
words = [word for word in words if word != '']
print(words)
これをリストtextsに対して繰り返します。
words_list = []
for text in texts:
words = re.split('\.|,| |:|;|\(|\)|\[|\]|\?|\!', text)
words = [word for word in words if word != '']
words_list.extend(words)
最後に、大文字を小文字に変換します。長さは1760627語でした。
lower_list = list(map(str.lower, words_list))
可視化
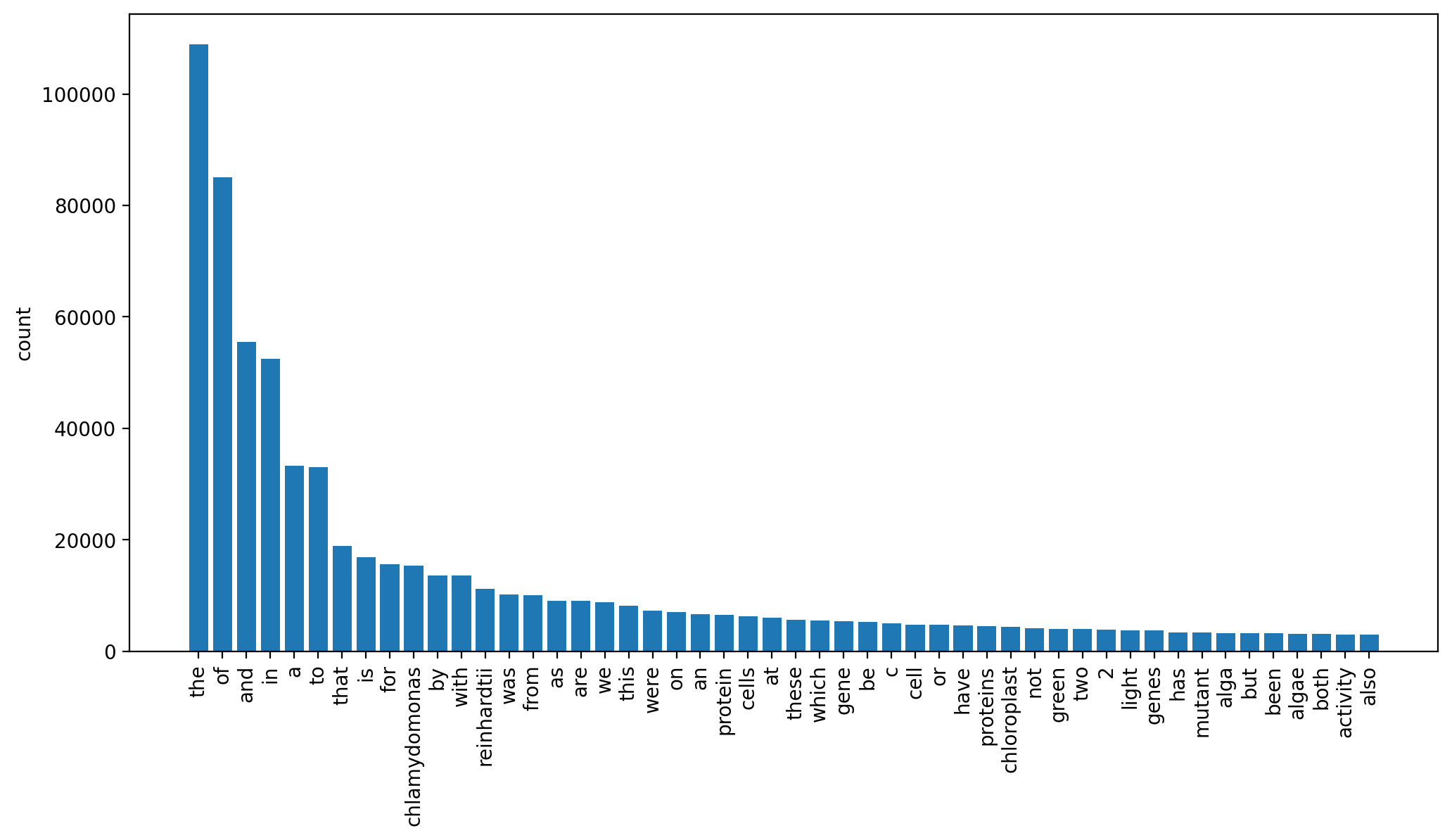
準備ができたので可視化していきます。以下、上位50位の頻度をグラフにします。
import collections
import matplotlib.pyplot as plt
words, counts = zip(*collections.Counter(lower_list).most_common())
plt.figure(figsize=[12, 6])
plt.bar(words[0:50], counts[0:50])
plt.xticks(rotation =90)
plt.ylabel('count')
plt.savefig('PubMed_bar', dpi=200, bbox_inches="tight")
the, of, and, in, a, ...という結果になったので、ストップワードを除去してみます。
ストップワード除去
nltkからストップワードを呼び出し、リストlower_listから削除します。
from nltk.corpus import stopwords
stop_words = stopwords.words('english')
lower_list2 = [word for word in lower_list if word not in stop_words]
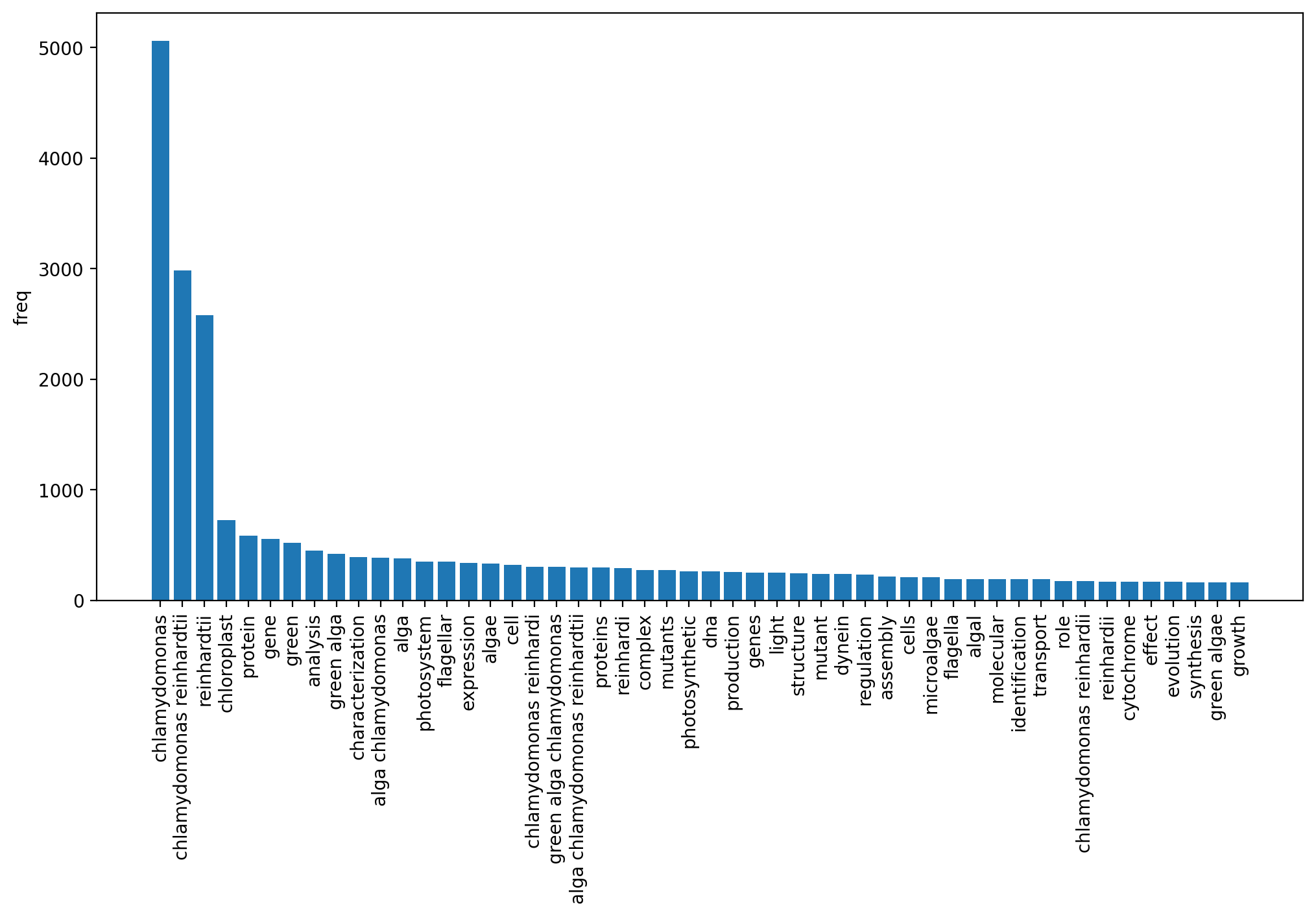
ストップワードを除去した場合の語の頻度を可視化した結果を以下に示します。
Chlamydomonasで論文検索しましたが、chlamydomonas, reinhardtiiが最も多いという当然の結果になりました(Chlamydomonas reinhardtiiの2語で種名です)。他にもprotein(s), cell(s), gene(s), chloroplastなどそれらしい単語が上位に来ています。
spaCy
ここからキーワード抽出ツールを使っていきます。
ここを参考に、spaCy, YAKE, rake-nltkの3種類を試してみました。まずspaCyです。
import spacy
nlp = spacy.load("en")
keywords_spacy = []
for text in texts:
doc = nlp(text)
keywords_spacy.extend(list(doc.ents))
keywords_spacy = [str(keyword) for keyword in keywords_spacy]
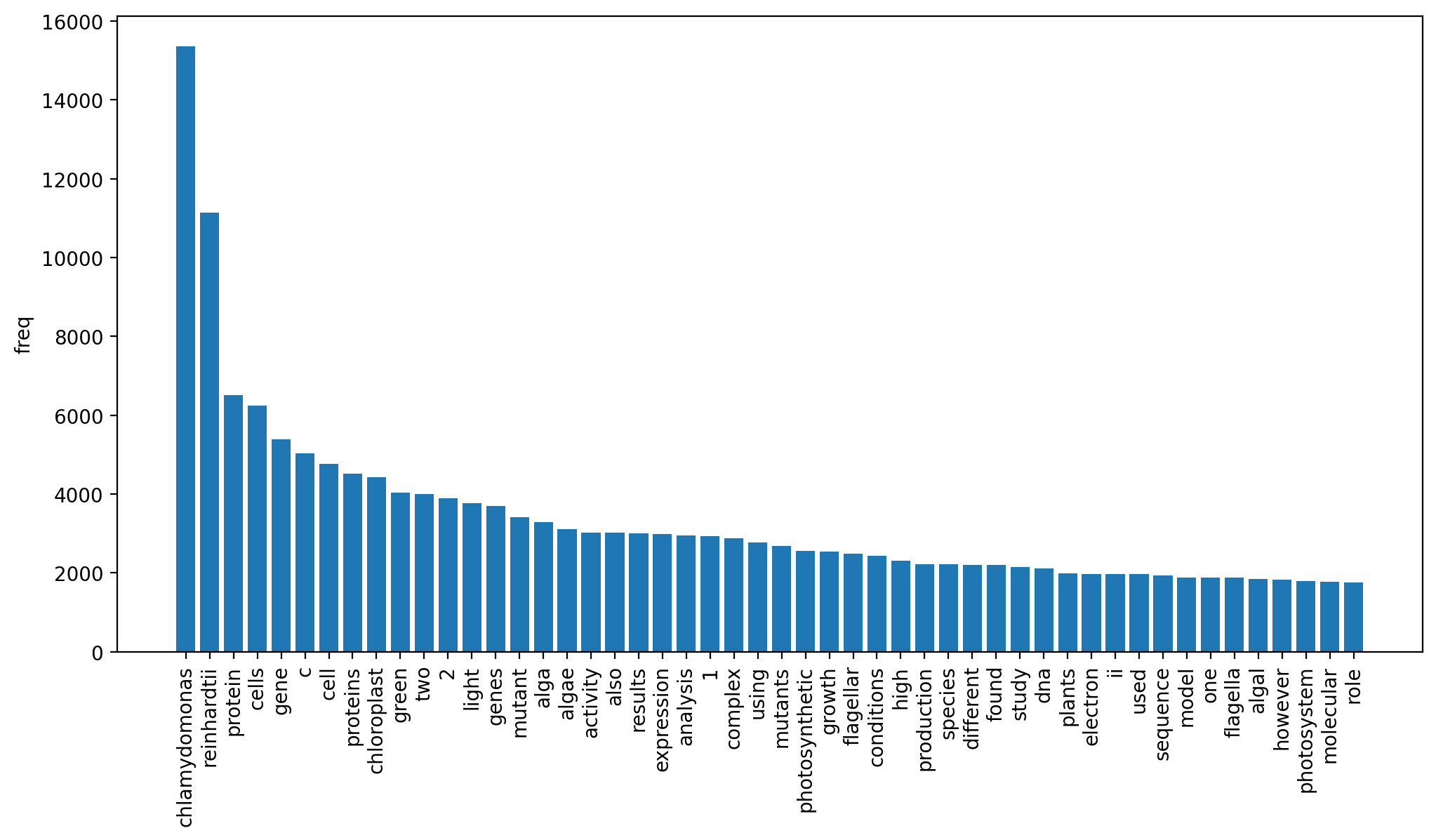
タイトルの大文字がそのまま入っているのかなと思いますが、このまま多い順に集計してみましょう。
102207キーワードが抽出されました。Chlamydomonas, C. reinhardtiiは当然入っているとして、PSII, RNA, ATP, PSIなど、大文字だけの略語が目立ちます。Arabidopsis, Escherichia, Chlorella, Volvoxなど、他の生物の属名も入っています。大文字があると重要と判定するのかもしれません。
YAKE
次にYAKEでキーワード抽出してみます。
import yake
kw_extractor = yake.KeywordExtractor()
keywords_yake = []
for text in texts:
keywords_y = kw_extractor.extract_keywords(text)
keywords_y = [t[0] for t in keywords_y]
keywords_yake.extend(keywords_y)
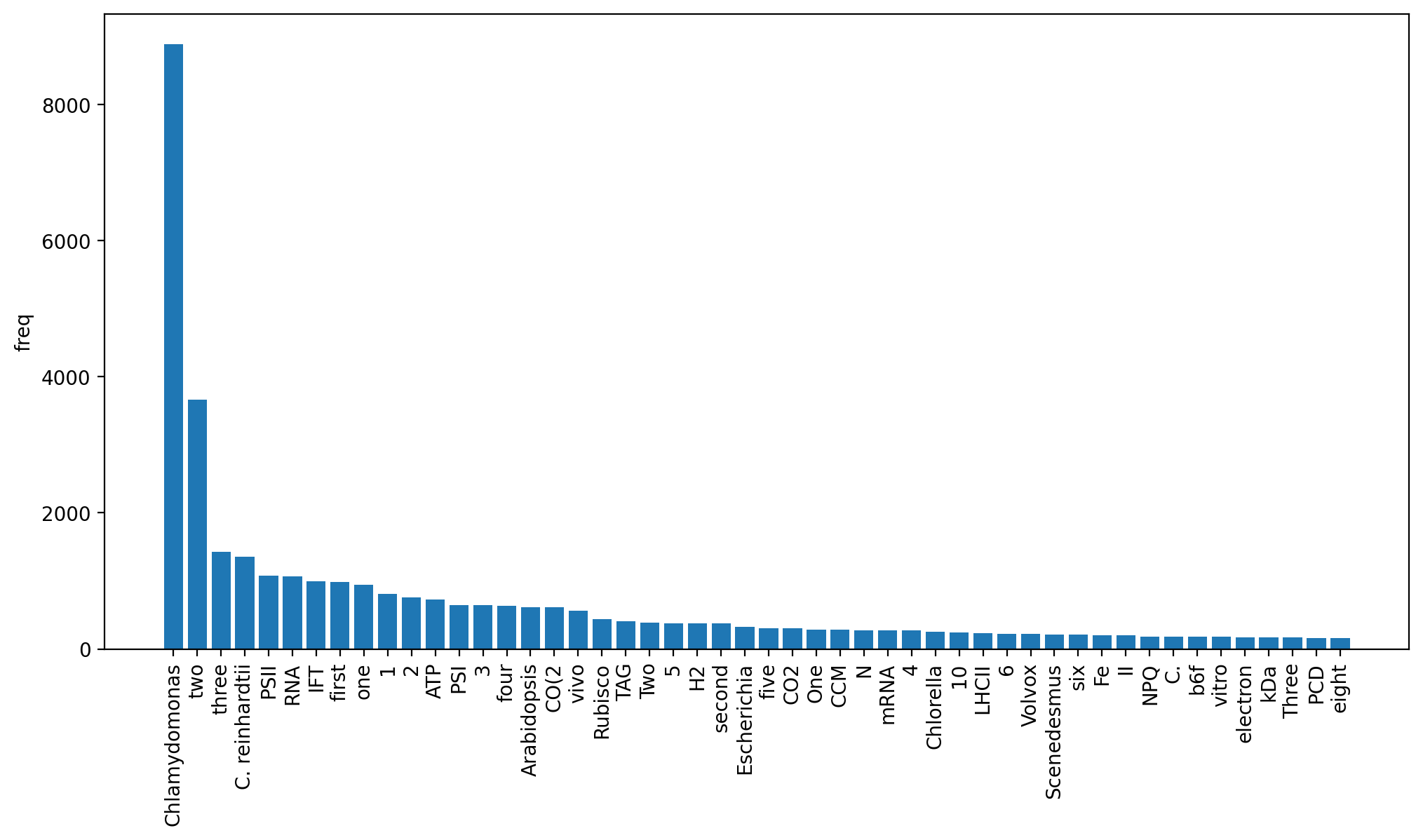
なんだか途中でValueError: max() arg is an empty sequenceが出て止まってしまい、原因が特定できなかったのですが、144272キーワードを抽出できていたのでこのまま可視化してみます。
chlamydomonas, chlamydomonas reinhardtii, reinhardtiiが上位3つを占めています。chlamydomonas reinhardiなどの表記ゆれが入っているのも特徴的です。数字は含まれていません。
rake-nltk
最後にrake-nltkです。
from rake_nltk import Rake
rake_nltk_var = Rake()
keywords_rake = []
for text in texts:
rake_nltk_var.extract_keywords_from_text(text)
keyword_extracted = rake_nltk_var.get_ranked_phrases()
keywords_rake.extend(keyword_extracted)
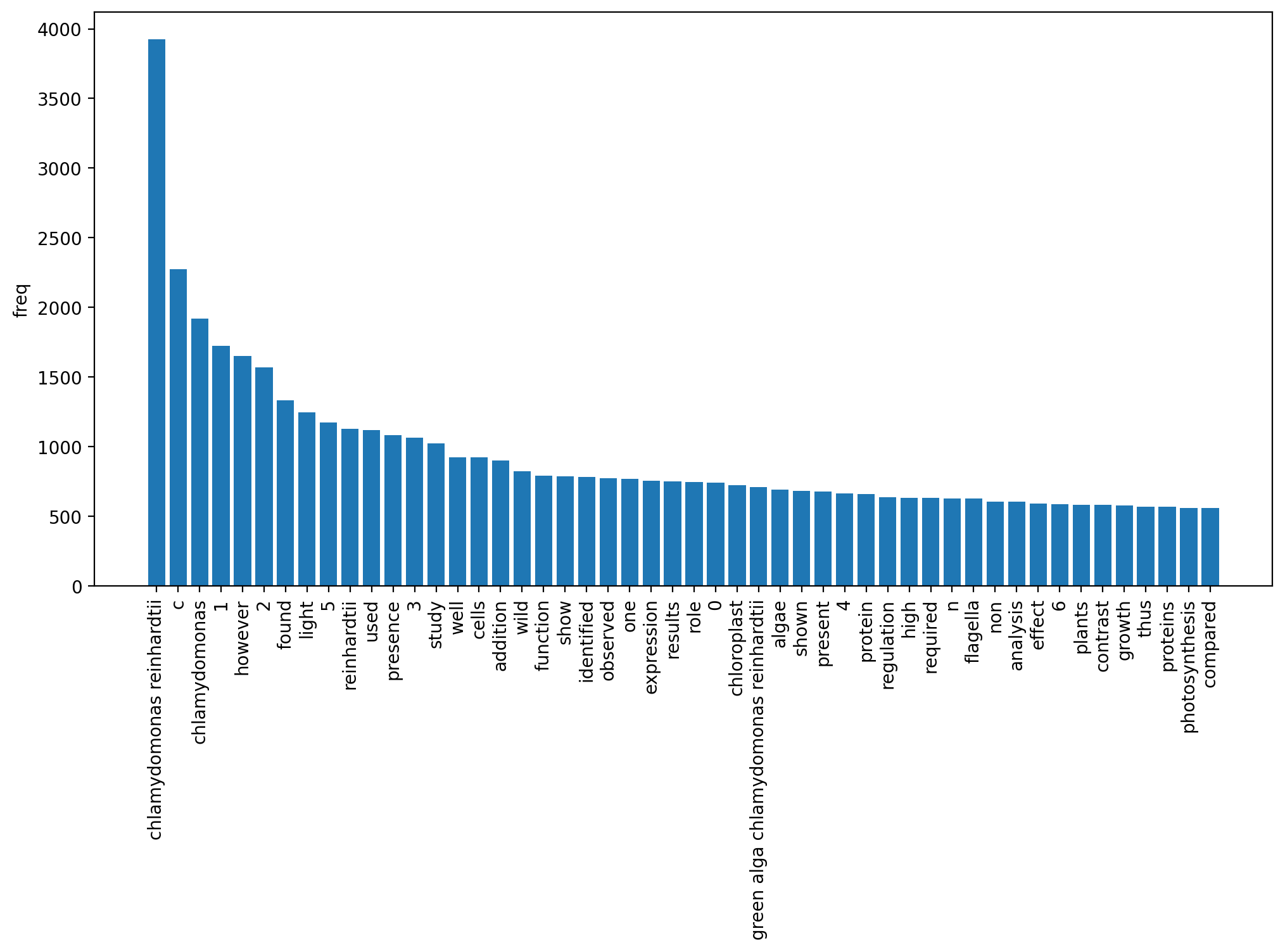
chlamydomonas reinhardtii, green alga chlamydomonas reinhardtiiはフレーズで抽出されているものの、他は単語のみが上位に来るという結果になりました。found, used, show, identified, observedなどの動詞が多いのが特徴的です。printした結果を見ると、一般性の乏しい長いフレーズが抽出される傾向があり、それで集計すると上位に単語が多くなるのかなと思います。
まとめと所感
PubMedのデータから語の頻度を可視化し、spaCy, YAKE, rake-nltkでキーワード抽出を試してみました。今回はYAKEが一番キーワードっぽい結果になっているかなと思いますが、それぞれ一長一短ありそうです。個人的には、ストップワード除去して語の頻度だけ出すで研究の動向欄を書く(助けにする)という目的には十分かなという感じもありました。
参考
- 【自然言語処理】科研費データベースからMeCab-ipadic-neologdとtermextractでキーワードを抽出する
- Biopython を利用したNCBIのEntrez データベースへのアクセス
- Keyword Extraction process in Python with Natural Language Processing(NLP)
- 自然言語のpythonでの前処理のかんたん早見表(テキストクリーニング、分割、ストップワード、辞書の作成、数値化)
- spacy.load('en')が実行できない
- Python リストの要素を違う型に変換する
- Python: リスト内の文字列を全て大文字or小文字にする
- リストの空の要素を駆逐する
- Pythonで文字列を分割(区切り文字、改行、正規表現、文字数)