PythonのRDkitライブラリを用いてSMILESから立体異性体をxyz形式で全列挙する方法を紹介します。
Python version: 3.9.12
RDKit version: 2024.03.3
RDKitが古すぎなければ正常に動作すると期待されます。Pythonのバージョンによって実行時間が変化する可能性はありますが今回は特に検証しておりません。
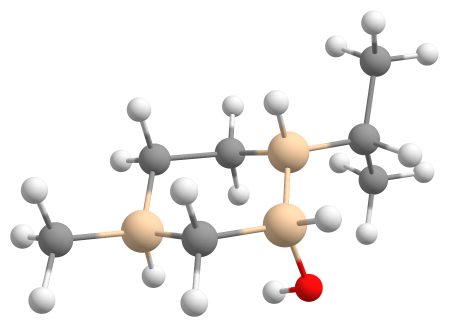
メントールの例
メントールは3個の不斉炭素を有するため、8個の立体異性体が存在します。下記プログラムにより、立体異性体を全列挙してみます。
C[C@@H]1CC[C@H]([C@@H](C1)O)C(C)C
入力するSMILESが異なると列挙される立体異性体の順序が変わりますが、立体異性体の組そのものは網羅されています。出力ファイル名は
sorted_isomers.xyzとしています。結果は記事末尾に示しています。
import os
from rdkit import Chem
from rdkit.Chem import AllChem
import itertools
input_smiles = 'C[C@@H]1CC[C@H]([C@@H](C1)O)C(C)C'
def binary_to_decimal(binary_str):
"""Convert a comma-separated binary string to an integer."""
try:
return int(''.join(binary_str.split(',')), 2)
except ValueError:
print(f"Error converting binary string to decimal: {binary_str}")
return 0
def sort_xyz_file(input_file, output_file):
"""Sort the generated XYZ file based on the binary R/S labels as integer values."""
with open(input_file, 'r') as infile:
lines = infile.readlines()
isomers = []
current_isomer = []
for line in lines:
if len(current_isomer) == 0 and line.strip().isdigit():
# Start of a new isomer block
current_isomer = [line.strip()]
elif len(current_isomer) == 1 and line.startswith('R/S:'):
# Add R/S label to the current isomer block
current_isomer.append(line.strip())
elif line.strip() == '':
# End of the current isomer block
if current_isomer:
isomers.append(current_isomer)
current_isomer = []
else:
# Add line to the current isomer block
current_isomer.append(line.strip())
# Add the last isomer block
if current_isomer:
isomers.append(current_isomer)
# Sort isomers based on the binary label as an integer
isomers.sort(key=lambda x: binary_to_decimal(x[1].split('[')[1].split(']')[0].strip()))
# Write sorted isomers to the output file
with open(output_file, 'w') as outfile:
for isomer in isomers:
for line in isomer:
outfile.write(f"{line}\n")
outfile.write("\n") # Add spacing between isomers
def main(input_smiles):
# Filepath and output file settings
filepath = os.path.join(os.path.expanduser('~'), 'Downloads') # Change to preferred path
tmp_filename = 'tmp_output_isomers.xyz'
output_filename = 'sorted_isomers.xyz'
# Target molecule SMILES
target_smiles = input_smiles # Menthol SMILES
# Generate mol object from SMILES
target_mol = Chem.MolFromSmiles(target_smiles)
# Identify chiral centers
chiral_centers = [atom.GetIdx() for atom in target_mol.GetAtoms() if atom.GetChiralTag() != Chem.rdchem.ChiralType.CHI_UNSPECIFIED]
print(f"Chiral centers: {[index + 1 for index in chiral_centers]}")
# Create a list to store isomers
isomers = set()
num_chiral_centers = len(chiral_centers)
# Generate stereoisomers
for combination in itertools.product([0, 1], repeat=num_chiral_centers):
new_mol = Chem.RWMol(target_mol)
for i, chirality in enumerate(combination):
if chirality == 1:
new_mol.GetAtomWithIdx(chiral_centers[i]).SetChiralTag(Chem.rdchem.ChiralType.CHI_TETRAHEDRAL_CCW)
else:
new_mol.GetAtomWithIdx(chiral_centers[i]).SetChiralTag(Chem.rdchem.ChiralType.CHI_TETRAHEDRAL_CW)
# Add hydrogens and optimize geometry
new_mol = Chem.AddHs(new_mol)
AllChem.EmbedMolecule(new_mol, randomSeed=42)
AllChem.UFFOptimizeMolecule(new_mol)
# Generate R/S labels
label_value = sum(bit << i for i, bit in enumerate(reversed(combination)))
r_s_labels = [str(chirality) for chirality in combination]
label_string = ','.join(r_s_labels)
# Convert to XYZ format
xyz_block = Chem.MolToXYZBlock(new_mol)
lines = xyz_block.strip().split('\n')
atom_count = lines[0]
xyz_coordinates = '\n'.join(lines[2:]) # Exclude the header lines
# Add the formatted isomer
isomers.add(f"{atom_count}\nR/S: ({label_value}) [{label_string}]\n{xyz_coordinates}\n")
# Write unsorted isomers to a temporary XYZ file
with open(os.path.join(filepath, tmp_filename), 'w') as xyz_file:
for isomer in isomers:
xyz_file.write(isomer)
xyz_file.write("\n")
# Sort the XYZ file and save the sorted file
sort_xyz_file(os.path.join(filepath, tmp_filename), os.path.join(filepath, output_filename))
os.remove(os.path.join(filepath, tmp_filename)) # Clean up temporary file
print(f"XYZ file sorted and saved as '{output_filename}'.")
if __name__ == "__main__":
main(input_smiles)
スクリプトの内容は以下の通りです。
- SMILES入力から分子オブジェクトを生成:
指定されたSMILES文字列から分子構造を生成し、不斉中心(キラル中心)を特定します。# Identify chiral centersの部分について、if atom.GetChiralTag() != Chem.rdchem.ChiralType.CHI_UNSPECIFIEDは不斉炭素でない原子(キラリティが指定されていない原子)を除外するための条件分岐として機能しています。- 異性体生成:
itertools.productを使用して、全ての可能な立体配置の組み合わせを網羅的に生成します。各組み合わせについて、不斉炭素のキラルタグ(CWまたはCCW)を設定します。ここでCWは"clockwise"(時計回り)、CCWは"counterclockwise"(反時計回り)を意味します。- 水素の追加とエネルギー最適化:
分子構造に水素原子を追加し、EmbedMoleculeとUFFOptimizeMoleculeを使用して簡便にエネルギーを最適化した構造を得ます。- ラベル付けと座標生成:
各異性体に対して2進表記のR/Sラベルを生成します。分子の3D座標をxyz形式で取得し、座標データを整形します。- 一時ファイルへの書き込み:
異性体ごとのデータ(ラベルと座標)を一時ファイルに保存します。- 異性体のソート:
R/Sラベルを十進数に変換して異性体をソートします。ラベルはZero-based numbering(ゼロ始まり)としています。- ソートされたデータを保存して出力:
ソートされた異性体のデータを最終xyzファイルとして保存し、処理結果(異性体の数と保存先ファイル名)を出力します。
実行すると Chiral centers: [2, 5, 6] と出力されるはずです。これは2, 5, 6番目の原子が不斉炭素であることを意味しています。
入力するSMILESが異なる場合は不斉中心となる炭素原子の順番が変わるため、出力される立体異性体の順序が変化します。
2進表記のR/Sラベルを反転させる(全ての0と1を入れ替える)操作は鏡映操作に対応するため、例えば R/S: (0) [0,0,0] のエナンチオマー(鏡像異性体)は R/S: (7) [1,1,1] に相当します。また、R/S: (0) に対して R/S: (7) 以外との組はジアステレオマーに対応します。
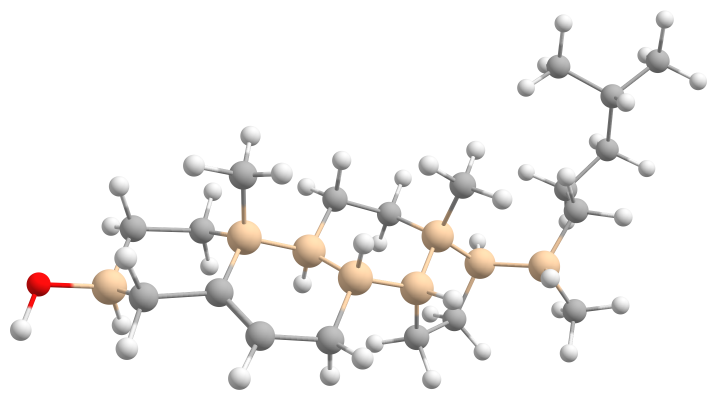
コレステロールの例
コレステロール(Cholesterol)には8個の不斉炭素が存在するため、256個の立体異性体が存在します。
C[C@H](CCCC(C)C)[C@H]1CC[C@@H]2[C@@]1(CC[C@H]3[C@H]2CC=C4[C@@]3(CC[C@@H](C4)O)C)C
このSMILESを input_smiles に入力して実行すると、Chiral centers: [2, 9, 12, 13, 16, 17, 21, 24] と出力され、計256個の立体異性体が得られます。(出力結果は長大なため省略)
※ 不斉炭素数に応じて指数的に実行時間が増大するので注意。
※ SMILESは例えばPubChemなどのデータベースから取得できます。
参考資料
31
R/S: (0) [0,0,0]
C 3.666624 -0.451215 -0.089647
C 2.234151 0.088819 -0.011500
C 1.407198 -0.410813 -1.210703
C -0.037494 0.098006 -1.140472
C -0.715769 -0.347440 0.175870
C 0.097133 0.108636 1.420529
C 1.575892 -0.326585 1.321945
O -0.012794 1.488143 1.671582
C -2.233282 0.023367 0.268787
C -3.097412 -0.978526 -0.514145
C -2.569162 1.454773 -0.188524
H 4.267795 -0.064192 0.760652
H 3.671376 -1.562029 -0.055854
H 4.149490 -0.119669 -1.033664
H 2.298307 1.197803 -0.059341
H 1.867086 -0.049174 -2.156329
H 1.405182 -1.523389 -1.236570
H -0.595679 -0.311748 -2.008784
H -0.033861 1.204638 -1.233238
H -0.660374 -1.460658 0.191669
H -0.328692 -0.412176 2.306316
H 1.626464 -1.433553 1.426791
H 2.141962 0.114241 2.171656
H 0.394240 1.977224 0.913030
H -2.544291 -0.062488 1.333874
H -2.929678 -2.010831 -0.139750
H -2.860446 -0.946029 -1.598324
H -4.174975 -0.742510 -0.380739
H -2.395315 1.577815 -1.278410
H -3.640168 1.671150 0.012706
H -1.973508 2.208412 0.360587
31
R/S: (1) [0,0,1]
C 3.459622 -0.530203 0.032562
C 2.181193 0.289861 -0.188444
C 1.350365 -0.289079 -1.355811
C -0.118430 0.121874 -1.248749
C -0.778805 -0.511587 0.003469
C 0.163081 -0.502930 1.260144
C 1.365701 0.457545 1.123116
O 0.602463 -1.799380 1.586932
C -2.192330 0.076306 0.315125
C -3.194757 -0.213500 -0.812853
C -2.186987 1.580999 0.640043
H 4.103165 -0.037491 0.792281
H 3.223791 -1.557211 0.378650
H 4.036758 -0.597602 -0.914419
H 2.512035 1.308486 -0.494015
H 1.763994 0.082875 -2.318742
H 1.414266 -1.398983 -1.382538
H -0.648901 -0.217246 -2.164424
H -0.168795 1.229734 -1.217713
H -0.953459 -1.580960 -0.255880
H -0.410187 -0.153718 2.145644
H 2.022068 0.358344 2.015556
H 0.970836 1.496954 1.151257
H 1.026409 -2.194108 0.783608
H -2.577058 -0.451574 1.216650
H -4.217221 0.086672 -0.498325
H -2.933848 0.349784 -1.733490
H -3.213712 -1.299301 -1.046058
H -1.970071 2.191058 -0.260819
H -3.186309 1.883063 1.019919
H -1.444880 1.821319 1.427326
31
R/S: (2) [0,1,0]
C 3.701383 0.106318 0.365415
C 2.178960 0.203137 0.515507
C 1.556571 0.939292 -0.680563
C 0.046887 1.172769 -0.476698
C -0.649902 0.065801 0.368569
C 0.080943 -1.294226 0.214074
C 1.549416 -1.201162 0.694314
O -0.569409 -2.293011 0.961182
C -2.190243 -0.023920 0.117554
C -2.576008 -0.510969 -1.291423
C -2.890283 1.308616 0.425312
H 4.137192 -0.421509 1.240443
H 3.970290 -0.447340 -0.559853
H 4.146526 1.122958 0.316179
H 1.972595 0.802016 1.431543
H 2.056703 1.921582 -0.828985
H 1.715476 0.338937 -1.604205
H -0.413526 1.267455 -1.479989
H -0.095371 2.149369 0.036241
H -0.530426 0.355211 1.438414
H 0.103614 -1.567650 -0.868373
H 2.138611 -1.965052 0.139485
H 1.609776 -1.469514 1.772584
H -1.149937 -2.801412 0.337061
H -2.604778 -0.762320 0.838983
H -2.280636 0.215486 -2.074794
H -2.117467 -1.494245 -1.516716
H -3.677560 -0.642587 -1.351018
H -2.602921 2.089346 -0.310407
H -3.992921 1.178193 0.384843
H -2.623557 1.658431 1.445320
31
R/S: (3) [0,1,1]
C 3.683001 0.047586 -0.154392
C 2.185090 0.247902 0.103182
C 1.410974 0.342420 -1.224097
C -0.094487 0.625439 -1.016736
C -0.607576 0.252368 0.400967
C 0.080019 -1.030320 0.945499
C 1.626675 -0.885161 0.999913
O -0.280337 -2.177398 0.214585
C -2.164243 0.253114 0.551165
C -2.900773 -0.829258 -0.258945
C -2.754670 1.635544 0.228777
H 4.231677 -0.005775 0.810141
H 3.862375 -0.890528 -0.722466
H 4.088626 0.901808 -0.737423
H 2.070649 1.216039 0.640670
H 1.844558 1.150889 -1.853144
H 1.536532 -0.607461 -1.788810
H -0.657332 0.079994 -1.800343
H -0.273599 1.709918 -1.183834
H -0.260711 1.069179 1.075192
H -0.274731 -1.187770 1.987853
H 2.093498 -1.856849 0.722913
H 1.934725 -0.677194 2.048486
H 0.068075 -2.081149 -0.707825
H -2.390287 0.059325 1.623707
H -3.999029 -0.700408 -0.150223
H -2.659958 -0.774964 -1.338949
H -2.669220 -1.841485 0.125924
H -2.671255 1.858917 -0.855975
H -3.828906 1.667468 0.510737
H -2.229359 2.427810 0.803452
31
R/S: (4) [1,0,0]
C 3.599667 -0.626878 -0.489910
C 2.073651 -0.653541 -0.345796
C 1.429466 0.490048 -1.150215
C -0.115190 0.456296 -1.101083
C -0.681417 -0.344396 0.103329
C 0.187225 -0.161427 1.378702
C 1.662750 -0.591674 1.146559
O 0.131370 1.153905 1.875350
C -2.215243 -0.158601 0.345471
C -3.030278 -0.593930 -0.883290
C -2.643087 1.255154 0.779672
H 4.013703 0.332611 -0.111895
H 3.885362 -0.743894 -1.557072
H 4.051259 -1.464137 0.083917
H 1.720098 -1.619102 -0.772376
H 1.755892 0.430513 -2.211921
H 1.792953 1.464277 -0.755632
H -0.485756 -0.008526 -2.040617
H -0.481171 1.502390 -1.091347
H -0.568588 -1.419986 -0.165833
H -0.229996 -0.822596 2.169856
H 2.335401 0.101474 1.699656
H 1.820212 -1.600349 1.588635
H 0.542660 1.757337 1.205865
H -2.502713 -0.847620 1.171068
H -2.728789 -1.611614 -1.210876
H -2.886584 0.115414 -1.725742
H -4.112318 -0.624690 -0.632794
H -2.313185 2.028423 0.058168
H -3.750596 1.309317 0.850932
H -2.256759 1.495802 1.789219
31
R/S: (5) [1,0,1]
C 3.684178 -0.308200 -0.182188
C 2.160139 -0.418280 -0.303677
C 1.543744 0.931548 -0.705544
C 0.030898 0.807891 -0.978558
C -0.669488 -0.299630 -0.137701
C 0.063111 -0.509488 1.216271
C 1.537179 -0.945593 1.012974
O -0.607807 -1.436029 2.034515
C -2.208430 -0.078049 0.021056
C -2.907378 0.041631 -1.341867
C -2.584552 1.114247 0.919081
H 3.963481 0.443183 0.587469
H 4.115393 -1.291898 0.101716
H 4.124682 -0.003999 -1.155605
H 1.943141 -1.149692 -1.114959
H 2.043553 1.324499 -1.618133
H 1.711347 1.668465 0.111537
H -0.117628 0.578248 -2.056681
H -0.424639 1.801796 -0.796661
H -0.552675 -1.252451 -0.704103
H 0.078318 0.463925 1.753552
H 2.122664 -0.589022 1.889602
H 1.610929 -2.055396 1.007455
H -0.672142 -2.288852 1.529745
H -2.628700 -0.987967 0.503974
H -2.635436 -0.817329 -1.991630
H -2.624485 0.985697 -1.853809
H -4.010134 0.033949 -1.206652
H -2.220476 2.074995 0.503336
H -3.689681 1.179501 1.012694
H -2.179107 0.982300 1.942789
31
R/S: (6) [1,1,0]
C 3.453084 -0.422116 0.598727
C 2.203844 -0.041582 -0.206913
C 1.408843 1.090857 0.490669
C 0.265350 0.546827 1.358678
C -0.763241 -0.289205 0.539592
C -0.133117 -0.930420 -0.735989
C 1.339751 -1.295554 -0.485407
O -0.841201 -2.094948 -1.077400
C -2.087781 0.500943 0.275606
C -3.174404 -0.377221 -0.362594
C -1.903770 1.790471 -0.543318
H 3.171739 -0.778295 1.612882
H 4.013852 -1.227049 0.077144
H 4.125821 0.456031 0.700819
H 2.555075 0.354493 -1.186328
H 0.985672 1.761331 -0.286835
H 2.078317 1.720756 1.116498
H -0.229151 1.389140 1.888842
H 0.710801 -0.095639 2.150499
H -1.046611 -1.134237 1.208994
H -0.168307 -0.209478 -1.582887
H 1.385537 -2.000495 0.375320
H 1.749771 -1.828470 -1.371401
H -0.628506 -2.295355 -2.026440
H -2.485262 0.807985 1.269278
H -4.150617 0.152979 -0.342770
H -2.928690 -0.608404 -1.420532
H -3.289704 -1.326062 0.203277
H -1.513356 1.579950 -1.559167
H -2.880771 2.307776 -0.654041
H -1.222969 2.494992 -0.024803
31
R/S: (7) [1,1,1]
C 3.667157 -0.443082 -0.106721
C 2.150929 -0.521382 -0.318003
C 1.576028 0.879649 -0.597601
C 0.055521 0.828788 -0.788490
C -0.636698 0.225533 0.455757
C -0.070470 -1.181439 0.798984
C 1.469890 -1.160117 0.911691
O -0.502734 -2.178863 -0.093454
C -2.200086 0.255494 0.393305
C -2.792693 -0.178515 -0.959959
C -2.742971 1.644632 0.766219
H 3.910466 0.195987 0.769329
H 4.082041 -1.459295 0.064694
H 4.157498 -0.016946 -1.007955
H 1.976317 -1.163236 -1.208949
H 2.037657 1.293876 -1.520532
H 1.819083 1.567238 0.243021
H -0.172886 0.235919 -1.699307
H -0.310882 1.863349 -0.958389
H -0.341898 0.869108 1.316848
H -0.458405 -1.461348 1.803149
H 1.839029 -2.200647 1.045120
H 1.749789 -0.587929 1.824482
H -0.138772 -1.974037 -0.991149
H -2.589130 -0.448037 1.162849
H -3.899671 -0.234816 -0.883837
H -2.542466 0.551130 -1.758846
H -2.433140 -1.181124 -1.259615
H -2.412289 2.410694 0.033792
H -3.853860 1.630271 0.785277
H -2.392355 1.939143 1.778291