Roary
本稿はPart.1の続きになります。
Roary公式サイト: https://sanger-pathogens.github.io/Roary/
ゲノムデータベースを作成する
パンゲノムはPart1で説明したように、要はゲノム同士の比較なのでデータベース的なものが必要になる。例えば、Bacillus subtilisのシーケンスをしたとしたら、従来シーケンスされたBacillus subtilisと比較すると、そのstrainは過去の研究対象とはどこが違うのかを考察できる。なので、各自の研究対象のゲノムデータを一括ダウンロードして、データベースを作りたいとなるわけである。ただ、ゲノムデータを一つ一つダウンロードして、一つ一つアノテーションをさせるのは死ぬほどめんどくさい。例えば、当方は80種のゲノムデータと自分のドラフトゲノムを比較したが、そんなことを自分でやるのはめんどくさすぎる。そこで、今回はパッとゲノムを落とし、ダァーっとアノテーションする方法を紹介する。
ncbi-genome-downloadの使い方
基本的な使い方は上坂先生が記載してくださっている。色々と使い方があるが、基本的には枠を指定して一括ダウンロードという感じである。枠というのはバクテリアという大きな括りを指定することもできるし、Bacillus subtilisのように細かい設定までできる。今回はBacillus subtilisでやると宣言したので、この使い方を説明する。また、ncbi-genome-downloadはRefSeqとGenbankの両方からデータを落とせる。違いについては井上先生が書いてくださっていた。今回はRefSeqを利用する。
Bacillus subtilisというスペシフィックな菌のゲノムデータを落とすにはtaxid (多分Taxonomy IDのこと) が必要になる。Googleで"Bacillus subtilis taxid" 調べてみる。
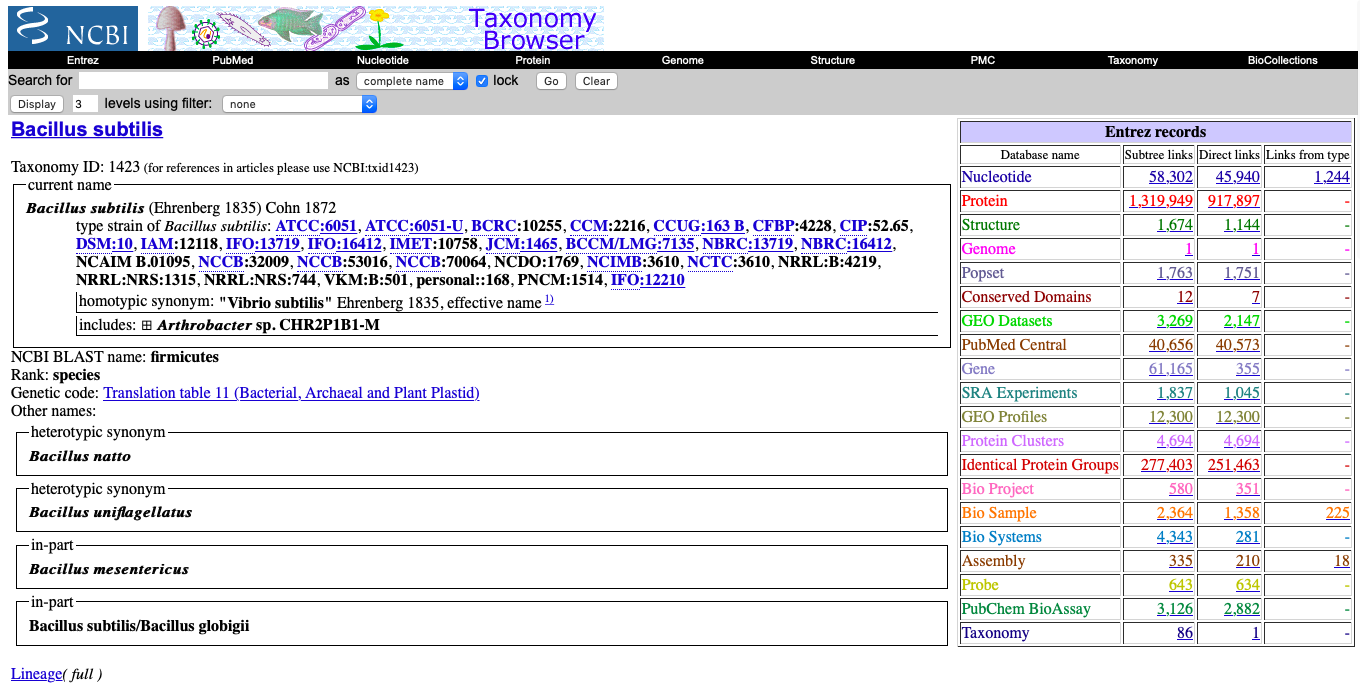
図1. NIHのサイト
するとNCBIのTaxonomy Browserが出てきて、左上にtaxidが書かれていることがわかる。したがって、今回はTaxonomy ID: 1423を利用する。いきなりゲノムを落として想像より量が多いとかは困るので、数を推定するためにどれくらいのゲノムがTaxonomy ID: 1423を利用すると落とせるのか確かめる。--dry-runを利用するとテキストファイルで事前に中身を知ることができる。
ncbi-genome-download --dry-run --taxid 1423 bacteria > list.txt
すると、約190ゲノムがダウンロードされるようである。当方は今Macbook air 128GBを使っている。軟弱マイPCにバクテリアとはいえ190個のゲノムを落とすのは涙が出る。そこで、今回はAssemblyレベルをCompleteにしてダウンロードするゲノム数を80まで減らすことにした。Prokkaの利用を考えて、formatはFASTAにしておくと良い。
ncbi-genome-download --dry-run -l complete --taxid 1423 bacteria > list.txt
ncbi-genome-download --format fasta -l complete --taxid 1423 bacteria
ダウンロード終了後に一度絶望する。理由はカレントディレクトリにFASTAファイルが落とされていくわけではなく (実際にダウンロードされるのはgz圧縮ファイル) 、RefSeq > Bacteria > GCF_****** などという感じに保存されるため、FASTAを一個一個ディレクトリ移動しないといけないためである。ただ、安心してほしい。MacユーザーならFinderを利用して、簡単に移動できる。Finder上でRefSeq > Bacteriaと移動し、.fna.gzとして検索をかける。これをコピーするなり、そのまま移動するなりして希望のフォルダに移せば良い。そのディレクトリに対して
$ gunzip *.gz
しておく。Prokkaでここで出てくるFastaファイルをアノテーションする。
Prokka
gunzipと同様に*を使って、ディレクトリ内のファイル全てをProkkaでアノテーションしていきたいが、、、
prokka *.fna
としてもうまくいかない。Prokkaはアウトプットディレクトリの名前がかぶるとデータ出力ができないので、一つ一つアノテーションする作業が必要になる。つまり、ファイル一つ一つに対して別のアウトプットディレクトリ名が必要となるということ。ここでもう一つ裏技を行う。for文の利用である。今回データベースとして利用する全てのFASTAファイルを指定し、COMMAND+Cでコピーする。これを標準テキスト化したテキストエディットに貼る。すると、ファイル名が改行されて記載できる。
改行区切りだと使えないので、間をタブ区切りに変えていくこれを改行区切りからタブ区切りに変えるには以下のようなウェブサイトを利用するといい。
http://tma.main.jp/copy/csv.php#output
今回なら以下のテキストになった。
GCF_000789295.1_ASM78929v1_genomic.fna GCF_002216085.1_ASM221608v1_genomic.fna GCF_002269175.1_ASM226917v1_genomic.fna GCF_002269195.1_ASM226919v1_genomic.fna GCF_002893805.1_ASM289380v1_genomic.fna GCF_002982175.1_ASM298217v1_genomic.fna GCF_003148355.1_ASM314835v2_genomic.fna GCF_003184225.1_ASM318422v1_genomic.fna GCF_003612735.1_ASM361273v1_genomic.fna GCF_004101345.1_ASM410134v1_genomic.fna GCF_004101365.1_ASM410136v1_genomic.fna GCF_004101405.1_ASM410140v1_genomic.fna GCF_004101445.1_ASM410144v1_genomic.fna GCF_004101465.1_ASM410146v1_genomic.fna GCF_004101485.1_ASM410148v1_genomic.fna GCF_004101565.1_ASM410156v1_genomic.fna GCF_004103535.1_ASM410353v1_genomic.fna GCF_004103555.1_ASM410355v1_genomic.fna GCF_004103595.1_ASM410359v1_genomic.fna GCF_004119535.1_ASM411953v1_genomic.fna GCF_004119595.1_ASM411959v1_genomic.fna GCF_004119615.1_ASM411961v1_genomic.fna GCF_004119635.1_ASM411963v1_genomic.fna GCF_004119655.1_ASM411965v1_genomic.fna GCF_006364495.1_ASM636449v1_genomic.fna GCF_008831405.1_ASM883140v1_genomic.fna GCF_009363835.1_ASM936383v1_genomic.fna GCF_009497795.1_ASM949779v1_genomic.fna GCF_009662155.1_ASM966215v1_genomic.fna GCF_009662175.1_ASM966217v1_genomic.fna GCF_009662195.1_ASM966219v1_genomic.fna GCF_009662215.1_ASM966221v1_genomic.fna GCF_009662255.1_ASM966225v1_genomic.fna GCF_009662275.1_ASM966227v1_genomic.fna GCF_009662335.1_ASM966233v1_genomic.fna GCF_009662355.1_ASM966235v1_genomic.fna GCF_009662415.1_ASM966241v1_genomic.fna GCF_009662435.1_ASM966243v1_genomic.fna GCF_009662455.1_ASM966245v1_genomic.fna GCF_009665145.1_ASM966514v2_genomic.fna GCF_002290305.1_ASM229030v1_genomic.fna GCF_003610955.1_ASM361095v1_genomic.fna GCF_004101425.1_ASM410142v1_genomic.fna GCF_004101945.1_ASM410194v1_genomic.fna GCF_004119555.1_ASM411955v1_genomic.fna GCF_004119675.1_ASM411967v1_genomic.fna GCF_009497815.1_ASM949781v1_genomic.fna GCF_009662235.1_ASM966223v1_genomic.fna GCF_009662375.1_ASM966237v1_genomic.fna
これでfor文が使える。for文は同じ作業を繰り返しやるときに非常に便利な文法である。使いこなせると爆速で作業が終えられるので、当方は多用する。for文は以下のように書く。
$ for X in 入れたい文字
do
コマンド ${X}
done
${X}に入れたい文字が入っていき、最終的に同じ作業を何度も繰り返せる。早速今回のパターンをやってみる。
$ for X in GCF_000789295.1_ASM78929v1_genomic.fna GCF_002216085.1_ASM221608v1_genomic.fna GCF_002269175.1_ASM226917v1_genomic.fna GCF_002269195.1_ASM226919v1_genomic.fna GCF_002893805.1_ASM289380v1_genomic.fna GCF_002982175.1_ASM298217v1_genomic.fna GCF_003148355.1_ASM314835v2_genomic.fna GCF_003184225.1_ASM318422v1_genomic.fna GCF_003612735.1_ASM361273v1_genomic.fna GCF_004101345.1_ASM410134v1_genomic.fna GCF_004101365.1_ASM410136v1_genomic.fna GCF_004101405.1_ASM410140v1_genomic.fna GCF_004101445.1_ASM410144v1_genomic.fna GCF_004101465.1_ASM410146v1_genomic.fna GCF_004101485.1_ASM410148v1_genomic.fna GCF_004101565.1_ASM410156v1_genomic.fna GCF_004103535.1_ASM410353v1_genomic.fna GCF_004103555.1_ASM410355v1_genomic.fna GCF_004103595.1_ASM410359v1_genomic.fna GCF_004119535.1_ASM411953v1_genomic.fna GCF_004119595.1_ASM411959v1_genomic.fna GCF_004119615.1_ASM411961v1_genomic.fna GCF_004119635.1_ASM411963v1_genomic.fna GCF_004119655.1_ASM411965v1_genomic.fna GCF_006364495.1_ASM636449v1_genomic.fna GCF_008831405.1_ASM883140v1_genomic.fna GCF_009363835.1_ASM936383v1_genomic.fna GCF_009497795.1_ASM949779v1_genomic.fna GCF_009662155.1_ASM966215v1_genomic.fna GCF_009662175.1_ASM966217v1_genomic.fna GCF_009662195.1_ASM966219v1_genomic.fna GCF_009662215.1_ASM966221v1_genomic.fna GCF_009662255.1_ASM966225v1_genomic.fna GCF_009662275.1_ASM966227v1_genomic.fna GCF_009662335.1_ASM966233v1_genomic.fna GCF_009662355.1_ASM966235v1_genomic.fna GCF_009662415.1_ASM966241v1_genomic.fna GCF_009662435.1_ASM966243v1_genomic.fna GCF_009662455.1_ASM966245v1_genomic.fna GCF_009665145.1_ASM966514v2_genomic.fna GCF_002290305.1_ASM229030v1_genomic.fna GCF_003610955.1_ASM361095v1_genomic.fna GCF_004101425.1_ASM410142v1_genomic.fna GCF_004101945.1_ASM410194v1_genomic.fna GCF_004119555.1_ASM411955v1_genomic.fna GCF_004119675.1_ASM411967v1_genomic.fna GCF_009497815.1_ASM949781v1_genomic.fna GCF_009662235.1_ASM966223v1_genomic.fna GCF_009662375.1_ASM966237v1_genomic.fna
do
prokka ${X} -o ${X}_out
done
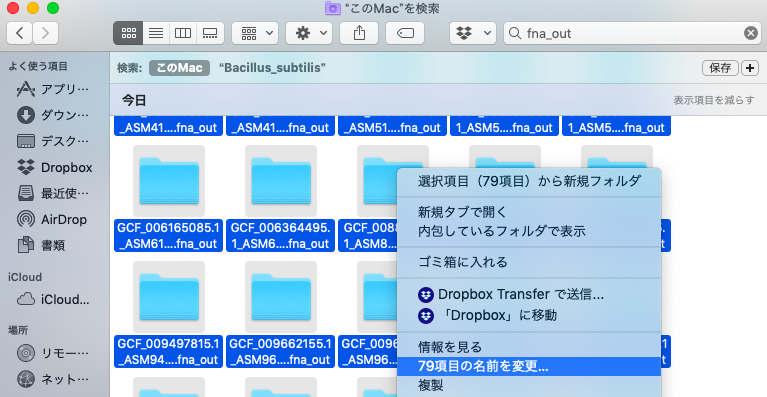
すると、フォルダはこのようになり、非常にいい感じである。
図4. いい感じになったフォルダ
フォルダ・ファイル名の変更 (今回はやらないで)
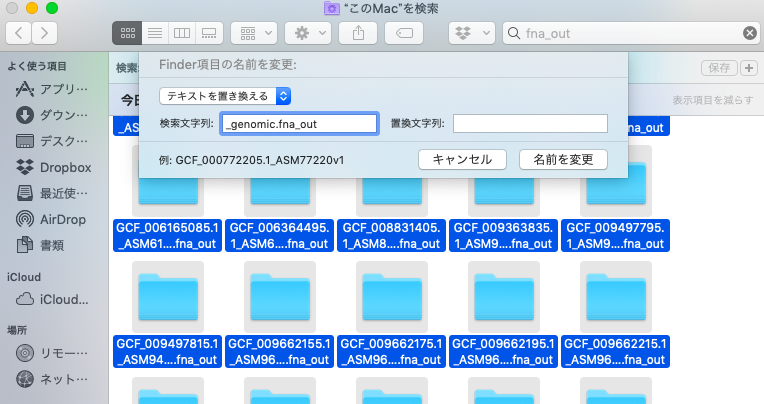
名前を変えたい場合は最終的に変えられる。Part3でもfor構文から始まるので、今回は変えてはいけないが変え方を紹介する。やり方はまず全ての名前を変えたいファイルを指定する。そして、optionとクリックを押して、名前の変更を押す。
名前が共通する部分は消したり、変更したりできる。今回なら例えば_genomic.fna_outとかを消すと綺麗になりそうであろう。再度確認するが、次はProkkaでアノテーションしたGFFファイルの名前の変更から行うので名前を変えないでほしい。
Part3ではRoaryを実際に動かし、SNPの検討、系統樹作成を行う。。次もできるだけ早く更新します!
Roaryシリーズ
Part.1: https://qiita.com/danryo_official/items/fe9273f13368ac5ad780
Part.2: https://qiita.com/danryo_official/items/3e9dc463daf9e35870e7
Part.3: https://qiita.com/danryo_official/items/11353fdf2700c9b6ffe3