公開されているコロナウィルス関係データとフリーウェアのR言語を用いてグラフを描く話第2段。今回は「組みグラフ」を作りたい。
おさらい
4月25日公開の第1弾記事『コロナウィルスデータを解析してみよう : R言語入門』は、R言語の簡単な紹介と「折れ線グラフ」を描くまでの必要最小限の話であった。第1弾記事をまだ読んでない方はそちらを先にチェックしていただきたい。
まずは同記事にて紹介したデータ"Data on COVID-19 by Our World in Data : owid/covid-19-data"をダウンロードしよう。前記事で述べたようにデータは毎日更新されている。
次に、Rスクリプトを再度掲出する。
library(ggplot2)
coviddata <- read.csv("owid-covid-data.csv", header = T, stringsAsFactors = F)
coviddata <- subset(coviddata, date>"2020-03-01")
coviddata$date <- as.Date(coviddata$date)
countries <- c("Japan", "United States", "United Kingdom", "France", "Italy", "Germany", "Spain", "South Korea", "China")
c_total_death <- subset(coviddata, location %in% countries, select=c("date","location","total_deaths"))
g <- ggplot(c_total_death, aes(x=date, y=total_deaths, color=location)) + geom_line()
plot(g)
RStudioを起動し、当該スクリプトを読み込んで実行し、グラフが表示されているところまでを確認した上で、当記事を読み勧めていただければ幸いである。
2つのグラフを新たに描く
第1弾では国別の「死者の累積人数」を取り上げた。
しかし、規模が様々である国家間の被害程度の比較に使うには少し問題がある。「総人口10万人の国で1千人亡くなりました」と「総人口2億人の国で1千人亡くなりました」とでは、同じ「1千人」でも深刻度が全く違うと言わねばならないだろう。両国の被害の度合いを比較するなら、「人口X人当たりの死者」を比較するべきだ。
また、各国の医療体制がうまくいっているのかは、「患者数と死者数の比」を見るのがもっとも直接的であるように思える。
この二種類のグラフを今回新たに作成してみたい。
「人口X人当たりの死者」
当該の公開データには人数を100万人当たりに換算した値も含まれている。total_cases_per_million ・new_cases_per_million・ total_deaths_per_million・new_deaths_per_millionなどだ。
ありがたく使わせてもらおう。
「患者数と死者数の比」=死亡率
残念ながら、こちらは元データに直接的には記述されていない。こちらで割り算を実行して用意するしかないだろう。
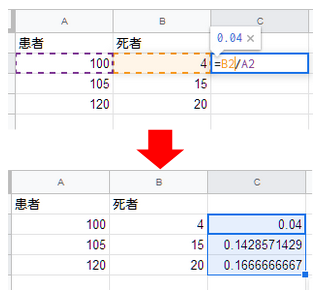
エクセルやグーグルのスプレッドシートシートなら、「死者」「患者」の項から次のように計算式を立てて「死亡率」の項を追加することになるだろう。
R言語でもこれと本質的に同じことができる。たった1行の式を追加するだけでいい。
coviddata$death_rate <- coviddata$total_deaths/coviddata$total_cases
これを前掲スクリプトの4行目、coviddata$dateを再定義した直後あたりに挿入すればOKである1。
組グラフを作る
データをバラバラに見るのは疲れるので、できれば上記2セットのデータを一つにまとめたグラフを作りたい。正確には、死亡率のグラフと死者のグラフを、日付を揃えて縦に並べたい。
ggplot2はこういった要求にスマートに応えられるように作られている。
グラフ用のデータセットを作る
元データcoviddataから、下のような形式のデータを作る。
元データではそれぞれ独立した列として格納されている「死亡率」と「100万人あたり死者数」の数値をdataという一つの列にまとめる。そして、それぞれがどちらの項目のデータなのかを区別するためにdatatype列を追加している。
Date Country data datatype
3029 2020-03-01 Germany 0.000000000 sr
4249 2020-03-01 France 0.020000000 sr
6561 2020-03-01 Japan 0.020920502 sr
6937 2020-03-01 South Korea 0.004821327 sr
12346 2020-03-01 United States 0.014492754 sr
30291 2020-03-01 Germany 0.000000000 tdpm
42491 2020-03-01 France 0.031000000 tdpm
65611 2020-03-01 Japan 0.040000000 tdpm
69371 2020-03-01 South Korea 0.332000000 tdpm
123461 2020-03-01 United States 0.003000000 tdpm
[2020-06-03追記] library(tidyverse)が使えるなら、この処理は簡単である。白状すると、私は最初にtidyverseのルーツたるtidyrの説明を見たとき今ひとつピンとこず、それ以来、食わず嫌いで勉強を避けていたのだが、誤った姿勢であった。この種の統計データを使いまくる仕事をするなら、早い段階でtidyverseをマスターすべきだ。
具体的には、次のような感じになるだろう。
library(tidyverse)
c_subset <- subset(coviddata, location %in% countries, select = c("date", "location", "total_deaths_per_million", "death_rate"))
c_tdpm_sr <- pivot_longer(c_subset, cols=c("total_deaths_per_million", "death_rate"))
以上が追記。この記述どおりにうまくいったなら、この下の旧記述は読み飛ばしてもらって構わない。すぐに「いよいよグラフを描く」節に移動していただきたい。
これより旧記述。
ここで少しややこしいのは、Date、Countryは独立した列として維持しておきたい点だ。
最初に提示したスクリプトで作成したデータcoviddataを対象として次のような処理を追加する。
c_tdpm <- subset(coviddata, location %in% countries, select = c("date","location","total_deaths_per_million"))
c_sr <- subset(coviddata, location %in% countries, select = c("date","location","death_rate"))
colnames(c_sr) <- c("Date","Country","data")
colnames(c_tdpm) <- c("Date","Country","data")
c_sr$datatype <- "sr"
c_tdpm$datatype <- "tdpm"
c_tdpm_sr <- rbind(c_sr, c_tdpm)
「死亡率」と「100万人あたり死者数」を、まず独立した2つのサブセットとして作成し、最後に連結している。
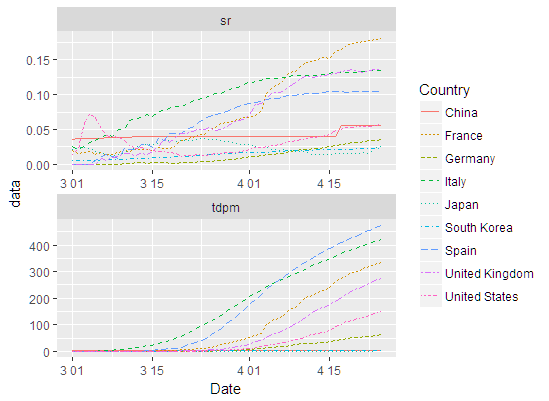
いよいよグラフを描く
g1 <- ggplot(c_tdpm_sr, aes(x=Date, y=data, color=Country, linetype=Country)) +
geom_line() +
facet_wrap(~datatype,
ncol = 1,
scales = "free"
)
plot(g1)
関数facet_wrapが組グラフ作成で重要な役割を担っている。ここでは
- datatype列の内容に基づいてデータを分割する
- 子グラフを作ってそれらを1行あたり1列で(つまり縦一列に)配置する
- 縮尺はそれぞれの子グラフで独自に設定する
という指示を行っている。
結果として次のようなグラフが得られるだろう。
続きは
次はグラフの見栄えを整える
とりあえずグラフはできた。しかし、一人で眺めている分には良いが、これを外に出す(レポートやwebページやポスターなどに貼り付ける)となると、ちょっと検討の余地があるだろう。ざっと次のような不満が出てくるはずだ。
- 線が細すぎて見にくい
- 色分けが今ひとつ。国別の識別がしにくい。できれば日本だけもっと目立たせられない?
- srとかtdpmとか意味不明
- Y軸には「死者数」とかラベルが要るよね
ggplotにはいろいろなオプションが用意されていて、それらを追加していくことによってグラフの見栄えを様々に調節できる。次回はそのあたりを追求してみたい。
-
R言語の文法や仕様について説明すると長くなってしまうので、ここでは「Rの式は常に値の集合(ベクトル)」を対象にしているとだけ述べておこう。この仕様故に、ひとつの式だけで全行のデータが自動的に追加されるのだ。 ↩