やること
Roaryはパンゲノム解析パイプラインで、Prokkaなどのアノテーションツールから出てくるGFF3ファイルを用いてパンゲノムを計算する。簡単に言えば、遺伝子レベルでのゲノム比較を簡単にできるということである。今回はデータインポートから解析まで一連の流れを3章に分けてこなしてみる。
Roary公式サイト: https://sanger-pathogens.github.io/Roary/
パンゲノム
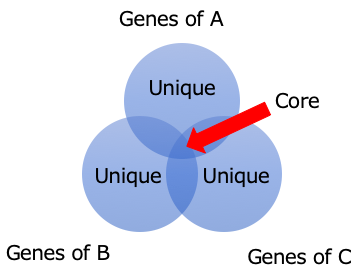
まず、パンゲノムは小麦のゲノムのことではない。ある集団における全ての遺伝子セットのことを指す。このパンゲノムには大きく2つの要素があり、コアゲノムとアクセサリーゲノムである。1つ目のコアゲノムは集団に共通する遺伝子セットのことを指す。つまり、ある種で比較したならば、その系統群に必要不可欠 (生存に必要という意味ではなくて、その種たる所以という意味) な遺伝子とも言える。対してアクセサリーゲノムはユニークゲノムと表現されることもあるが、集団において一部、もしくはわずかな数のサンプルが保有する遺伝子セットである。Wikipediaには'dispensable' genesと書いてあったが、特定の株の所以とも言えるところであろうか。
なぜパンゲノム解析をするかと言えば、進化を考えられる、遺伝子セットで見るので個別細菌を見るよりも様々な組成がわかるというような点に落ち着くと思う。ゲノムアセンブリをする理由は特定の菌を詳しく考えるということだと考えているが、その点では他者とは何が違うのかという視点が必要になる。その際にパンゲノムの概念が必要になってくるんだろうと青二才ながら偉そうなことを書いてみる。当方は進化論的なところから、ある菌の形質を探りたくて勉強した。
手順
Roaryを動かすにはざっくり4段階必要になる。
- ゲノムデータの準備
- Prokka (DFAST) によるアノテーション (GFFファイル作成)
- Roaryを動かす
- 作図
一番めんどくさいのはRoaryのところである。扱うゲノム数が多いほど、時間がとてもかかる。Roaryは超高速パンゲノム計算ツールだが、それでも数時間はかかる。DFASTとProkkaではアノテーション名が異なる場合があるので、どちらかで全てのゲノムをアノテーションして比較すべきだろうと考えている。Prokkaが推奨で、実際比較後の名前などがProkkaの方が綺麗。ただ、DFASTでも現在のところ問題なく動いている。
ゲノムデータの準備にはncbi-genome-downloadを利用。アノテーションではProkkaを使用する。Roaryを動かして、最終的にPythonとRで作図という感じで行きたい。全てのソフトウェアはminiconda条件で動くので、事前にminicondaを入れておくこと。さらに、以下ソフト、環境を構築しているものとする。ただし、代替できるソフトは山ほどあるので、各自好みがあればそれでいいと思う。
・SnapGene
・MEGA X
・R studio
インストール
ncbi-genome-download
$ conda install -y -c bioconda ncbi-genome-download
参考: https://github.com/kblin/ncbi-genome-download/blob/master/README.md
Prokka
conda install -c conda-forge -c bioconda -c defaults prokka
参考: https://github.com/tseemann/prokka
Roary
conda config --add channels r
conda config --add channels defaults
conda config --add channels conda-forge
conda config --add channels bioconda
conda install roary
参考: https://github.com/sanger-pathogens/Roary/blob/master/README.md#installation
RoaryはRequired dependenciesとして
・bedtools
・cd-hit
・ncbi-blast+
・mcl
・parallel
・prank
・mafft
・fasttree
がある。Condaを使っていれると多分勝手にインストールされる。mafftとかはインストールした記憶がないのに、conda listに含まれているので多分そうだと思う。Krakenはオプションとして入っている。Krakenはインストールされないことを確認した。ただ、Condaで入れられる。Krakenの稼働には別に色々必要らしい。(参照: http://kazumaxneo.hatenablog.com/entry/2017/08/28/235248) 今回は利用しないので、インストールの必要はない。
なお、RoaryはDockerでも利用できる。もしも、うまくいかない点があればGithubに詳しく記載されているので、ぜひ一度見て欲しい。これで準備完了。Part2で、ncbi-genome-downloadとprokkaを利用してGFFファイルを作成し、Part3でRoaryを動かし、作図をしていこうと考えている。
Roaryシリーズ
Part.1: https://qiita.com/danryo_official/items/fe9273f13368ac5ad780
Part.2: https://qiita.com/danryo_official/items/3e9dc463daf9e35870e7
Part.3: https://qiita.com/danryo_official/items/11353fdf2700c9b6ffe3