- English article available at here
概要
「Deep Neural Networkはブラックボックス、中で何が行われて出力が出たのか知ることはできない」と思っていませんか?
実際、多くの層からなるNNは複雑で数学的解析は難しいですが、全くわからないというわけではなく、上記のように予測根拠を可視化することによってモデルのデバッグや新たな知見の導出を試みようという研究があります。
今回、このSaliency mapの計算手法のいくつかを実装し、その可視化機能がChainer Chemistry でマージされましたので紹介します。
本記事少し長くなってしまいましたが、最後まで読んでいただければこのような可視化ができるようになりますのでぜひ使ってみてください!
始めに理論、手法の簡単な説明を入れましたが、そんなものはいいから使ってみたい・結果が見たいという方は、実例まで読み飛ばしてください。
実験に用いたコードはgithubで公開してます。
NNの予測根拠可視化とは?
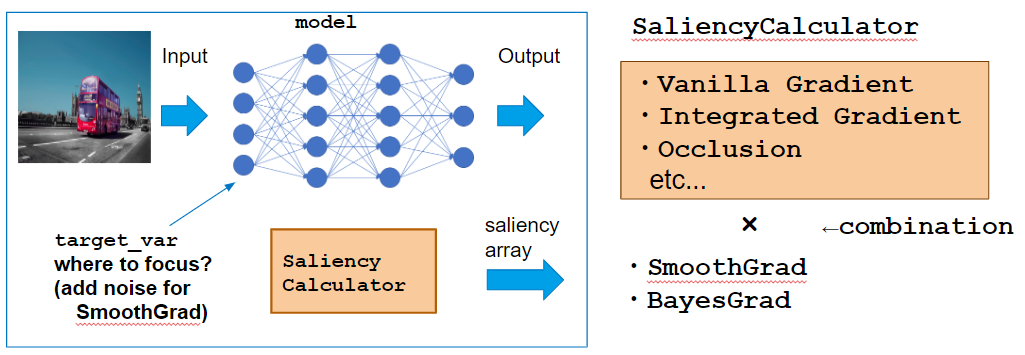
今回実装した手法は以下3つです。
これら手法は、各Dataに対してModelの予測の寄与度を算出する手法です。
※ Random forest, XGBoostなどで使われるFeature importanceは”モデルに対して”算出される重要度であり、
データ点は必要ないという部分に違いがあります。
手法の概要説明 - VanillaGrad
今回は一番簡単なVanillaGradの手法のみ簡単に説明をします。
この手法では output y の input x に対する微分値を計算することで、 input の寄与度を計算します。
s_i = \frac{dy}{dx_i}
各inputデータの$i$ 番目の要素 $x_i$ に対して、$s_i$ がSaliency score として計算されます。
Gradient の値が大きい要素ほど、その要素を少し動かすだけで出力が大きく変わってしまう
→ 影響度(saliency) が大きい、ということになります。
実装としては、Chainer 上では以下の計算をするだけです。
y = model(x)
y.backward()
s = x.grad
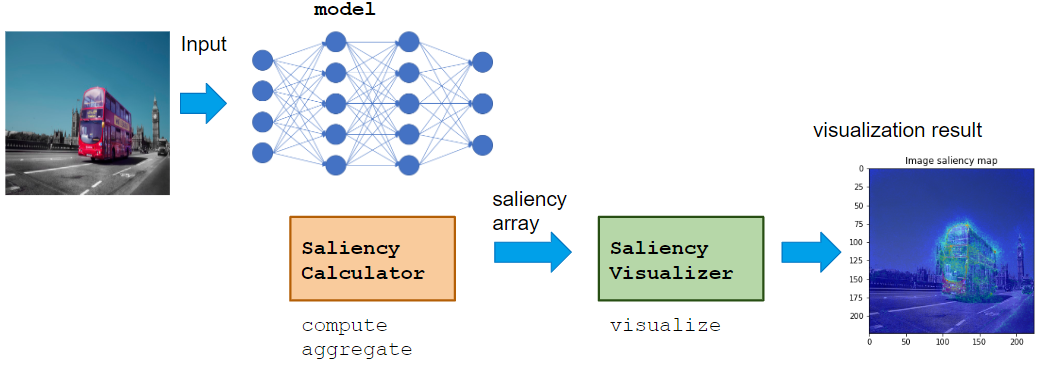
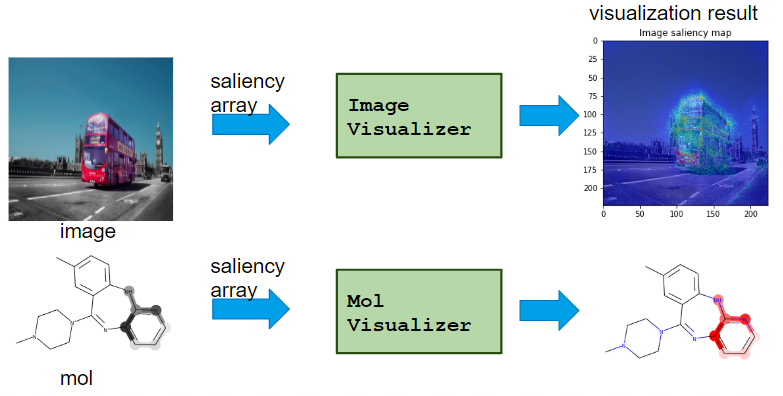
Saliency module の使い方
Calculator class が VaillaGrad, IntegratedGradient, Occlusion などの saliency score の算出を担当し、 Visualizer class がその可視化を担当するという設計にしています。
Calculatorは、NNのモデルや適用ドメインにかかわらず使えるようになっており、VisualizerをApplicationに合ったものを用いることでドメインごとに最適な可視化をすることができます。
使い方の基本的な流れは以下のように Calculatorでcompute, aggregate -> Visualizerでvisualize という形で呼んでいくことで saliency の計算、可視化を行うことができます。
# model is chainer.Chain, x is dataset
calculator = GradientCalculator(model)
saliency_samples = calculator.compute(x)
saliency = calculator.aggregate(saliency_samples)
visualizer = ImageVisualizer()
visualizer.visualize(saliency)
Calculator class
ここでは上記で説明したVanillaGrad を計算するための GradientCalculator を例にして各Methodの呼び方を見ていきます。
Instance化
Instance化する際に、Saliencyを計算したい model を渡します。
calculator = GradientCalculator(model)
compute method
compute method でデータ x に対して saliency samples を計算します。
# x (bs, num_feature) -> saliency_samples (M, bs, num_feature)
saliency_samples = calculator.compute(x, M=1)
ここで、M sample のsaliency を計算することができます。
VanillaGradの計算時にはgradの計算結果は毎回同じのため M=1でよいのですが、
下記に示すSmoothGradやBayesGradの計算ではSamplingが必要となります。
ここでちょっと寄り道をしてSmoothGrad とBayesGradの説明をします。
- SmoothGrad -
VanillaGrad の適用を実際に行うとNoisyな saliency map が得られやすいということから、
input x を $\epsilon$ だけ微小変化させた x + $ \epsilon$ でのgrad を計算しその平均値を最小値
とするという手法です。
s_{mi} = \frac{dy}{dx_i} |_{x=x+\epsilon_m}
s_{i} = \frac{1}{M} \sum_{m=1}^{M}{s_{mi}}
本ライブラリでは compute method で各 saliency sample $s_{mi}$ を計算し、 aggregate methodで saliency $s_i = \frac{1}{M} \sum_{m}^{M} s_{mi}$ を計算します。
- project page: https://pair-code.github.io/saliency/
- BayesGrad -
SmoothGradでは入力 x にGaussian noiseを加えた分布上でのSamplingを考えましたが、
BayesGradでは学習データD を 学習した後のNeural NetworkのParameter $\theta$ に対する予測分布 $y_\theta \sim p(\theta|D)$ 上でのSamplingを考えます。
式で書くと以下のような形になります。
s_{mi} = \frac{dy_\theta}{dx_i} |_{\theta \sim p(\theta|D)}
s_{i} = \frac{1}{M} \sum_{m=1}^{M}{s_{mi}}
aggregate method
compute method で計算されたM個の saliency sample $s_{mi}$ から saliency を計算するための method です。
# saliency_samples (M, bs, num_feature) -> saliency (bs, num_feature)
saliency = calculator.aggregate(saliency_samples, method='raw')
Aggregate の方法は論文によって異なっており、現在以下の3つのmethodをサポートしています。
'raw': 平均値をとります。
$$s_i = \frac{1}{M} \sum_{m}^{M} s_{mi}$$
'abs': 絶対値の平均をとります。
$$s_i = \frac{1}{M} \sum_{m}^{M} |s_{mi}|$$
'square': 二乗平均をとります。
$$s_i = \frac{1}{M} \sum_{m}^{M} s_{mi}^2$$
Visualizer class
Calcualtor class で計算された saliency を可視化するためのclassです。
-
TableVisualizer: テーブルデータの各列の重要度をプロット -
ImageVisualizer: 画像データのsaliency map をプロット -
MolVisualizer: 分子データのsaliency mapをプロット
というように適用するドメインごとのVisualizerを用意しています。
visualize method
Visualizerは visualize methodを呼んで可視化を行います。
注意する点はCalculatorで計算された saliency は batch size 分の計算がされていますが、
可視化は特定のデータごとに行うため、その指定が必要です。
# Instantiation
visualizer = ImageVisualizer()
# Visualize `i`-th data
i = 0
visualizer.visualize(saliency[i])
可視化したデータを保存したい場合は save_filepath を指定することで保存できます。
# Save saliency map
visualizer.visualize(saliency[i], save_filepath='saliency.png')
実例
前置きが長くなりました。。早速実際に使ってみましょう!
テーブルデータでFeature importanceを算出
Neural NetworkはMLP (Multi Layer Parceptron)、Datasetは sklearn で提供されている iris データセットを使ってみます。
iris データセットは3つの花の種類 'setosa', 'versicolor', 'virginica' を、 4つの特徴量 'sepal length (cm)', 'sepal width (cm)', 'petal length (cm)', 'petal width (cm)' から分類するというデータセットです。
# model
from chainer.functions import relu, dropout
from chainer_chemistry.models.mlp import MLP
from chainer_chemistry.models.prediction.classifier import Classifier
def activation_relu_dropout(h):
return dropout(relu(h), ratio=0.5)
out_dim = len(iris.target_names)
predictor = MLP(out_dim=out_dim, hidden_dim=48, n_layers=2, activation=activation_relu_dropout)
classifier = Classifier(predictor)
# dataset
import sklearn
from sklearn import datasets
import numpy as np
from chainer_chemistry.datasets.numpy_tuple_dataset import NumpyTupleDataset
iris = datasets.load_iris()
# All dataset is to train for simplicity
dataset = NumpyTupleDataset(iris.data.astype(np.float32), iris.target.astype(np.int32))
train = dataset
Modelの訓練部分は省略します。こちらにコードがあります。
Modelの訓練後、実際にsaliency module を使ってみます。
まずCalculator を用いて compute -> aggregate でsaliencyを計算します。
from chainer_chemistry.saliency.calculator.gradient_calculator import GradientCalculator
# 1. instantiation
gradient_calculator = GradientCalculator(classifier)
# 2. compute
saliency_samples_vanilla = gradient_calculator.compute(train, M=1)
# 3. aggregate
saliency_vanilla = gradient_calculator.aggregate(
saliency_samples_vanilla, ch_axis=None, method='square')
次にVisualizerの visualize methodを使って可視化をしてみます。
from chainer_chemistry.saliency.visualizer.table_visualizer import TableVisualizer
from chainer_chemistry.saliency.visualizer.common import normalize_scaler
visualizer = TableVisualizer()
# Visualize saliency of `i`-th data
i = 0
visualizer.visualize(saliency_vanilla[i], feature_names=iris.feature_names,
scaler=normalize_scaler)
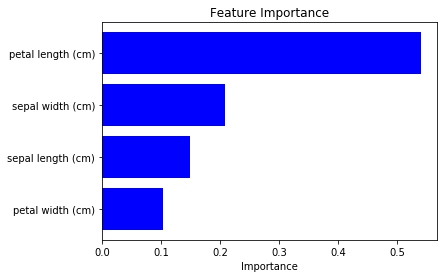
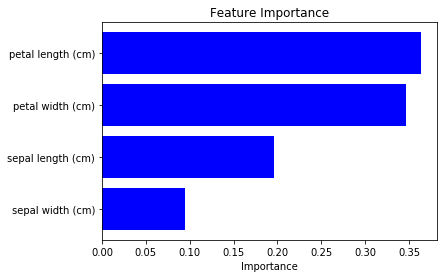
このように、それぞれの特徴量がどのくらい予測誤差に影響しているかを可視化することができます。
上の例では 0番目のデータに対するsaliency を見てみましたが、
データセット全体での平均としてそれぞれの特徴量の影響度を計算することで、
Feature importance(もどき)を可視化することもできます。
saliency_mean = np.mean(saliency_vanilla, axis=0)
visualizer.visualize(saliency_mean, feature_names=iris.feature_names, num_visualize=-1,
scaler=normalize_scaler)
今回は petal lengthとpetal width がより重要という結果になりました。
(Modelの訓練状況によって結果がある程度異なっていたので注意が必要そうです。)
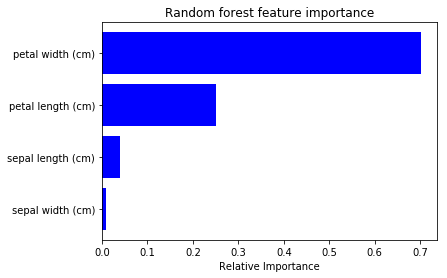
上記結果がどのくらい妥当なのか、sklearn のRandom Forestを用いたFeature importanceをプロットしてみると以下のようになりました。
(コードはこちら)
それぞれの重要度は違いますが、大まかな順序は一致しているようです。
SaliencyはNNを用いた際の特徴量選択等でも使えるかもしれません。
画像データで分類モデルの着目箇所を可視化
CNNの学習は時間がかかるので、pre-trained model を使います。今回はChainerで提供されている VGG16 モデルを用いてみます。
from chainer.links.model.vision.vgg import VGG16Layers
predictor = VGG16Layers()
これだけで自動で学習済みモデルのパラメータもダウンロードしてくれます。
ImageNetの正解ラベルの対応表はこちらからダウンロードしてきました。
import numpy as np
with open('imagenet1000_clsid_to_human.txt') as f:
lines = f.readlines()
def extract_value(s):
quote_str = s[s.index(':') + 2]
return s[s.find(quote_str)+1:s.rfind(quote_str)]
classes = np.array([extract_value(line) for line in lines])
classes は以下のような 1000クラスの正解ラベルとなります。
array(['tench, Tinca tinca', 'goldfish, Carassius auratus',
'great white shark, white shark, man-eater, man-eating shark, Carcharodon carcharias',
'tiger shark, Galeocerdo cuvieri', 'hammerhead, hammerhead shark',
'electric ray, crampfish, numbfish, torpedo', 'stingray', 'cock', ...
推論に用いる画像データはPexelsで、CC0で提供されている以下の3つの画像を用意しました。
- [Basketballの画像](https://www.pexels.com/photo/brown-basketball-on-grey-floor-945471/) - [バスの画像](https://www.pexels.com/photo/black-and-white-traffic-red-street-45923/) - [犬の画像](https://www.pexels.com/photo/photographer-animal-photography-dog-58997/)まずは予測してみます。
from PIL import Image
import numpy as np
import chainer
import chainer.functions as F
# basketball, bus, dog
image_paths = ['./input/pexels-photo-945471.jpeg', './input/pexels-photo-45923.jpeg',
'./input/pexels-photo-58997.jpeg']
imgs = [Image.open(fp) for fp in image_paths]
x = xp.asarray([chainer.links.model.vision.vgg.prepare(img) for img in imgs])
with chainer.using_config('train', False):
result = predictor.forward(x, layers=['prob'])
prob = result['prob']
lables_pred = np.argsort(cuda.to_cpu(prob.array), axis=1)[:, ::-1]
for i in range(len(lables_pred)):
print('i', i, 'labels_pred', lables_pred[i, :5], classes[lables_pred[i, :5]])
i 0 classes ['basketball' 'punching bag, punch bag, punching ball, punchball'
'rugby ball' 'barrel, cask' 'barbell']
i 1 classes ['trailer truck, tractor trailer, trucking rig, rig, articulated lorry, semi'
'passenger car, coach, carriage'
'streetcar, tram, tramcar, trolley, trolley car'
'fire engine, fire truck' 'trolleybus, trolley coach, trackless trolley']
i 2 classes ['basenji' 'Pembroke, Pembroke Welsh corgi' 'Ibizan hound, Ibizan Podenco'
'dingo, warrigal, warragal, Canis dingo' 'kelpie']
ということで、1番目の画像はきちんとBasketballと予測できていますが、2番目の画像はtrailer truck ということで乗用車ではあるが bus ではなく間違っています。3番目の画像はbasenjiという犬の種類がTopとなりました。(ImageNetの正解ラベルは犬の種類が多く、正解かどうか自分にはわかりません!笑)
VanillaGrad
それではSaliencyの計算に移りましょう。今回は"Top predictionのラベルを予測した根拠"の可視化をしたいと思います。2番目の画像の例であれば、なぜ・画像のどこを見て trailer truck と予測したのかを可視化します。実際にその予測が合っているかどうかは関係ありません。
そのためにoutput_var として "Top predictionラベルとのsoftmax cross entropy" を計算します。
import chainer.functions as F
from chainer import cuda
def eval_fun(x):
result = predictor(x, layers=['fc8'])
out = result['fc8']
xp = cuda.get_array_module(out.array)
labels_pred = xp.argmax(out.array, axis=1).astype(xp.int32)
loss = F.softmax_cross_entropy(out, labels_pred)
return loss
ここまで準備ができたら後は Calculatorで compute -> aggregate, ImageVisualizer で visualize するだけで可視化結果を見ることができます。
from chainer_chemistry.saliency.calculator.gradient_calculator import GradientCalculator
# 1. instantiation
gradient_calculator = GradientCalculator(predictor, eval_fun=eval_fun, device=device)
# --- VanillaGrad ---
# 2. compute
saliency_samples_vanilla = gradient_calculator.compute(x)
# 3. aggregate
saliency_vanilla = gradient_calculator.aggregate(
saliency_samples_vanilla, ch_axis=2, method='abs')
# saliency_samples (1, 3, 3, 224, 224) -> M, minibatch, ch, h, w
print('saliency_samples', saliency_samples_vanilla.shape)
# saliency (3, 224, 224) -> minibatch, h, w
print('saliency', saliency_vanilla.shape)
aggregate をする際に ch_axis を指定していますが、通常の(minibatch, ch, h, w) という画像 shape に sampling_axis が足されているため、 axis=2がch_axis となっていることに注意してください。
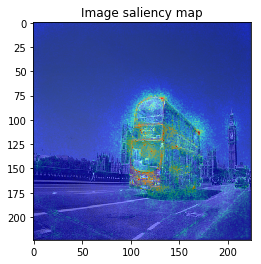
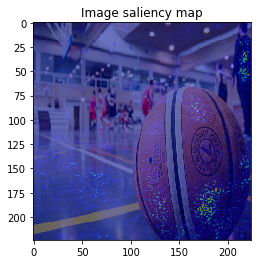
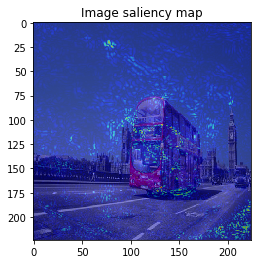
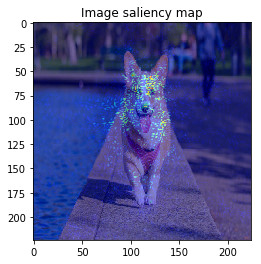
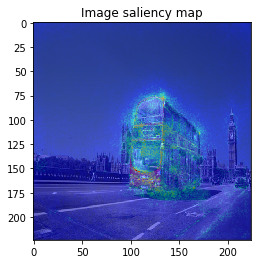
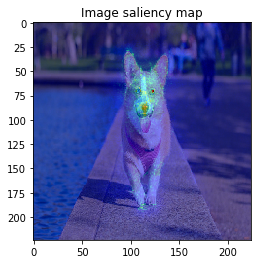
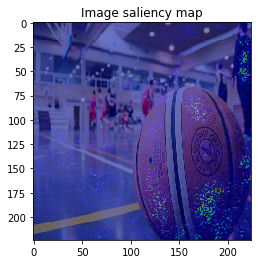
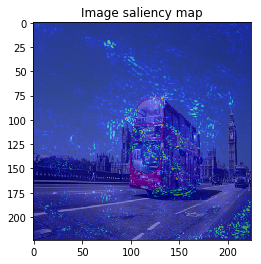
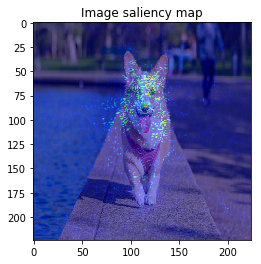
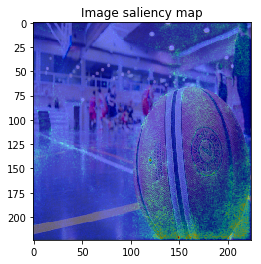
ImageVisualizerで可視化してみると以下のような結果となりました。
from chainer_saliency.visualizer.image_visualizer import ImageVisualizer
visualizer = ImageVisualizer()
for index in range(len(saliency_vanilla)):
image = imgs[index].resize(saliency_vanilla[index].shape)
visualizer.visualize(saliency_vanilla[index], image, show_colorbar=False)
何となくそれぞれの物体にきちんと着目できていそうですが、、、バスの画像などは特に飛び飛びで少し見づらいですね。
SmoothGrad
次はSmoothGradを計算してみましょう。これは、Calculatorのcompute method に noise_sampler を指定することで計算することができます。
from chainer_chemistry.saliency.calculator.common import GaussianNoiseSampler
M = 30
# --- SmoothGrad ---
# 2. compute
saliency_samples_smooth = gradient_calculator.compute(x, M=M, noise_sampler=GaussianNoiseSampler())
# 3. aggregate
saliency_smooth = gradient_calculator.aggregate(
saliency_samples_smooth, ch_axis=2, method='abs')
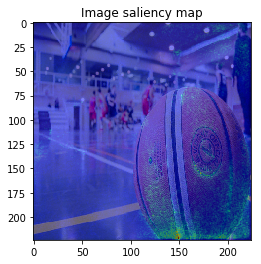
for index in range(len(saliency_vanilla)):
image = imgs[index].resize(saliency_smooth[index].shape)
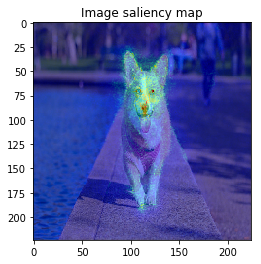
visualizer.visualize(saliency_smooth[index], image, show_colorbar=False)
aggregate, visualize 部分はVanillaGradの時と全く同じです。
以上のように、物体のEdgeの部分などに着目しており、VanillaGradと比べてかなりいい見た目の結果が得られました!
BayesGrad
最後に、VGG16はdropout を含むCNNとなっているためBayesGradの適用も可能です。BayesGradを適用してみた結果を載せてみます。
Calculatorのcompute method に train=True を指定することで自動的にdropout を利用したモデルの予測分布を考慮した saliency_samples を計算することができます。
M = 30
# --- BayesGrad ---
# 2. compute
saliency_samples_bayes = gradient_calculator.compute(x, M=M, train=True)
こちらはVanillaGradとあまり結果が変わりませんでした。
ちなみに、SmoothGradとBayesGrad両方を合わせることもでき、その場合は以下のようになります。
分子データで回帰モデルの予測寄与度を可視化
回帰タスクでは、Saliency を計算することで予測が正負どちらに寄与したのかを計算・可視化することもできます。
ここでは、Chainer ChemistryのGraph convolution モデルを用いて分子データに対する水溶性の可視化を行ってみます。
データセットはESOL datasetを使用します。
import numpy as np
import chainer
from chainer.functions import relu, dropout
from chainer_chemistry.models.ggnn import GGNN
from chainer_chemistry.datasets.numpy_tuple_dataset import NumpyTupleDataset
from chainer_chemistry.datasets.zinc import get_zinc250k
from chainer_chemistry.dataset.preprocessors.ggnn_preprocessor import GGNNPreprocessor
from chainer_chemistry.models.mlp import MLP
from chainer_chemistry.models.prediction.regressor import Regressor
# Model
def activation_relu_dropout(h):
return dropout(relu(h), ratio=0.25)
class GraphConvPredictor(chainer.Chain):
def __init__(self, graph_conv, mlp=None):
"""Initializes the graph convolution predictor.
Args:
graph_conv: The graph convolution network required to obtain
molecule feature representation.
mlp: Multi layer perceptron; used as the final fully connected
layer. Set it to `None` if no operation is necessary
after the `graph_conv` calculation.
"""
super(GraphConvPredictor, self).__init__()
with self.init_scope():
self.graph_conv = graph_conv
if isinstance(mlp, chainer.Link):
self.mlp = mlp
if not isinstance(mlp, chainer.Link):
self.mlp = mlp
def __call__(self, atoms, adjs):
x = self.graph_conv(atoms, adjs)
if self.mlp:
x = self.mlp(x)
return x
n_unit = 32
conv_layers = 4
class_num = 1
device = 0 # -1 for CPU
ggnn = GGNN(out_dim=n_unit, hidden_dim=n_unit, n_layers=conv_layers)
mlp = MLP(out_dim=class_num, hidden_dim=n_unit, activation=activation_relu_dropout)
predictor = GraphConvPredictor(ggnn, mlp)
regressor = Regressor(predictor, device=device)
# Dataset
preprocessor = GGNNPreprocessor()
result = get_molnet_dataset('delaney', preprocessor, labels=None, return_smiles=True)
train = result['dataset'][0]
smiles = result['smiles'][0]
モデルの訓練後、(コードは省略)可視化を行います。
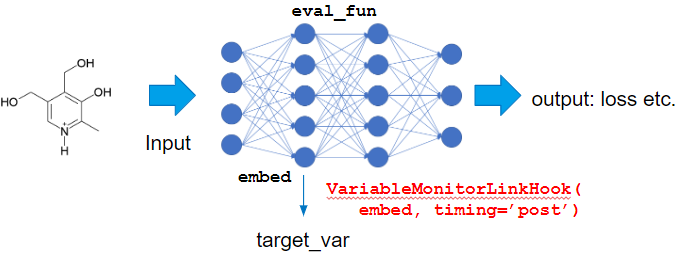
今回はロスに対する寄与度ではなく、予測値そのものを output_var として扱いたいので以下のように predictor のoutputを見るように eval_fun を定義します。
また、今回の入力である x は原子番号のlabel となっており、それ自身にはgradient は計算されないため、gradient は embed layer がかけられた後の隠れ層の値に対するものを採用することにします。
このように、中間層のVariableを target_var としたい場合には VariableMonitorLinkHook を設定することで対応することができます。
以下では IntegratedGradientsCalculator を用いてsaliency の計算を行っています。
import chainer.functions as F
from chainer_chemistry.saliency.calculator.gradient_calculator import GradientCalculator
from chainer_chemistry.saliency.calculator.integrated_gradients_calculator import IntegratedGradientsCalculator
from chainer_chemistry.link_hooks.variable_monitor_link_hook import VariableMonitorLinkHook
def eval_fun(x, adj, t):
pred = predictor(x, adj)
pred_summed = F.sum(pred)
return pred_summed
# 1. instantiation
calculator = IntegratedGradientsCalculator(
predictor, steps=5, eval_fun=eval_fun, target_extractor=VariableMonitorLinkHook(ggnn.embed, timing='post'),
device=device)
実際に可視化を行うと以下のような結果が得られました。
from chainer_chemistry.saliency.visualizer.mol_visualizer import SmilesVisualizer
from chainer_chemistry.saliency.visualizer.common import abs_max_scaler
visualizer = SmilesVisualizer()
# 2. compute
saliency_samples_vanilla = calculator.compute(
train, M=1, converter=concat_mols)
method = 'raw'
saliency_vanilla = calculator.aggregate(
saliency_samples_vanilla, ch_axis=3, method=method)
i = 153
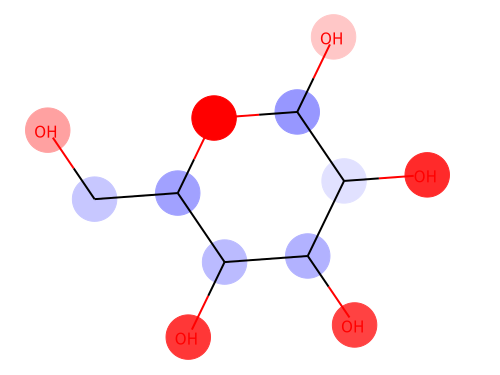
visualizer.visualize(saliency_vanilla[i], smiles[i])
赤色が水に溶けやすくなる方向に寄与した部分(親水基)、青色が水に溶けにくくなる方向に寄与した部分(疎水基)を表しています。
OHなどの分極が生じる場所で親水性が上がっており、Cの鎖が続いているところでは親水性が下がるという一般傾向がきちんと可視化できました。
おわりに
いろいろなドメインで使えるようにライブラリ化したsaliency module を紹介しました。
画像のsaliency map可視化でも、pre-trained model を使って学習は省略すればお手元のCPUのみのPCでも
十分試せるので、是非試してみてください!