Juliaの分散並列パッケージDistributedArraysが遅いの続編です。
DistributedArraysを使った計算が遅い、という話ですが、一つ問題が解決したおかげで前に一歩進めました。しかし、まだ問題が残っています。2並列にすると1並列時の200倍くらい遅くなってしまいます。
環境
Julia Version 1.6.0
Commit f9720dc2eb (2021-03-24 12:55 UTC)
Platform Info:
OS: macOS (x86_64-apple-darwin19.6.0)
CPU: Intel(R) Core(TM) i7-8700B CPU @ 3.20GHz
WORD_SIZE: 64
LIBM: libopenlibm
LLVM: libLLVM-11.0.1 (ORCJIT, skylake)
コード
以下のコードを見てください。
using Distributed
using BenchmarkTools
@everywhere using DistributedArrays
@everywhere function loop_tr(A,NC,NV)
i_local = localindices(A)
s = 0
for i = 1:(i_local[3][end]-i_local[3][1]+1)
for k=1:NC
s += A.localpart[k,k,i]
end
end
return s
end
function partial_tr(A::DArray,NC,NV)
s = 0
for id in workers()
s += remotecall_fetch(loop_tr,id,A,NC,NV)
end
return s
end
function partial_tr(A::Array{T,3},NC,NV) where {T <: Number}
s = 0
for i=1:NV
for k=1:NC
s += A[k,k,i]
end
end
return s
end
function test()
NC = 3
NV = 256
A_normal = rand(ComplexF64,NC,NC,NV)
num_workers = nworkers()
println("workers: ", num_workers)
A_dist = distribute(A_normal,procs= workers(),dist= [1,1,num_workers])
@btime tr_normal = partial_tr($A_normal,$NC,$NV)
tr_normal = partial_tr(A_normal,NC,NV)
println("normal: $tr_normal")
@btime tr_dist = partial_tr($A_dist,$NC,$NV)
tr_dist = partial_tr(A_dist,NC,NV)
println("DArray: $tr_dist")
end
test()
このコードを実行しますと、
>julia paratr.jl
workers: 1
1.127 μs (0 allocations: 0 bytes)
normal: 383.7897468470404 + 387.62678654165785im
3.780 μs (11 allocations: 384 bytes)
DArray: 383.7897468470404 + 387.62678654165785im
となります。(3,3,256)のサイズの3次元配列を作り、これを(3,3)の行列が256個並んでいるとみなして行列のトレースをとりその和を計算しています。DistributedArraysの方が3倍遅いですが、まあremotecallを使っているから何か余計な計算をしている、と考えればまあいいでしょう。
問題は次の場合です。元々DistributedArraysを使いたいのは並列計算がしたいからですから、並列にトレースの計算がしたいです。ですので、
julia -p 2 paratr.jl
workers: 2
1.116 μs (0 allocations: 0 bytes)
normal: 376.1643269152916 + 386.4124089637121im
214.621 μs (136 allocations: 4.73 KiB)
DArray: 376.16432691529167 + 386.4124089637116im
をしてみました。この場合、workerの数は2なので2個の並列になっているはずで、計算結果は同じですからちゃんと計算できているはずですが、計算時間が200倍遅いです。
トレースの計算では、256個の3x3行列のトレースの和をとる際に、256個を128と128の二つのプロセスにわけ、それぞれのトレースの和をとって、最後に二つを足しています。値が同じになっていますから計算自体は動いているはずですが、めちゃめちゃ遅いです。remotecall_fetchではなく、remotecallやspawnatもやってみましたが、遅さは変わりませんでした。
function partial_tr(A::DArray,NC,NV)
s = 0
f = []
for id in workers()
fi = @spawnat id loop_tr(A,NC,NV)
#fi = remotecall(loop_tr,id,A,NC,NV)
push!(f,fi)
end
for fi in f
wait(fi)
s += fetch(fi)
end
return s
end
原因不明です。
解決策を募集します。この問題を解決できると、格子量子色力学シミュレーションパッケージLatticeQCD.jlが並列化できて大変助かるのですが、今のところ解決できていません。LatticeQCD.jlでは、トレースの他にも、3x3行列だと思って二つの積を計算したりもしますが、こちらも遅いです。
追記
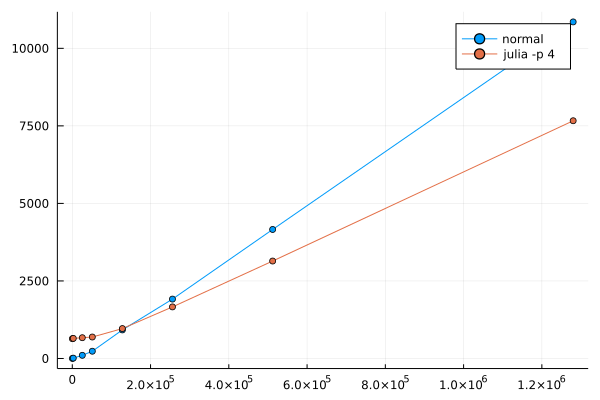
どのくらい遅くなっているかを確認するために、グラフを描いてみました。2並列が青、4並列が赤、普通のArrayが緑です。縦軸は時間、横軸は配列の3次元目のサイズです。
みてわかるのは、何かオーバーヘッドがあり、それは並列数分だけかかっている、ということですね。オーバーヘッドがあっても良いのですが、並列数分かかってしまうと遅すぎます。これはremotecall_fetchの影響なのか、何なのでしょう。また、傾きを見ると赤と青は似た感じですが、緑だけ傾きが小さいです。
この結果だと、並列化すればするほど計算時間がかかってしまいます。書き方が悪いのでしょうか?
追記2
@antimon2 さんのコメントにあるように
function partial_tr(A::DArray{T},NC,NV) where {T}
s = zero(T)
fs = [remotecall(loop_tr, id, A, NC, NV) for id in workers()]
for f in fs
s += fetch(f)
end
return s
end
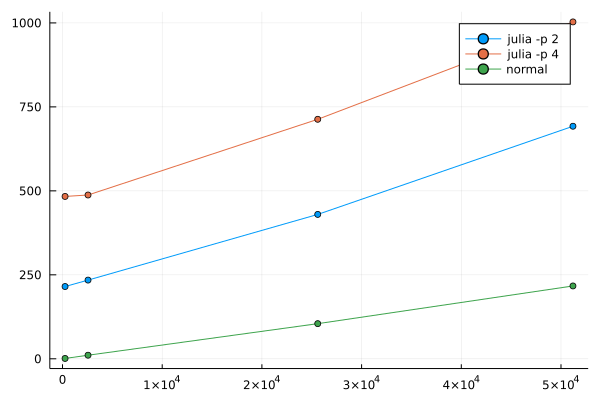
とすることで並列に動作するようになりました。
remotecall_fetchをすると並列化の恩恵が全く受けられていなかったようです。4並列でやってみた結果のグラフを載せます。縦軸は時間、横軸は配列の3次元目のサイズです。
4並列の方が速くなっていることから、何らかの並列化は効いているようです。ただし、オーバーヘッドの数百マイクロ秒を引いたとしても1/4にはなっていません。扱う問題がDistributedArraysに向いていなかったのかもしれません。MPI.jlの使用も考えたいと思います。