はじめに
PythonでCHAIDアルゴリズムを使いたいとき、一応"パッケージ"はあるが、可視化機能がイケていない。それが理由でほぼ使っていなかった。
ただ以前書いた記事「ete3を使って決定木可視化(SPSS Modelerにできるだけ寄せる)」でCARTの可視化を良い感じにできたので、それを応用してCHAIDの可視化もできそうだなと、時間があったらトライしようと思っていた。
今回トライして成功したのでCARTの可視化と合わせて記録に残す。
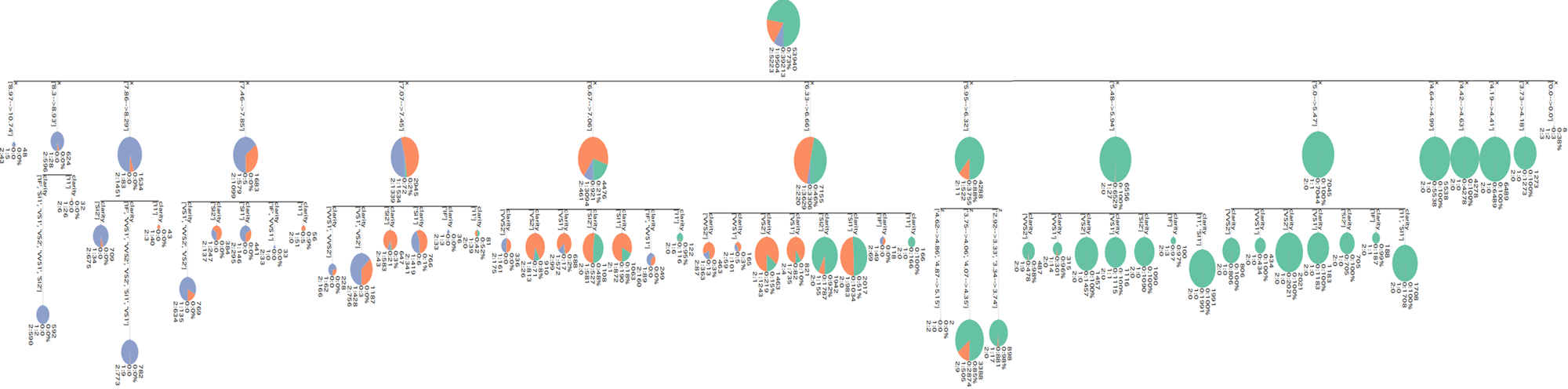
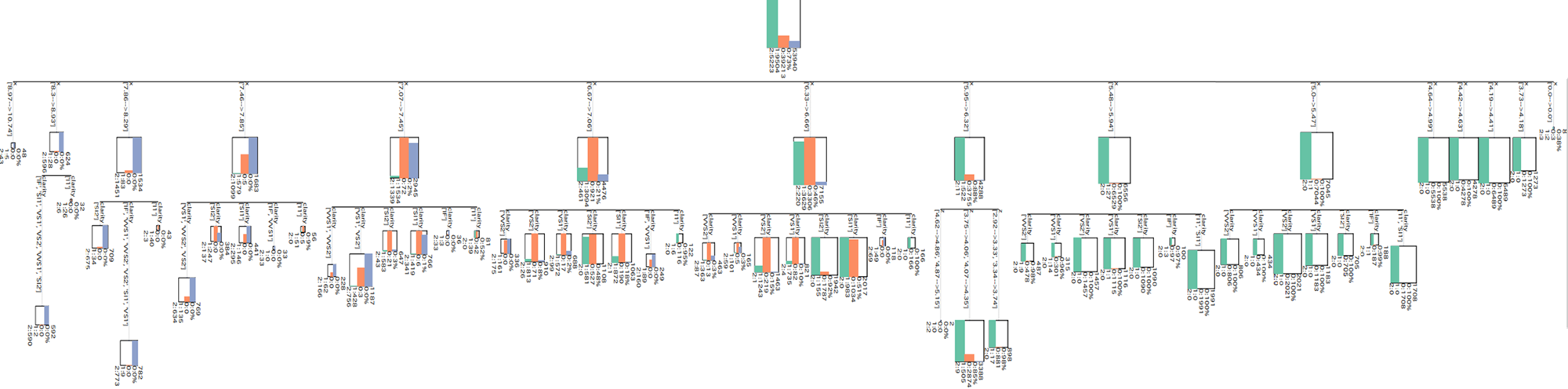
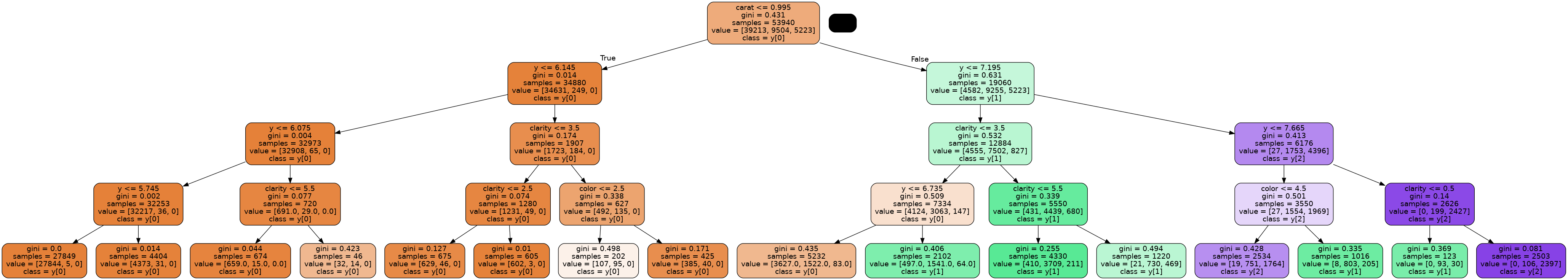
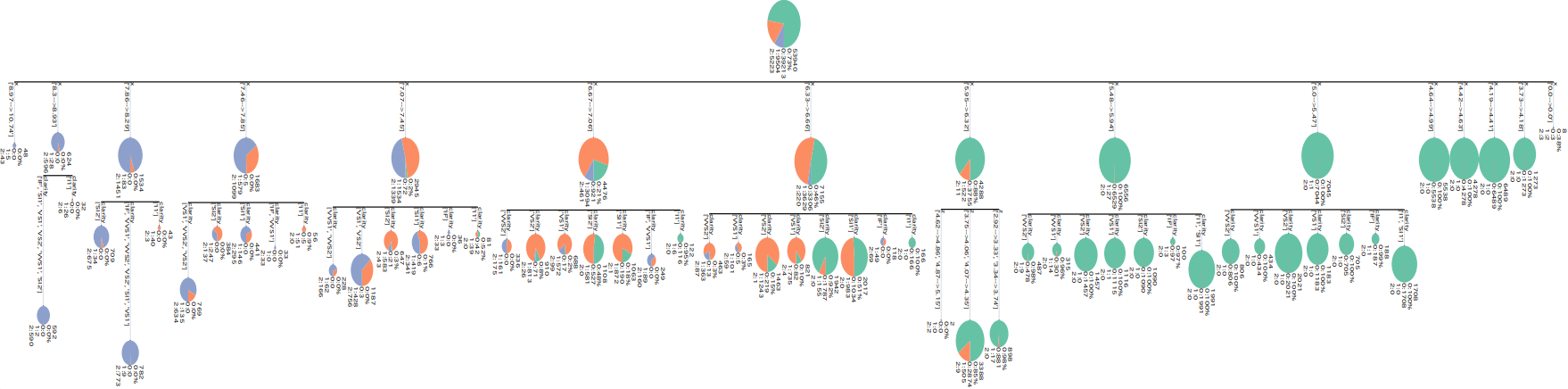
ちなみにCHAIDの結果は以下のような感じ。
※ただし分類木にしか対応していない
参考
- [Github]CHAID
- ete3を使って決定木可視化(SPSS Modelerにできるだけ寄せる)
- Scikit-learnで学習した決定木をETEを使って可視化するモジュール(eteview)を構築してみた
環境
-
DockerのPython環境(Debian)
-
Python 3.10.11
-
Jupyter Lab
-
CHAIDの構築、可視化に必要なパッケージと使用したバージョン
- ete3 --'3.1.3'
- CHAID --'5.4.1'
- PyQt5 --'5.15.10'
ete3の内部で使う - sklearn2pmml --'0.105.2'
CHAIDのwrapper。sklearnライクに使える - svglib --'1.5.1'
svgファイルをpdfファイルやpngファイルに変換するために使う - reportlab --'4.2.0'
svgファイルをpdfファイルやpngファイルに変換するために使う - rlPyCairo --'0.3.0'
svgファイルをpdfファイルやpngファイルに変換するために使う - pykakasi --'2.2.1'
必須ではない。現状日本語のカラムやカテゴリーを可視化できないのでこれでローマ字に変えたりすると良いかも。今回は使っていない。
追記
ete3で可視化時に、例えば下記のように「floatやめろ。intにしろ。」って怒られた場合、
<略>
752 ii= QImage(w, h, QImage.Format_ARGB32)
753 ii.fill(QColor(Qt.white).rgb())
--> 754 ii.setDotsPerMeterX(dpi / 0.0254) # Convert inches to meters
755 ii.setDotsPerMeterY(dpi / 0.0254)
756 pp = QPainter(ii)
TypeError: setDotsPerMeterX(self, int): argument 1 has unexpected type 'float'
(関連:issues/635)
(関連:pull/617)
エラー解消のために以下のリンクのように修正する。
と言っても、手動でスクリプトを修正するのも面倒なので「[Github]ete/ete3/treeview/」に修正されたface.pyとmain.pyとqt.pyがあり、自分はこれをダウンロードして上書きして対処した。
# ダウンロードした後、上書きするコマンドを実行
mv <your path>/faces.py /usr/local/lib/python3.10/site-packages/ete3/treeview --force
mv <your path>/qt.py /usr/local/lib/python3.10/site-packages/ete3/treeview --force
mv <your path>/main.py /usr/local/lib/python3.10/site-packages/ete3/treeview --force
パッケージインポート
import numpy as np
import pandas as pd
import seaborn as sns
import matplotlib as mpl
import matplotlib.pyplot as plt
import matplotlib.font_manager as fm
import japanize_matplotlib
import os
import sys
import ast
import json
import glob
import tqdm as tq
from tqdm import tqdm
import scipy
import gc
import pickle
import datetime as dt
import collections
import time
from dateutil.relativedelta import relativedelta
import sklearn
from sklearn.tree import DecisionTreeRegressor, DecisionTreeClassifier
from sklearn.preprocessing import StandardScaler, LabelEncoder
from sklearn.preprocessing import KBinsDiscretizer
from sklearn.datasets import load_breast_cancer
import string
import re
from collections import OrderedDict
from collections import Counter
import itertools
os.environ['QT_QPA_PLATFORM']='offscreen' # ete3をJupyterで使うときこれをしないとカーネルが落ちる
sns.set()
# 可視化用パッケージ
import ete3
from ete3 import Tree, TreeStyle, TextFace, PieChartFace, BarChartFace
import PyQt5
import sklearn2pmml
from sklearn2pmml.tree.chaid import CHAIDClassifier
import CHAID
from CHAID import Tree as chaidTree
import svglib
from svglib.svglib import svg2rlg
import reportlab
from reportlab.graphics import renderPDF, renderPM
import rlPyCairo
import pykakasi # 日本語の可視化ができないのでこれでローマ字に変えたりしましょう
from six import StringIO # sklearnのCART可視化用
import pydotplus # sklearnのCART可視化用
from IPython.display import Image # sklearnのCART可視化用
import dtreeviz # sklearnのCART可視化用
# 日本語フォント読み込み
jpn_font = japanize_matplotlib.get_font_ttf_path()
prop = fm.FontProperties(fname=jpn_font)
print(jpn_font)
# plt.rcParams['font.family'] = prop.get_name() #全体のフォントを設定
使用データ
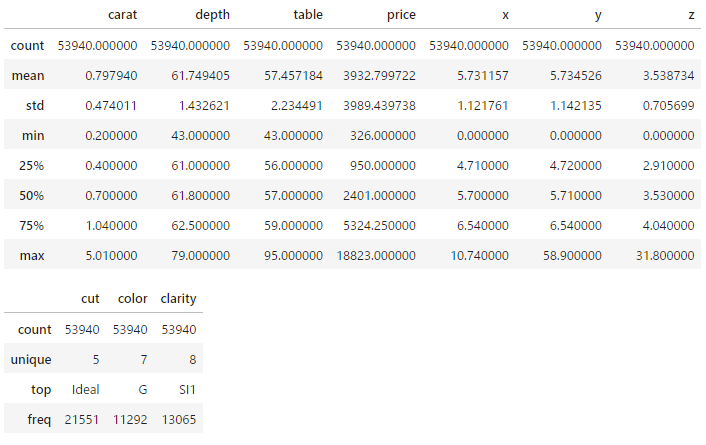
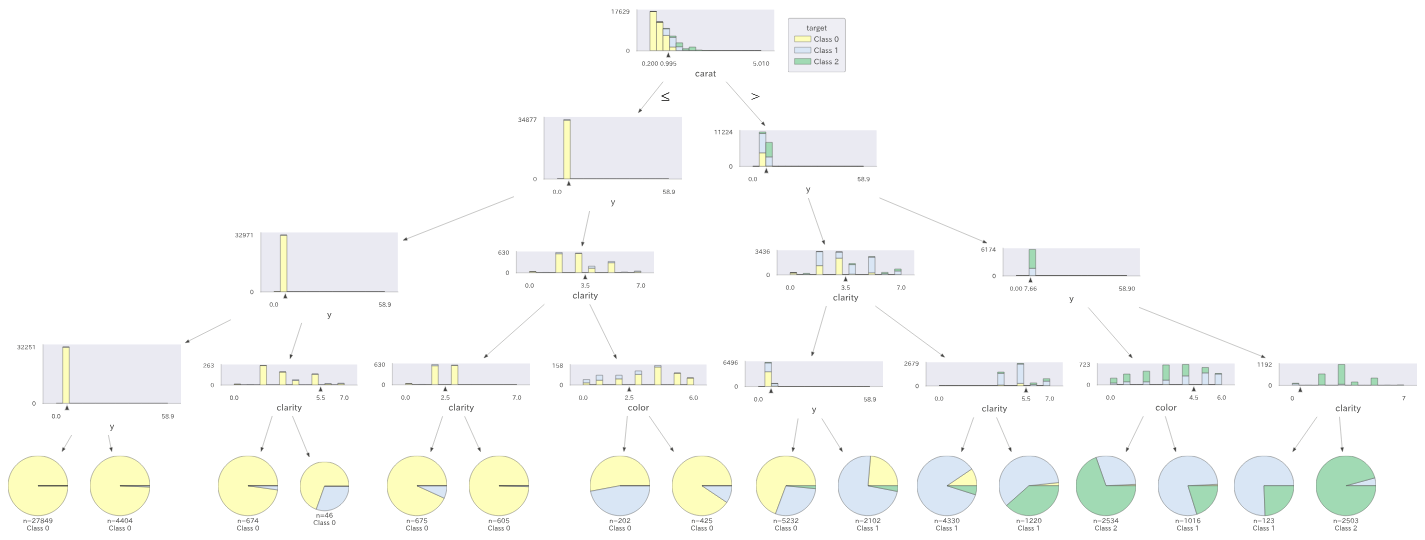
seabornにあるデータセット。ダイヤモンドの価格データで5万レコードくらいある。
- price(価格):ダイヤモンドの価格(USドル)(326~18,823ドル)
- carat(カラット):ダイヤモンドの重量(0.2~5.01カラット)
- cut(カット):カットの品質(Fair, Good, Very Good, Premium, Ideal)
- color(カラー):ダイヤモンドの色、J(最低)からD(最高)まで
- clarity(クラリティ):ダイヤモンドの透明度の尺度(I1(最低)、SI2、SI1、VS2、VS1、VVS2、VVS1、IF(最高))
- x:長さ(mm)(0~10.74)
- y:幅(mm)(0~58.9)
- z:深さ(mm)(0~31.8)
- depth(深さ):全体の深さの割合 = z / mean(x, y) = 2 * z / (x + y)(43~79)
- table(テーブル):ダイヤモンドの最も広い部分に対するテーブル(天面)の幅の割合(43~95)
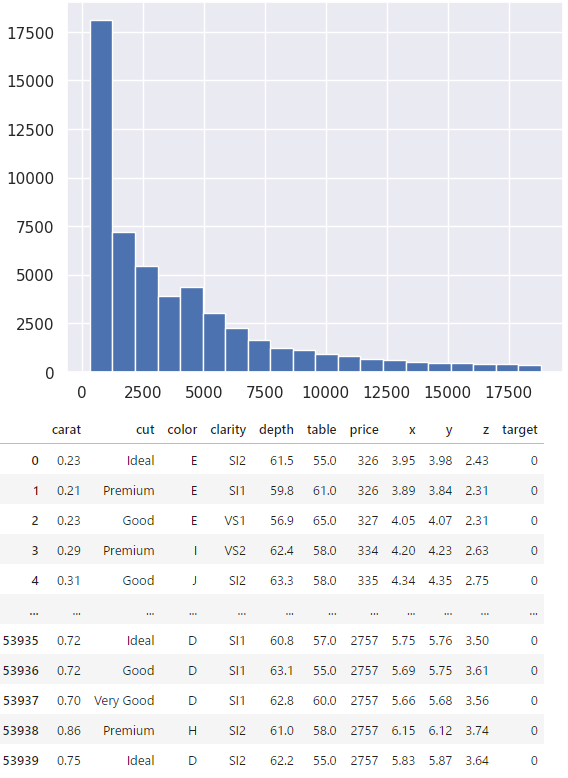
分類問題にしたいのでpriceを3段階に分ける処理をしておく。
# Load diamonds dataset from seaborn
df = sns.load_dataset('diamonds')
display(df.describe())
display(df.describe(include='category'))
plt.hist(df['price'], bins=20)
plt.show()
# priceを質的変数へ
df.loc[(df['price']<5000), 'target'] = 0 #'lower_price'
df.loc[(df['price']>=5000)&(df['price']<10000), 'target'] = 1 #'middle_price'
df.loc[(df['price']>=10000), 'target'] = 2 #'upper_price'
df['target'] = df['target'].astype(int)
display(df)
前処理として質的変数をラベルエンコーディングしておく。
エンコーディング前後のラベルのマスタは辞書として作っておく。
# LabelEncoding
category_cols = df.select_dtypes('category').columns.to_list()
le_list = {} # ラベルマスタ辞書
dfle = df.copy()
for c in category_cols:
le = sklearn.preprocessing.LabelEncoder()
dfle[c] = le.fit_transform(df[c])
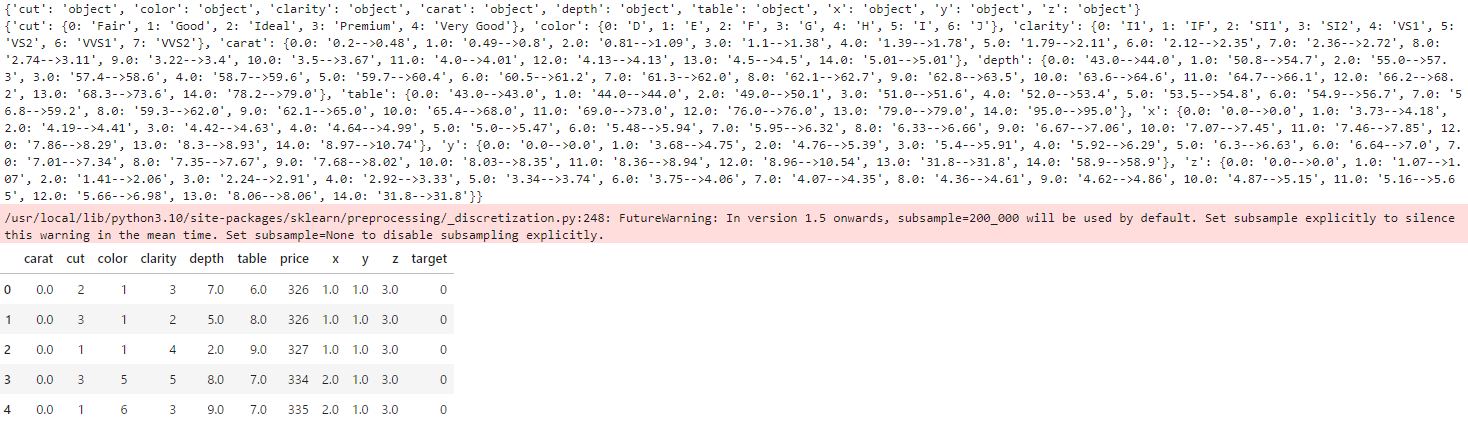
label_tmp = {k:v for k, v in zip(le.transform(le.classes_), le.classes_)}
le_list[c] = label_tmp
print(le_list) # ラベルマスタ辞書{<カラム名>:{<ラベル>:<元のカテゴリ>}}
# >> {'cut': {0: 'Fair', 1: 'Good', 2: 'Ideal', 3: 'Premium', 4: 'Very Good'}, 'color': {0: 'D', 1: 'E', 2: 'F', 3: 'G', 4: 'H', 5: 'I', 6: 'J'}, 'clarity': {0: 'I1', 1: 'IF', 2: 'SI1', 3: 'SI2', 4: 'VS1', 5: 'VS2', 6: 'VVS1', 7: 'VVS2'}}
説明変数カラムと目的変数カラムの定義。
# 説明変数と目的変数
colx = ['cut', 'color', 'clarity', 'carat', 'depth', 'table', 'x', 'y', 'z']
coly = 'target'
CART
まずはCARTアルゴリズムによる決定木の可視化をする。
説明変数がすべて連続値の場合
何も気にせずそのまま突っ込み学習。
# 学習
df_add = dfle.copy()
clf = DecisionTreeClassifier(min_samples_split=100, max_depth=4, random_state=0)
clf.fit(df_add[colx].to_numpy(), df_add[coly].to_numpy())
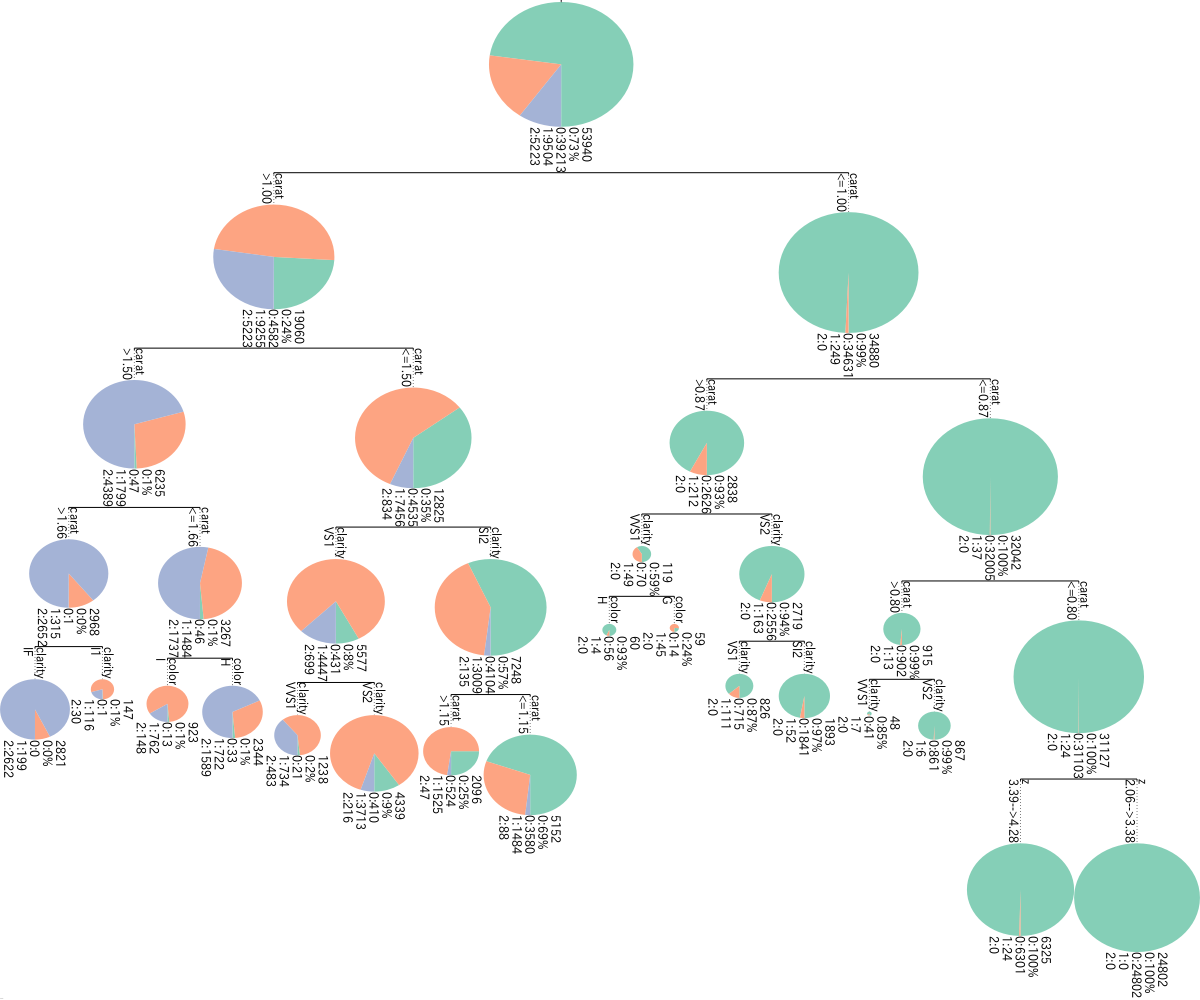
とりあえずsklearn標準の可視化。見づらい。
# 決定木の可視化 sklearn標準
dot_data = StringIO()
sklearn.tree.export_graphviz(clf, out_file=dot_data
, feature_names=colx
, class_names=True
, filled=True, rounded=True)
graph = pydotplus.graph_from_dot_data(dot_data.getvalue())
Image(graph.create_png())
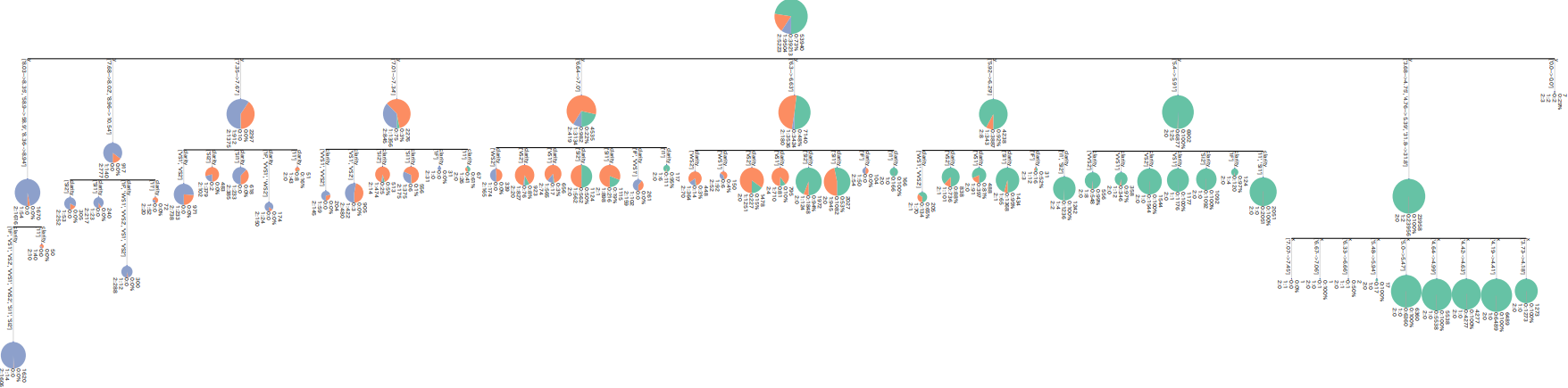
dtreevizでも可視化。悪くはないけどすべて円グラフの方が嬉しい…。
# dtreevizでも可視化
viz = dtreeviz.model(clf#,tree_index=1
, X_train=df_add[colx],y_train=df_add[coly]
, feature_names=colx
, target_name=coly)
viz.view(fontname=prop.get_name())
ETE Toolkitによる可視化関数定義。
(詳細は「ete3を使って決定木可視化(SPSS Modelerにできるだけ寄せる)」)
※目的変数は数字にしか対応していない(文字列不可)
# ete3によるCARTアルゴリズムの可視化関数
def makeTreeEte3CART(clffit, tree_cols, obj_unique, le_list_rename=None, dtype_arr=None, fsize=100, height=300, width=300, diameter=100, rotation=0, node_face_type='pie', name='tmp'):
# eteのTreeインスタンスを構築
tree = Tree()
# node size
# 各ノードのサンプル数の順に木のノードの大きさを変える
# tree_.n_node_samplesはノードごとのサンプル数を返す
nodesize={}
for i, n in enumerate(np.sort(clffit.tree_.n_node_samples)):
nodesize[n] = i+1
# 各ノードを設定していく
# tree_.node_countはノード数を返す
for i in range(clffit.tree_.node_count):
#i=0、つまりルートノードの名称を0にする
if i == 0:
tree.name = str(0)
# 設定するノードを指定
# name=str(i)であるete3親ノードの設定をする
node = tree.search_nodes(name=str(i))[0]
# ノードごとに目的変数別の%を計算し、配分の円グラフを作成
if node_face_type == 'pie':
Graph_Object = PieChartFace(percents=clffit.tree_.value[i][0] / clffit.tree_.value[i].sum() * 100 # 目的変数別の割合
, width=nodesize[clffit.tree_.n_node_samples[i]]*diameter # nodesize辞書内の数値×diameter
, height=nodesize[clffit.tree_.n_node_samples[i]]*diameter # nodesize辞書内の数値×diameter
, colors=ete3.COLOR_SCHEMES['set2']) # グラフの色
# ノードごとに目的変数別の数を計算し、barグラフを作成
elif node_face_type == 'bar':
Graph_Object = BarChartFace(values=clffit.tree_.value[i][0] # 目的変数別のサンプル数
, width=nodesize[clffit.tree_.n_node_samples[i]]*diameter # nodesize辞書内の数値×diameter
, height=nodesize[clffit.tree_.n_node_samples[i]]*diameter # nodesize辞書内の数値×diameter
, colors=ete3.COLOR_SCHEMES['set2']) # グラフの色
else:
print('Node face type is selected as `PieChartFace`')
Graph_Object = PieChartFace(percents=clffit.tree_.value[i][0] / clffit.tree_.value[i].sum() * 100 # 目的変数別の割合
, width=nodesize[clffit.tree_.n_node_samples[i]]*diameter # nodesize辞書内の数値×diameter
, height=nodesize[clffit.tree_.n_node_samples[i]]*diameter # nodesize辞書内の数値×diameter
, colors=ete3.COLOR_SCHEMES['set2']) # グラフの色
Graph_Object.rotation = 360 - rotation # rotation度回転
Graph_Object.opacity = 0.8
#Graph_Object.hz_align = 2 # 0 left, 1 center, 2 right
#Graph_Object.vt_align = 2 # 0 left, 1 center, 2 right
#グラフをセット
# position='aligned', 'branch-top', 'float-behind', 'branch-bottom', 'float', 'branch-right'
node.add_face(Graph_Object, column=2, position="branch-right")
# 左下の子ノードの設定をする
if clffit.tree_.children_left[i] > -1: # 左下に子ノードがある場合(-1の時、子ノードはない)
# ノード名称はsklearnのtreeのリストIDと一致させる
node.add_child(name=str(clffit.tree_.children_left[i])) # ete3子ノード追加
# 対象を子ノードに移す
node = tree.search_nodes(name=str(clffit.tree_.children_left[i]))[0]
# 分岐条件を追加
# position='aligned', 'branch-top', 'float-behind', 'branch-bottom', 'float', 'branch-right'
if le_list_rename and dtype_arr:
if dtype_arr[tree_cols[clffit.tree_.feature[i]]]=='object':
object_col = tree_cols[clffit.tree_.feature[i]]
node.add_face(TextFace(object_col, fsize=fsize) # 特徴量の名前
, column=0, position="branch-top") # Text位置
category_number = int(np.floor(clffit.tree_.threshold[i]))
node.add_face(TextFace(le_list_rename[object_col][category_number], fsize=fsize) # 特徴量の分岐の閾値
, column=1, position="branch-bottom") # Text位置
else:
node.add_face(TextFace(tree_cols[clffit.tree_.feature[i]], fsize=fsize) # 特徴量の名前
, column=0, position="branch-top") # Text位置
node.add_face(TextFace(u"<=" + "{0:.2f}".format(clffit.tree_.threshold[i]), fsize=fsize) # 特徴量の分岐の閾値
, column=1, position="branch-bottom") # Text位置
else:
node.add_face(TextFace(tree_cols[clffit.tree_.feature[i]], fsize=fsize) # 特徴量の名前
, column=0, position="branch-top") # Text位置
node.add_face(TextFace(u"<=" + "{0:.2f}".format(clffit.tree_.threshold[i]), fsize=fsize) # 特徴量の分岐の閾値
, column=1, position="branch-bottom") # Text位置
# 親ノードに対象を戻しておく
node = tree.search_nodes(name=str(i))[0]
# 右下の子ノードの設定をする
if clffit.tree_.children_right[i] > -1: # 右下に子ノードがある場合(-1の時、子ノードはない)
# ノード名称はsklearnのtreeのリストIDと一致させる
node.add_child(name=str(clffit.tree_.children_right[i])) # ete3子ノード追加
# 対象を子ノードに移す
node = tree.search_nodes(name=str(clffit.tree_.children_right[i]))[0]
# 分岐条件を追加
# position='aligned', 'branch-top', 'float-behind', 'branch-bottom', 'float', 'branch-right'
if le_list_rename and dtype_arr:
if dtype_arr[tree_cols[clffit.tree_.feature[i]]]=='object':
object_col = tree_cols[clffit.tree_.feature[i]]
node.add_face(TextFace(object_col, fsize=fsize) # 特徴量の名前
, column=0, position="branch-top") # Text位置
category_number = int(np.ceil(clffit.tree_.threshold[i]))
node.add_face(TextFace(le_list_rename[object_col][category_number], fsize=fsize) # 特徴量の分岐の閾値
, column=1, position="branch-bottom") # Text位置
else:
node.add_face(TextFace(tree_cols[clffit.tree_.feature[i]], fsize=fsize) # 特徴量の名前
, column=0, position="branch-top") # Text位置
node.add_face(TextFace(">" + "{0:.2f}".format(clffit.tree_.threshold[i]), fsize=fsize) # 特徴量の分岐の閾値
, column=1, position="branch-bottom") # Text位置
else:
node.add_face(TextFace(tree_cols[clffit.tree_.feature[i]], fsize=fsize) # 特徴量の名前
, column=0, position="branch-top") # Text位置
node.add_face(TextFace(">" + "{0:.2f}".format(clffit.tree_.threshold[i]), fsize=fsize) # 特徴量の分岐の閾値
, column=1, position="branch-bottom") # Text位置
# 親ノードに対象を戻しておく
node = tree.search_nodes(name=str(i))[0]
# ノード内のサンプル数や割合をテキストとして記す
obj_unique = sorted(obj_unique)
text1 = str(obj_unique[0])+":{0:.0f}".format(clffit.tree_.value[i][0][0] / clffit.tree_.value[i].sum() * 100) + "%"
text1_1 = "{0:.0f}".format(clffit.tree_.n_node_samples[i])
texts_ary = []
for obj_i, obj_val in enumerate(obj_unique):# 各クラスのサンプル数
txt = "{:.0f}:{:.0f}".format(obj_i, clffit.tree_.value[i][0][obj_i]*clf.tree_.n_node_samples[i])
texts_ary.append(txt)
# 情報を書き込み
# position='aligned', 'branch-top', 'float-behind', 'branch-bottom', 'float', 'branch-right'
node.add_face(TextFace(text1_1, fsize=fsize, fgcolor="black")
, column=4, position="branch-right")
node.add_face(TextFace(text1, fsize=fsize, fgcolor="black")
, column=4, position="branch-right")
for txt_i in texts_ary:
node.add_face(TextFace(txt_i, fsize=fsize)
, column=4, position="branch-right")
# 図の回転
ts = TreeStyle()
ts.show_leaf_name = True
ts.rotation = rotation
# 図の保存
tree.render(name+'.svg', h=height, w=width, tree_style=ts, dpi=250)
try:
from svglib.svglib import svg2rlg
from reportlab.graphics import renderPDF, renderPM
import rlPyCairo
import sys
filename = name+'.svg'
drawing = svg2rlg(filename)
renderPDF.drawToFile(drawing, filename + ".pdf")
drawing = svg2rlg(filename)
renderPM.drawToFile(drawing, filename + ".png", fmt="PNG", dpi=250)
except ModuleNotFoundError:
pass
return tree
関数実行。
# 説明変数がすべて連続値の場合
obj_unique = np.sort(dfle[coly].unique())
tree = makeTreeEte3CART(clf, colx, obj_unique
, le_list_rename=None
, dtype_arr=None
, fsize=200, height=1000, width=1200, rotation=0
, node_face_type='pie'
, name='sklearn_CART') # sklearn_CART.svgが保存される
# Jupyter上に可視化(Jupyter上だとテキストが表示されない場合がある。保存したファイルにはテキストがあるはず。)
ts = TreeStyle()
ts.show_leaf_name = True
ts.rotation = 0
tree.render("%%inline", tree_style=ts, h=1000, w=1200)
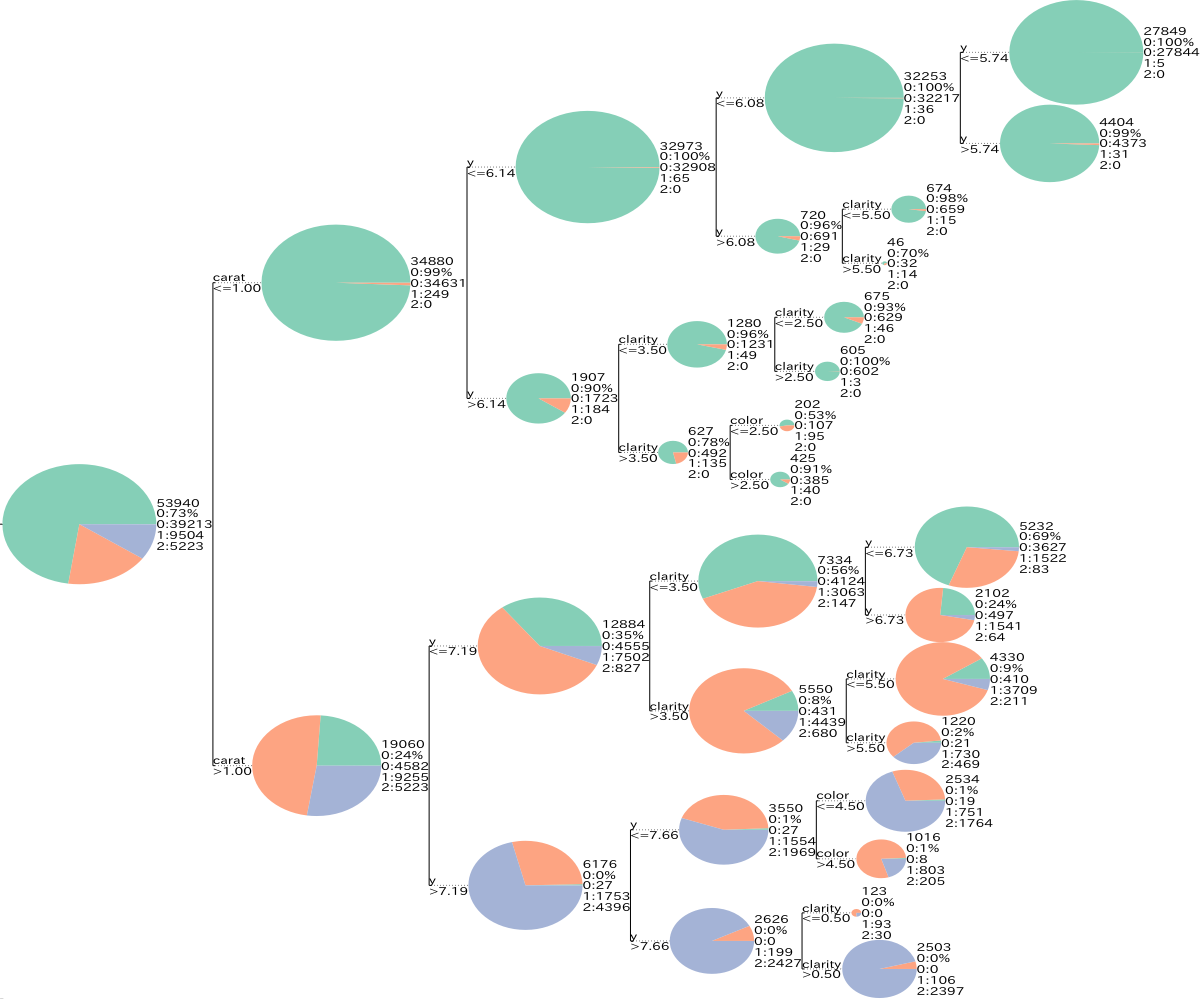
見やすい。各色の面積が各分類の割合を示している。テキストは各ノードのレコード数、フラグ0の割合、各フラグ別のレコード数を示している。
しかしすべて連続値として渡しているので、質的変数も数字の閾値が表示されていてどういうカテゴリーで分かれているかはわかりづらい。
ということで次。
説明変数が一部質的データの場合
質的変数3つに加え、x、y、zのカラムも質的変数に変換する。
6つの質的変数と3つの量的変数のマートを作成する。
# 質的データがある時のCART可視化
# 今回は一部のデータが質的データと仮定
df_add = dfle.copy()
# ビニングにより順序尺度データに変換
bin_dict = {} # 各カラムのビニング後の値とビニング前の数値の幅を辞書化する
dtype_arr = {} # 各カラムのデータ型指定('object'=質的データ。その他=量的データ。)
category_cols = category_cols
bin_cols = colx[6:] # 今回はx,y,zのカラムも質的データと仮定
continuous_cols = colx[3:6] # 残りのカラムが量的データと仮定
for i, col in enumerate(np.concatenate([category_cols,continuous_cols,bin_cols])):
if col in bin_cols:
dtype_arr[col] = 'object'
# 1次元クラスタリングで順序尺度化
est = KBinsDiscretizer(n_bins=5, strategy="kmeans", encode='ordinal')
bins_ = est.fit_transform(df_add[col].to_numpy().reshape(-1, 1))
df_add[col] = bins_
binmast = pd.DataFrame({'org':df[col].to_numpy(), 'bins':bins_.reshape(-1)})
binmast_min = binmast.groupby('bins')['org'].min().reset_index()
binmast_max = binmast.groupby('bins')['org'].max().reset_index()
binmast = pd.merge(binmast_min, binmast_max, on=['bins'], how='outer', suffixes=['_min','_max'])
binmast['ranges'] = binmast['org_min'].round(2).astype(str)+'-->'+binmast['org_max'].round(2).astype(str) # ビン化する前の数値の範囲(例:'11.61-->12.7')
bin_dict_val = {row['bins']:row['ranges'] for index, row in binmast.iterrows()}
bin_dict[col] = bin_dict_val
else:
if col in category_cols:
dtype_arr[col] = 'object'
bin_dict[col] = ''
else:
dtype_arr[col] = 'number'
bin_dict[col] = ''
print(dtype_arr)
bin_dict.update(le_list) # ラベルマスタ
print(bin_dict)
display(df_add.head())
bin_dictとdtype_arrを定義している。
bin_dictは質的変数のエンコーディング前後の対応関係のマスタの辞書。連続値のカラムは空白("")が入っている。{<カラム名>:{<ラベル>:<元のカテゴリ名>}}という形の辞書。量的変数を質的変数に変換した場合、<元のカテゴリ名>にはそのラベルの[最小値] --> [最大値] が文字列として入っている。
dtype_arrは各変数の型を記録している辞書。質的変数の場合'object'、連続値の場合numberを入れている(連続値の場合は'object'以外なら何でもいいが)。
質的変数への変換やそのマスタの定義を終えたので学習。
# 学習
clf = DecisionTreeClassifier(min_samples_split=100, max_depth=4, random_state=0)
clf.fit(df_add[colx].to_numpy(), df_add[coly].to_numpy())
ETE Toolkitで可視化。さっきの可視化関数を使うが引数le_list_renameとdtype_arrを設定する。le_list_rename引数にはbin_dict、dtype_arr引数にはdtype_arrを設定。
obj_unique = np.sort(df_add[coly].unique())
tree = makeTreeEte3CART(clf, colx, obj_unique
, le_list_rename=bin_dict
, dtype_arr=dtype_arr
, fsize=200, height=1000, width=1200, rotation=90
, node_face_type='pie'
, name='sklearn_CARTbin') # sklearn_CARTbin.svgが保存される
# Jupyter上に可視化(Jupyter上だとテキストが表示されない場合がある。保存したファイルにはテキストがあるはず。)
ts = TreeStyle()
ts.show_leaf_name = True
ts.rotation = 90
tree.render("%%inline", tree_style=ts, h=1000, w=1200)
第3分岐あたりや右下あたりを見ると、質的変数はどういうカテゴリーで分かれているのかテキストで書かれていて、閾値が数字ではなくなっている。これで分岐の条件が見やすくなった。
CHAID
CHAIDアルゴリズムによる決定木の可視化をする。
CHAIDは説明変数がすべて質的変数の必要がある。そのため連続値のカラムはすべてビニングして質的変数に変換する。
CARTの時と同じ方法。bin_dictとdtype_arrも定義。
# CHAIDは説明変数が質的データのみ適用可
# すべてのデータを質的データ化する
df_add = dfle.copy()
# ビニングにより順序尺度データに変換
bin_dict = {} # 各カラムのビニング後の値とビニング前の数値の幅を辞書化する
dtype_arr = {} # 各カラムのデータ型指定('object'=質的データ。その他=連続値。)
category_cols = category_cols # 質的データ
continuous_cols = colx[3:] # 量的データから質的データへ
for i, col in enumerate(np.concatenate([category_cols,continuous_cols])):
if col in continuous_cols:
dtype_arr[col] = 'object'
# 1次元クラスタリングで順序尺度化
est = KBinsDiscretizer(n_bins=15, strategy="kmeans", encode='ordinal')
bins_ = est.fit_transform(df_add[col].to_numpy().reshape(-1, 1))
df_add[col] = bins_
binmast = pd.DataFrame({'org':df[col].to_numpy(), 'bins':bins_.reshape(-1)})
binmast_min = binmast.groupby('bins')['org'].min().reset_index()
binmast_max = binmast.groupby('bins')['org'].max().reset_index()
binmast = pd.merge(binmast_min, binmast_max, on=['bins'], how='outer', suffixes=['_min','_max'])
binmast['ranges'] = binmast['org_min'].round(2).astype(str)+'-->'+binmast['org_max'].round(2).astype(str) # ビン化する前の数値の範囲(例:'11.61-->12.7')
bin_dict_val = {row['bins']:row['ranges'] for index, row in binmast.iterrows()}
bin_dict[col] = bin_dict_val
else:
dtype_arr[col] = 'object'
bin_dict[col] = ''
print(dtype_arr)
bin_dict.update(le_list) # ラベルマスタ
print(bin_dict)
display(df_add.head())
可視化関数を定義。
# CHAID可視化関数
def makeTreeEte3CHAID(tree_model, obj_unique, le_list_rename=None, fsize=100, height=300, width=300, diameter=100, rotation=90, node_face_type='pie', name='tmp'):
# CHAIDパッケージは必須
try:
import sklearn2pmml
import CHAID
except ModuleNotFoundError:
import CHAID
# CHAIDパッケージのオブジェクト
if type(tree_model)==CHAID.Tree:
print('CHAID Package Model', tree_model) # おまじない。tree_modelをprintすればOK。しないとtree_model.node_countが0になる。
tree_n_node_samples = [sum([v for v in tree_model.get_node(i).members.values()]) for i in range(tree_model.node_count)]
tree_node_count = tree_model.node_count
tree_value = [np.array([v for v in tree_model.get_node(i).members.values()]) for i in range(tree_model.node_count)]
tree_value_sum = [sum([v for v in tree_model.get_node(i).members.values()]) for i in range(tree_model.node_count)]
tree_node_id = [tree_model.get_node(i).node_id for i in range(tree_model.node_count)]
tree_column = [tree_model.get_node(i).split.column for i in range(tree_model.node_count)]
tree_groupings = [ast.literal_eval(tree_model.get_node(i).split.groupings) for i in range(tree_model.node_count)]
tree_children = [tree_model.to_tree().children(i) for i in range(tree_model.node_count)]
# CHAIDClassifierオブジェクト
elif type(tree_model)==sklearn2pmml.tree.chaid.CHAIDClassifier:
print('CHAIDClassifier Model for sklearn2pmml', tree_model)
tree_n_node_samples = [sum([v for v in model.tree_.get_node(i).members.values()]) for i in range(model.tree_.node_count)]
tree_node_count = model.tree_.node_count
tree_value = [np.array([v for v in model.tree_.get_node(i).members.values()]) for i in range(model.tree_.node_count)]
tree_value_sum = [sum([v for v in model.tree_.get_node(i).members.values()]) for i in range(model.tree_.node_count)]
tree_node_id = [model.tree_.get_node(i).node_id for i in range(model.tree_.node_count)]
tree_column = [model.tree_.get_node(i).split.column for i in range(model.tree_.node_count)]
tree_groupings = [ast.literal_eval(model.tree_.get_node(i).split.groupings) for i in range(model.tree_.node_count)]
tree_children = [model.tree_.to_tree().children(i) for i in range(model.tree_.node_count)]
else:
raise AttributeError('tree_model is incompatible')
# eteのTreeインスタンスを構築
tree = Tree()
nodesize={}
for i, n in enumerate(np.sort(tree_n_node_samples)):
nodesize[n] = i+1
# 各ノードを設定していく
# tree_.node_countはノード数を返す
for i in tqdm(range(tree_node_count)):
if i == 0:
tree.name = str(0)
# 設定するノードを指定
# name=str(i)であるete3親ノードの設定をする
node = tree.search_nodes(name=str(i))[0]
# ノードごとに目的変数別の%を計算し、配分の円グラフを作成
if node_face_type == 'pie':
Graph_Object = PieChartFace(percents=tree_value[i]/tree_value_sum[i] * 100 # 目的変数別の割合
, width=nodesize[tree_n_node_samples[i]]*diameter # nodesize辞書内の数値×100
, height=nodesize[tree_n_node_samples[i]]*diameter # nodesize辞書内の数値×100
, colors=ete3.COLOR_SCHEMES['set2'])# グラフの色
# ノードごとに目的変数別の数を計算し、barグラフを作成
elif node_face_type == 'bar':
Graph_Object = BarChartFace(values=tree_value[i] # 目的変数別のサンプル数
, width=nodesize[tree_n_node_samples[i]]*diameter # nodesize辞書内の数値×100
, height=nodesize[tree_n_node_samples[i]]*diameter # nodesize辞書内の数値×100
, colors=ete3.COLOR_SCHEMES['set2'])# グラフの色
else:
print('Node face type is selected as `PieChartFace`')
Graph_Object = PieChartFace(percents=tree_value[i]/tree_value_sum[i] * 100 # 目的変数別の割合
, width=nodesize[tree_n_node_samples[i]]*diameter # nodesize辞書内の数値×100
, height=nodesize[tree_n_node_samples[i]]*diameter # nodesize辞書内の数値×100
, colors=ete3.COLOR_SCHEMES['set2'])# グラフの色
Graph_Object.rotation = 360 - rotation # rotation度回転
Graph_Object.opacity = 0.8
node.add_face(Graph_Object, column=2, position="branch-right")
if len(tree_children[i]) > 0:
for chi in range(len(tree_children[i])):
# 子ノードid
child_id = tree_children[i][chi].identifier
node.add_child(name=str(child_id)) # ete3子ノード追加
# 対象を子ノードに移す
node = tree.search_nodes(name=str(child_id))[0]
# 分岐条件を追加
# position='aligned', 'branch-top', 'float-behind', 'branch-bottom', 'float', 'branch-right'
node.add_face(TextFace(tree_column[i], fsize=fsize) # 特徴量の名前
, column=0, position="branch-top") # Text位置
if le_list_rename:
replace_val = le_list_rename[tree_column[i]]
node.add_face(TextFace(str([replace_val[st] for st in tree_groupings[i][chi]]), fsize=fsize) # 特徴量の分岐の閾値
, column=1, position="branch-bottom") # Text位置
else:
node.add_face(TextFace(tree_groupings[i][chi], fsize=fsize) # 特徴量の分岐の閾値
, column=1, position="branch-bottom") # Text位置
# 親ノードに対象を戻しておく
node = tree.search_nodes(name=str(i))[0]
# ノード内のサンプル数や割合をテキストとして記す
obj_unique = sorted(obj_unique)
text1 = str(obj_unique[0])+":{0:.0f}".format(tree_value[i][0]/tree_value_sum[i] * 100) + "%"
text1_1 = "{0:.0f}".format(tree_n_node_samples[i])
texts_ary = []
for obj_i, obj_val in enumerate(obj_unique):# 各クラスのサンプル数
txt = "{:.0f}:{:.0f}".format(obj_i, tree_value[i][obj_i])
texts_ary.append(txt)
# 情報を書き込み
# position='aligned', 'branch-top', 'float-behind', 'branch-bottom', 'float', 'branch-right'
node.add_face(TextFace(text1_1, fsize=fsize, fgcolor="black")
, column=4, position="branch-right")
node.add_face(TextFace(text1, fsize=fsize, fgcolor="black")
, column=4, position="branch-right")
for txt_i in texts_ary:
node.add_face(TextFace(txt_i, fsize=fsize)
, column=4, position="branch-right")
# 図の回転
ts = TreeStyle()
ts.show_leaf_name = True
ts.rotation = rotation
# 図の保存
tree.render(name+'.svg', h=height, w=width, tree_style=ts, dpi=250)
try:
from svglib.svglib import svg2rlg
from reportlab.graphics import renderPDF, renderPM
import rlPyCairo
import sys
filename = name+'.svg'
drawing = svg2rlg(filename)
renderPDF.drawToFile(drawing, filename + ".pdf")
drawing = svg2rlg(filename)
renderPM.drawToFile(drawing, filename + ".png", fmt="PNG", dpi=250)
except ModuleNotFoundError:
pass
return tree
sklearnのCARTの場合、sklearnの学習済みオブジェクトからノードIDや分岐条件、子ノードの有無、カラム名などを抽出し、それをグラフ作成に使用していたが、CHAIDパッケージはsklearnに準拠していない。そのためCHAIDパッケージで作成したオブジェクトから、sklearnとは異なるやり方でノードIDや分岐条件、子ノードの有無、カラム名などを抽出する必要がある。
CHAIDパッケージで学習すると、CHAID.tree.Treeオブジェクトが返される。
CHAID.tree.Treeオブジェクトを見てみると、いろいろな情報が入っていることがわかる。
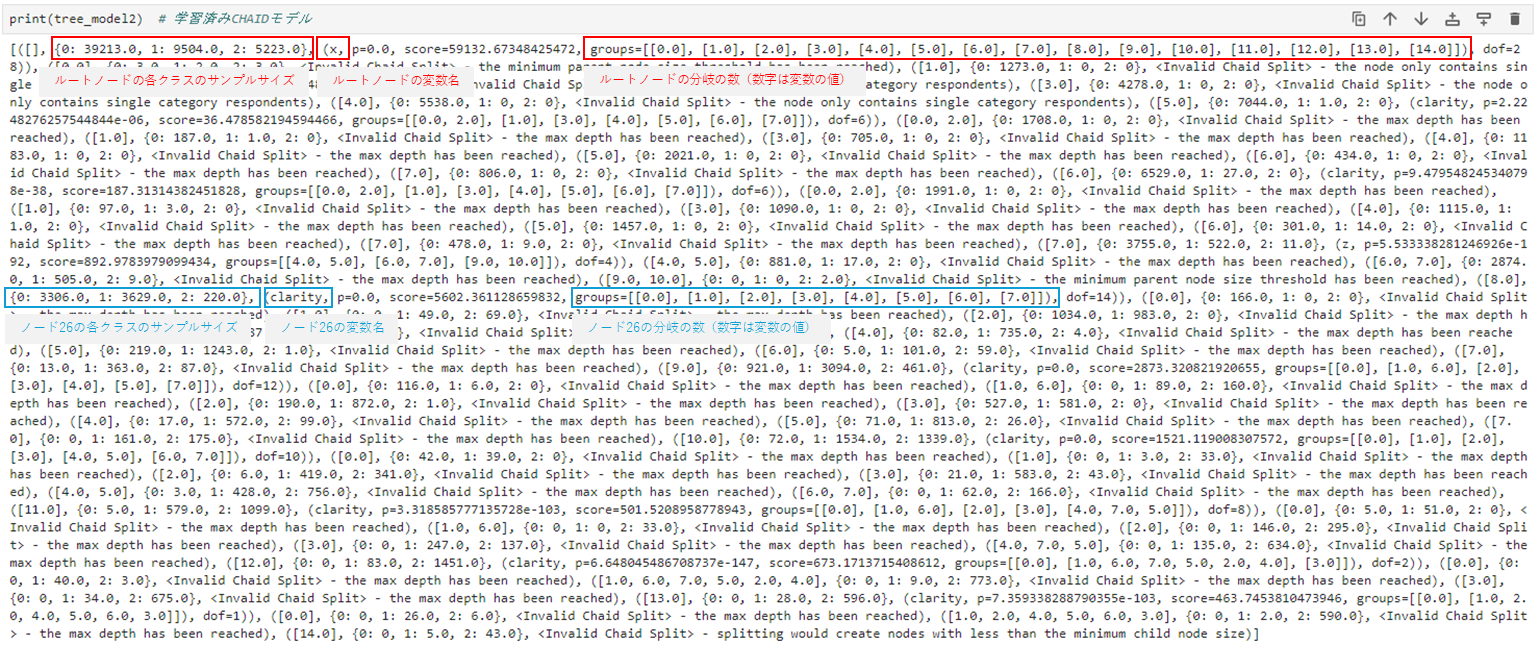
このあたりの情報を抜き出し、CARTで使った可視化関数を、CHAID用にカスタマイズしているのがmakeTreeEte3CHAID関数。情報を抜き出している箇所は以下のコードの部分である。
tree_n_node_samples = [sum([v for v in tree_model.get_node(i).members.values()]) for i in range(tree_model.node_count)]
tree_node_count = tree_model.node_count
tree_value = [np.array([v for v in tree_model.get_node(i).members.values()]) for i in range(tree_model.node_count)]
tree_value_sum = [sum([v for v in tree_model.get_node(i).members.values()]) for i in range(tree_model.node_count)]
tree_node_id = [tree_model.get_node(i).node_id for i in range(tree_model.node_count)]
tree_column = [tree_model.get_node(i).split.column for i in range(tree_model.node_count)]
tree_groupings = [ast.literal_eval(tree_model.get_node(i).split.groupings) for i in range(tree_model.node_count)]
tree_children = [tree_model.to_tree().children(i) for i in range(tree_model.node_count)]
各ノードのサンプルサイズ、ノードの数、ノードのクラス別のサンプルサイズとその合計、ノードID、ノードのカラム名、ノードの分岐条件、子ノードのIDをそれぞれ定義して、それを可視化の材料に使っている。
CHAIDパッケージでモデル学習&可視化。
CHAIDパッケージは質的変数に順序尺度か名義尺度かを指定できるのでtypes_dicとして指定している。
# CHAIDパッケージによりモデル構築
types_dic = dict(zip(colx[3:], ['ordinal'] * len(colx[3:]))) # 一部の変数が順序尺度と指定
types_dic2 = dict(zip(colx[:3], ['nominal'] * len(colx[:3]))) # 一部の変数が順序尺度と指定
types_dic.update(types_dic2) # dfのカラムの順番と揃えておく必要はある?一応揃えてはいる。
print(types_dic)
# モデル構築
tree_model2 = chaidTree.from_pandas_df(df_add, types_dic, coly, max_depth=2)
obj_unique = np.sort(df_add[coly].unique())
tree = makeTreeEte3CHAID(tree_model2, obj_unique, le_list_rename=bin_dict
, fsize=800, height=300, width=1200
, diameter=100, rotation=90, node_face_type='pie'
, name='CHAIDpackage_CHAID') # CHAIDpackage_CHAID.svgが保存される
# Jupyter上に可視化(Jupyter上だとテキストが表示されない場合がある。保存したファイルにはテキストがあるはず。)
ts = TreeStyle()
ts.show_leaf_name = True
ts.rotation = 90
tree.render("%%inline", tree_style=ts, h=300, w=1200)
CHAIDパッケージをsklearnライクに使えるsklearn2pmmlでも可視化できるようにしている。(sklearn2pmmlでは順序尺度の指定はできないが…。)
# sklearn2pmml パッケージのCHAIDClassifierによりモデル構築
# このCHAIDのwrapperでは順序尺度を選択できない(名義尺度のみ)
config = {"max_depth" : 2}
model = CHAIDClassifier(config = config)
model.fit(df_add[colx], df_add[coly])
obj_unique = np.sort(df_add[coly].unique())
tree = makeTreeEte3CHAID(model, obj_unique, le_list_rename=bin_dict
, fsize=800, height=300, width=1200
, diameter=100, rotation=90, node_face_type='pie'
, name='CHAIDClassifier_CHAID') # CHAIDClassifier_CHAID.svgが保存される
# Jupyter上に可視化(Jupyter上だとテキストが表示されない場合がある。保存したファイルにはテキストがあるはず。)
ts = TreeStyle()
ts.show_leaf_name = True
ts.rotation = 90
tree.render("%%inline", tree_style=ts, h=300, w=1200)
RのCHAIDと比較
RにもCHAIDパッケージがあるので比較してみる。
まずは先ほどと同様、質的変数へデータ変換するところまでをPythonで実施。
# CHAIDは説明変数が質的データのみ適用可
# すべてのデータを質的データ化する
df_add = dfle.copy()
# ビニングにより順序尺度データに変換
bin_dict = {} # 各カラムのビニング後の値とビニング前の数値の幅を辞書化する
dtype_arr = {} # 各カラムのデータ型指定('object'=質的データ。その他=連続値。)
category_cols = category_cols # 質的データ
continuous_cols = colx[3:] # 量的データから質的データへ
for i, col in enumerate(np.concatenate([category_cols,continuous_cols])):
if col in continuous_cols:
dtype_arr[col] = 'object'
# 1次元クラスタリングで順序尺度化
est = KBinsDiscretizer(n_bins=15, strategy="kmeans", encode='ordinal')
bins_ = est.fit_transform(df_add[col].to_numpy().reshape(-1, 1))
df_add[col] = bins_
binmast = pd.DataFrame({'org':df[col].to_numpy(), 'bins':bins_.reshape(-1)})
binmast_min = binmast.groupby('bins')['org'].min().reset_index()
binmast_max = binmast.groupby('bins')['org'].max().reset_index()
binmast = pd.merge(binmast_min, binmast_max, on=['bins'], how='outer', suffixes=['_min','_max'])
binmast['ranges'] = binmast['org_min'].round(2).astype(str)+'-->'+binmast['org_max'].round(2).astype(str) # ビン化する前の数値の範囲(例:'11.61-->12.7')
bin_dict_val = {row['bins']:row['ranges'] for index, row in binmast.iterrows()}
bin_dict[col] = bin_dict_val
else:
dtype_arr[col] = 'object'
bin_dict[col] = ''
print(dtype_arr)
bin_dict.update(le_list) # ラベルマスタ
print(bin_dict)
display(df_add.head())
RでCHAIDによる決定木可視化まで実施していく。
import rpy2
from rpy2 import robjects # Rを起動
%load_ext rpy2.ipython
%%R
library('tidyverse')
library('CHAID')
library('caret')
library('doParallel')
# Python側のデータを持ってくる
%R -i df_add
%R -i colx
%R -i coly
%R print(colx)
%R print(coly)
%%R
# factor型に変更
df_add_f <- df_add %>% mutate(across(everything(), as.factor))
# NaNを除外
# df_add_f <- na.omit(df_add_f)
print(nrow(df_add_f))
print(ncol(df_add_f))
head(df_add_f)
# 使用コア数
%R cl <- makePSOCKcluster(16)
%R registerDoParallel(cl)
# 変数名の配列定義
%R cols <- c(colx,coly)
%R head(df_add_f[, cols])
CHAID実行。
%%R
# CHAIDモデルの構築
# alpha2: カテゴリーをマージするかどうかを決定するために使用されるp値の閾値。この値を小さくするとマージが起こりにくくなり、より多くのカテゴリーが残る。デフォルト値は0.05
# alpha3: 分割後のノードでカテゴリーをマージするかどうかを決定するために使用されるp値の閾値。デフォルト値は-1(使用しない)
# alpha4: ノードを分割するかどうかを決定するために使用されるp値の閾値。この値を小さくすると分割が起こりにくくなり、より小さな木になる。デフォルト値は0.05
# minprob: 親ノードに対する子ノードのサイズの最小割合を指定する。この値を大きくすると、小さな子ノードが生成されにくくなり、より小さな木になる。デフォルト値は0.01
# minsplit: 分割の最小サイズ。ノードを分割するために必要な最小のレコード数を指定する。この値を大きくすると、小さなノードが生成されにくくなり、より小さな木になる。デフォルト値は20
# minbucket: 末端ノードの最小サイズ。末端ノード(葉ノード)に含まれるべき最小のレコード数を指定する。この値を大きくすると、小さな末端ノードが生成されにくくなり、より小さな木になる。デフォルト値は7
chaid_model <- chaid(target ~ .,
data = df_add_f[, cols],
control = chaid_control(alpha2=0.01, alpha3=-1, alpha4=0.1, maxheight=2, minprob = 0.01, minsplit = 100, minbucket = 100)
)
# PDFとして保存
pdf( "R_CHAID.pdf", width = 20, height = 10 )
plot(chaid_model
, gp = gpar(lty = "solid", lwd = 1, fontsize = 6, cex.node = 1., shape = "ellipse")
, type = "simple"
)
dev.off()
RのCHAID結果
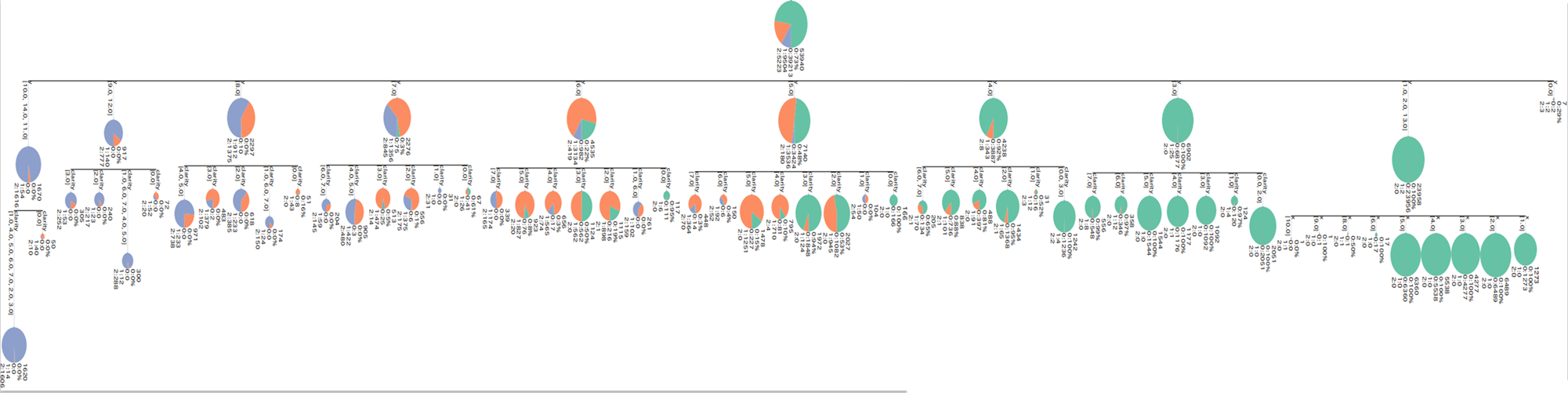
Rの第1分岐は変数yで9個に分かれる --> [0,1,2,13],[3],[4],[5],[6],[7],[8,12],[9],[10,11,14]
すべて名義尺度変数のPythonのCHAID結果とおおよそ同じ。
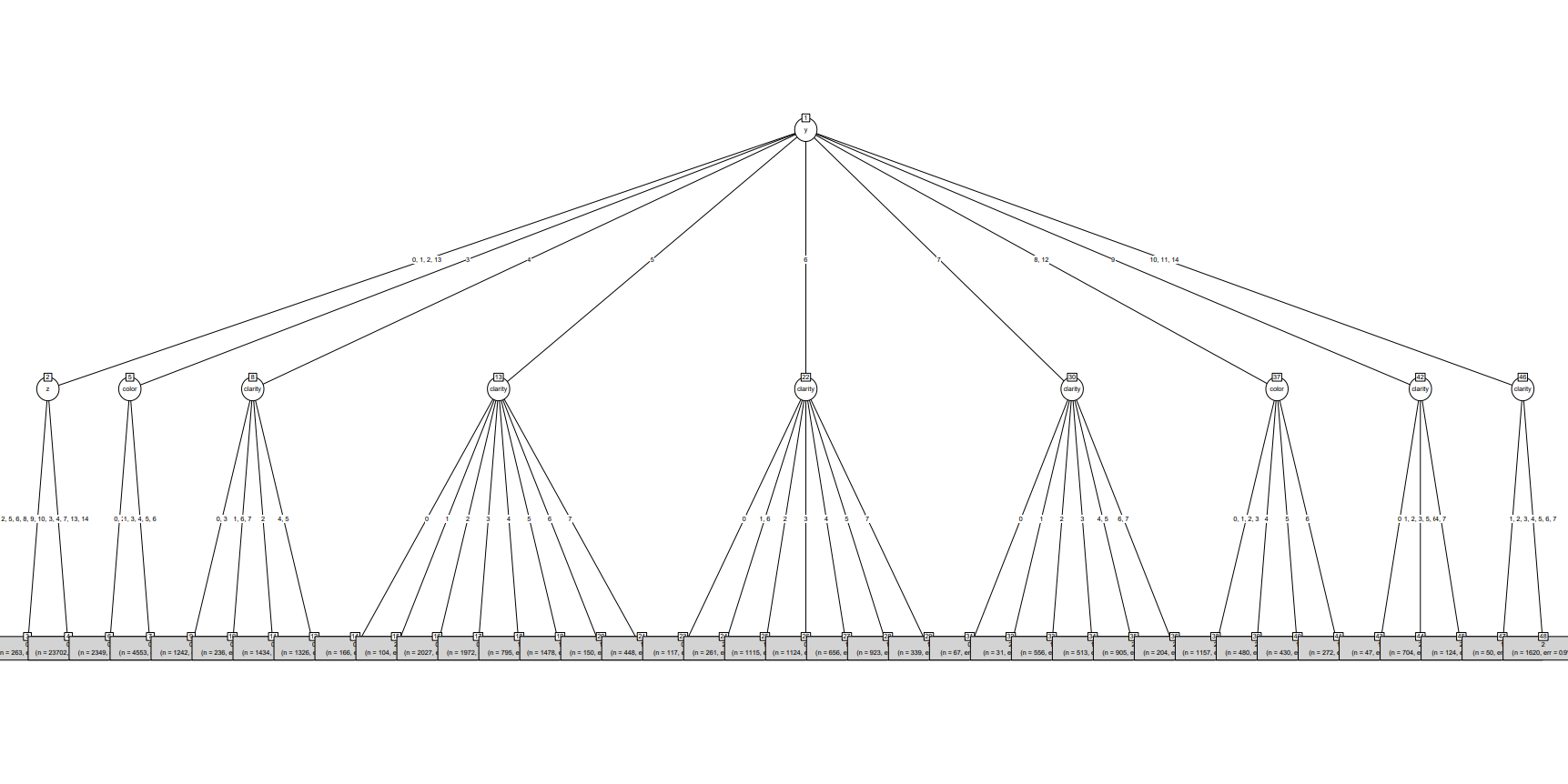
Pythonの第1分岐は変数yで10個に分かれる --> [0],[1,2,13],[3],[4],[5],[6],[7],[8],[9,12],[10,11,14]
おわりに
ETE Toolkitを使って、CART、CHAIDの決定木可視化を実施した。
SPSSのCHAIDは優秀だけど、有料ソフトなのでPythonでCHAIDできたらいいなぁと思っていたが可視化が壊滅的だったので避けていた。
今回の取り組みでPythonのCHAIDも使いやすくなった気がする。
以上!