Psi4による量子化学計算
物質はたくさんの分子の集合体です。それらの分子が物質の中でどのように配列しているかというのは、その物質の物性に影響します。そして、分子の配列を左右するのが、分子間相互作用です。したがって、分子間相互作用の理解は重要です。
近年、さまざまな量子化学計算ソフトが開発され、専門知識がなくても、お金をかけず、すぐに計算を行える環境が整ってきています。今回はフリーのソフトで、研究にも用いれらているPsi41というプログラムを使って導入から分子間相互作用計算までを実施しようという内容です。
次のサイトで詳しく解説があるのでそれを参考にしています。
ここで、Psi4はコマンドラインから実行可能プログラムPsi4を直接動作させる方法と、Pythonのモジュールとして実行する方法とがあります。公式のドキュメント2において、実行可能プログラムPsi4を直接動作させる方をPsithonと呼び、Pythonのモジュールとして実行する方をPsiAPIと呼んでいます。計算方法やインプットファイルの記述方法がそれぞれ異なるので別々の呼び名で呼ばれているのだと思います。今回はAnaconda Promptから実行可能プログラムPsi4を直接動作させます。つまりPsithonの方でやります。
環境
・Windows 10 Pro
すでにPython導入済み
すでにAnaconda導入済み
Psi4の導入
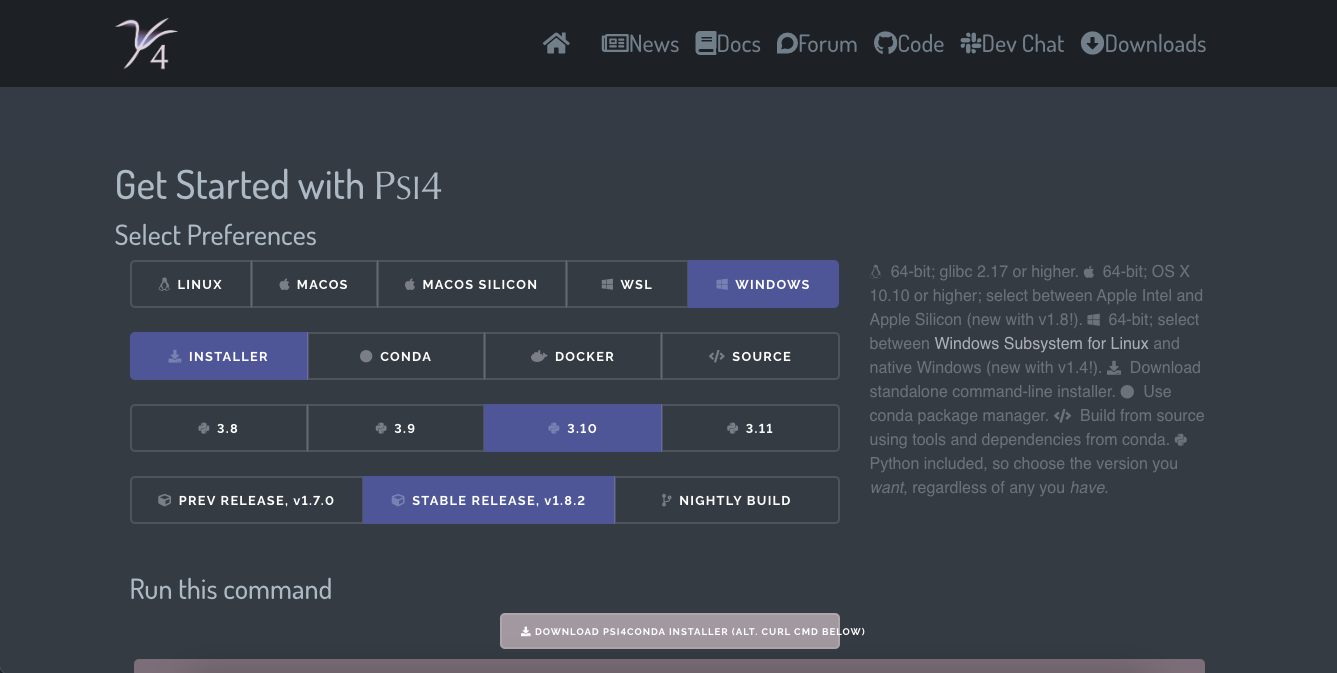
1)Psi4の公式サイトにアクセス
2)select preferencesから次を選択
→windows, installer, 3.10, stable release v1.8.2
3)DOWNLOAD PSI4CONDA INSTALLER (ALT. CURL CMD BELOW)を押す。インストーラーのexeファイル(Psi4conda-1.8.2-py310-Windows-x86_64.exe)がインストールされます
4)Psi4conda-1.8.2-py310-Windows-x86_64.exeを実行
5)Psi4conda 1.8.2 (64-bit) Setupが出てくるので、それに従いセットアップを行う。デフォルト通りにI Aguree, Next, Installを選択していきました
6)インストールが完了したらAnaconda Promptを立ち上げる
7)Anaconda Promptで次を実行し、Psi4のconda環境をアクティブにする(...には適切なディレクトリを指定)
conda activate C:\Users\...\psi4conda
8)Anaconda Promptに次のコマンドを入力してテストを実行しインストールされていることを確認する
psi4 --test
inputファイル
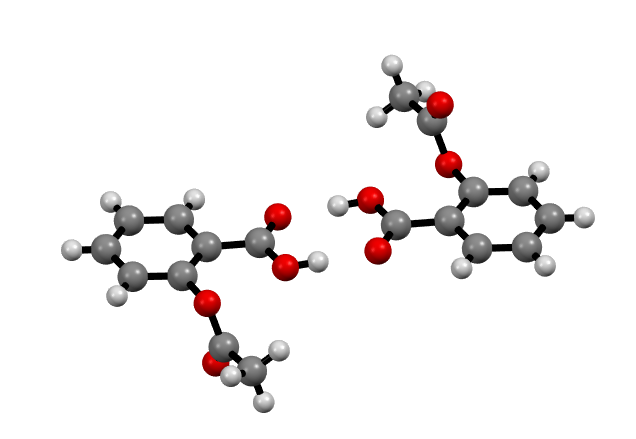
今回、薬効物質であるアスピリンの二量体間の分子間相互作用を計算してみます。これは別記事でCrystalExplorerというフリーソフトを使って計算した分子です。この別記事と合わせるため、インプットデータは結晶構造のcifファイルのデータをもとに作成しています。cifデータから計算に用いるdatファイルへの変換についてもまた別記事で紹介します。このcifデータはCrystallography Open Database(COD)3から無料でダウンロードしています。ファイル名は1515581.cif4です。CODからダウンロードした結晶構造中のアスピリンの二量体を以下に示します。
また、この構造をもとに作ったdatファイルの中身は以下になります。ファイル名はinput.datとしてあります。
memory 600 mb
molecule_dimer {
0 1
O 2.940102 -0.918391 4.082164
H 3.458846 -0.161802 4.194509
O 3.469622 -1.216108 6.225052
O 1.427586 -2.710257 2.851318
O -0.275446 -1.416743 3.548998
C 2.130667 -2.860021 5.190442
C 1.429665 -3.367434 4.091122
C 0.765145 -4.569950 4.160890
H 0.310709 -4.905852 3.397763
C 0.757408 -5.290295 5.342941
H 0.289139 -6.115487 5.393844
C 1.433557 -4.805534 6.451851
H 1.427715 -5.295472 7.265565
C 2.113929 -3.610137 6.369858
H 2.581935 -3.288474 7.131720
C 2.899197 -1.590842 5.206145
C 0.525220 -1.697632 2.701981
C 0.684015 -1.042655 1.378286
H 0.037567 -0.310661 1.296293
H 1.593416 -0.685395 1.297347
H 0.525781 -1.698926 0.668171
--
0 1
O 4.990314 0.918391 6.456804
H 4.471571 0.161803 6.344459
O 4.460795 1.216108 4.313916
O 6.502830 2.710257 7.687650
O 8.205862 1.416743 6.989971
C 5.799749 2.860021 5.348526
C 6.500751 3.367434 6.447846
C 7.165272 4.569950 6.378078
H 7.619707 4.905852 7.141205
C 7.173009 5.290295 5.196028
H 7.641278 6.115487 5.145124
C 6.496859 4.805534 4.087117
H 6.502701 5.295472 3.273404
C 5.816488 3.610137 4.169110
H 5.348482 3.288474 3.407248
C 5.031220 1.590842 5.332823
C 7.405197 1.697632 7.836988
C 7.246401 1.042655 9.160682
H 7.892849 0.310661 9.242675
H 6.337001 0.685395 9.241621
H 7.404635 1.698926 9.870798
units angstrom
}
set globals {
basis 3-21g
}
set sapt {
print 1
}
energy('sapt0')
計算の実行
1)Anaconda promptを開けます
2)Anaconda Promptで次を実行し、Psi4のconda環境をアクティブにする(...には適切なディレクトリを指定)
conda activate C:\Users...\psi4conda
3)cdコマンドでinput.datを保存したディレクトリへ移動
4)次を実行
psi4 input.dat output.dat
計算が終わると、計算結果をoutput.datに出力してくれます。自分のPCでは計算時間が9分20秒でした。
計算結果
output.datには色々なことが書いてあるので、相互作用エネルギーの計算結果が書いてある箇所を抜粋します。
SAPT Results
--------------------------------------------------------------------------------------------------------
Electrostatics -60.24665018 [mEh] -37.80534375 [kcal/mol] -158.17755827 [kJ/mol]
Elst10,r -60.24665018 [mEh] -37.80534375 [kcal/mol] -158.17755827 [kJ/mol]
Exchange 53.53230609 [mEh] 33.59202922 [kcal/mol] 140.54905027 [kJ/mol]
Exch10 53.53230609 [mEh] 33.59202922 [kcal/mol] 140.54905027 [kJ/mol]
Exch10(S^2) 52.61482928 [mEh] 33.01630383 [kcal/mol] 138.14021524 [kJ/mol]
Induction -19.71237464 [mEh] -12.36970183 [kcal/mol] -51.75483248 [kJ/mol]
Ind20,r -29.13733203 [mEh] -18.28395189 [kcal/mol] -76.50005470 [kJ/mol]
Exch-Ind20,r 17.02948819 [mEh] 10.68616517 [kcal/mol] 44.71091508 [kJ/mol]
delta HF,r (2) -7.60453080 [mEh] -4.77191512 [kcal/mol] -19.96569286 [kJ/mol]
Dispersion -5.63403486 [mEh] -3.53541025 [kcal/mol] -14.79215648 [kJ/mol]
Disp20 -7.32312603 [mEh] -4.59533096 [kcal/mol] -19.22686473 [kJ/mol]
Exch-Disp20 1.68909117 [mEh] 1.05992071 [kcal/mol] 4.43470825 [kJ/mol]
Disp20 (SS) -3.66156301 [mEh] -2.29766548 [kcal/mol] -9.61343237 [kJ/mol]
Disp20 (OS) -3.66156301 [mEh] -2.29766548 [kcal/mol] -9.61343237 [kJ/mol]
Exch-Disp20 (SS) 0.74570109 [mEh] 0.46793450 [kcal/mol] 1.95783795 [kJ/mol]
Exch-Disp20 (OS) 0.94339008 [mEh] 0.59198621 [kcal/mol] 2.47687030 [kJ/mol]
Total HF -26.42671873 [mEh] -16.58301637 [kcal/mol] -69.38334047 [kJ/mol]
Total SAPT0 -32.06075359 [mEh] -20.11842661 [kcal/mol] -84.17549695 [kJ/mol]
Special recipe for scaled SAPT0 (see Manual):
Electrostatics sSAPT0 -60.24665018 [mEh] -37.80534375 [kcal/mol] -158.17755827 [kJ/mol]
Exchange sSAPT0 53.53230609 [mEh] 33.59202922 [kcal/mol] 140.54905027 [kJ/mol]
Induction sSAPT0 -18.80588920 [mEh] -11.80087364 [kcal/mol] -49.37485530 [kJ/mol]
Dispersion sSAPT0 -5.54412396 [mEh] -3.47899031 [kcal/mol] -14.55609546 [kJ/mol]
Total sSAPT0 -31.06435726 [mEh] -19.49317848 [kcal/mol] -81.55945875 [kJ/mol]
--------------------------------------------------------------------------------------------------------
ここもたくさんの値が表示されていますが、とりあえずここでは最後の5行に示されているsSAPR0の値を見てみます。各値については公式ドキュメントを参照してください5。sSAPR0ではただ分子間相互作用を計算するのみならず、そのエネルギーの寄与を計算するエネルギー分割計算も行ってくれています。つまり分子間相互作用(Total sSAPT0)を、点電荷や永久双極子間の相互作用である静電相互作用(Electrostatics sSAPT0)、主に斥力となる交換相互作用(Exchange sSAPT0)、永久双極子-励起双極子間の相互作用である誘起力(Induction sSAPT0)、励起双極子-励起双極子間の相互作用である分散力(Dispersion sSAPT0)に分けてくれます。
アスピリンの二量体間の相互作用は-81.5 kJ/molでした。そのエネルギー分割計算の結果から静電相互作用が-158.1 kJ/molと大きく、二量体間には静電的な相互作用が強く働いていることが示唆されます。これは、二つの分子のカルボキシル基の水素原子と酸素原子間の静電相互作用、つまり水素結合が働いているからではないかと考察できます。この計算結果は、別記事でのCrystalExplorerによる計算と、傾向が一致します。
今日はここまで。
-
Pi4の参考文献
https://pubs.aip.org/aip/jcp/article/152/18/184108/972964/PSI4-1-4-Open-source-software-for-high-throughput ↩ -
Psi4の公式ドキュメント
https://psicode.org/psi4manual/master/index.html ↩ -
Crystallography Open Database
http://www.crystallography.net/cod/ ↩ ↩ -
COD ID:1515581
アスピリンの結晶構造の文献。
Varughese, Sunil, et al. Chemical Science 2.11 (2011): 2236-2242. ↩ ↩ -
Psi4の公式ドキュメントの分子間相互作用についてhttps://psicode.org/psi4manual/master/sapt.html ↩