はじめに
こんにちは!
皆さん次元圧縮手法といえばどのようなものを思いつきますか?
PCA,Isomap,t-SNEなどでしょう。
こちらの記事が大変よくまとめられています。
高次元データの次元削減および2次元プロット手法
今日ご紹介したいのはUMAPという次元削減手法です。
論文がarXivに2018年2月9日に上がったばかりの手法です。
なんと、t-SNEと同程度の次元削減を数倍の速さでできてしまいます。
論文については、リーマン幾何学と代数トポロジーを背景にされているようなのでじっくり読み込んで後日まとめたいと思います。
興味のある方はこちらからどうぞ
UMAP: Uniform Manifold Approximation and Projection for Dimension Reduction
使ってみよう
なんと早くもライブラリが公開されています。
ひとまず使ってみましょう。
インストール
pip3 install umap-learn
使い方は基本的にscikit-learnと同じです。
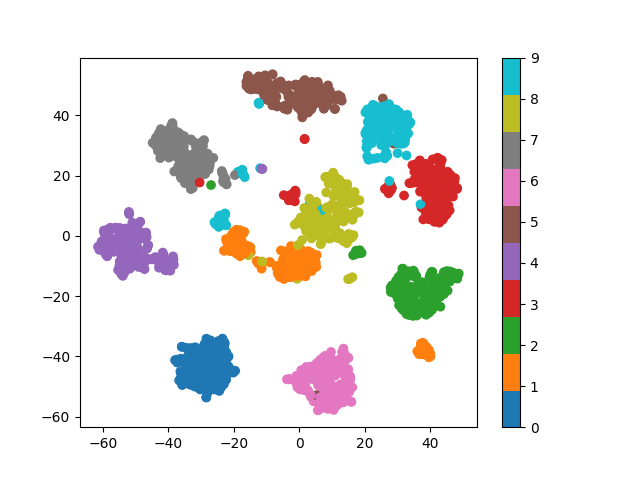
MNISTを使ってt-SNEと比較してみました。
t-SNEによるイケてる次元圧縮&可視化
公式GitHubには書いてあるのですが、
from scipy.sparse.csgraph import connected_components
を書かないとエラーが出てしまいます。
import umap
from sklearn.datasets import load_digits
from scipy.sparse.csgraph import connected_components
import matplotlib.pyplot as plt
import matplotlib.cm as cm
from sklearn.manifold import TSNE
import time
def main():
digits = load_digits()
digits.target = [float(digits.target[i]) for i in range(len(digits.target))]
# UMAP
start_time = time.time()
embedding = umap.UMAP().fit_transform(digits.data)
interval = time.time() - start_time
plt.scatter(embedding[:,0],embedding[:,1],c=digits.target,cmap=cm.tab10)
plt.colorbar()
plt.savefig('umap.png')
# t-SNE
plt.clf()
start_time2 = time.time()
tsne_model = TSNE(n_components=2)
tsne = tsne_model.fit_transform(digits.data)
interval2 = time.time() - start_time2
plt.scatter(tsne[:,0],tsne[:,1],c=digits.target,cmap=cm.tab10)
plt.colorbar()
plt.savefig('tsne.png')
print('umap : {}s'.format(interval))
print('tsne : {}s'.format(interval2))
if __name__ == "__main__":
main()
UMAPの方がよりはっきり分かれている印象ですね。
驚くべきは実行時間の差です。
umap : 5.383596897125244s
tsne : 34.21198582649231s
なんと僕の出力結果では6倍以上の速度を計測しました。
大変興味深いですね。
数学の復習をしながら論文を読んで、解説記事を更新したいと思います。