昨日の記事でも書いたように、ワークフローに文章や画像を関連づけさせたいのだが、PapermillというJupyter Notebookをバッチ実行するためのPythonパッケージがあったので、これを使ってワークフローを構築した。
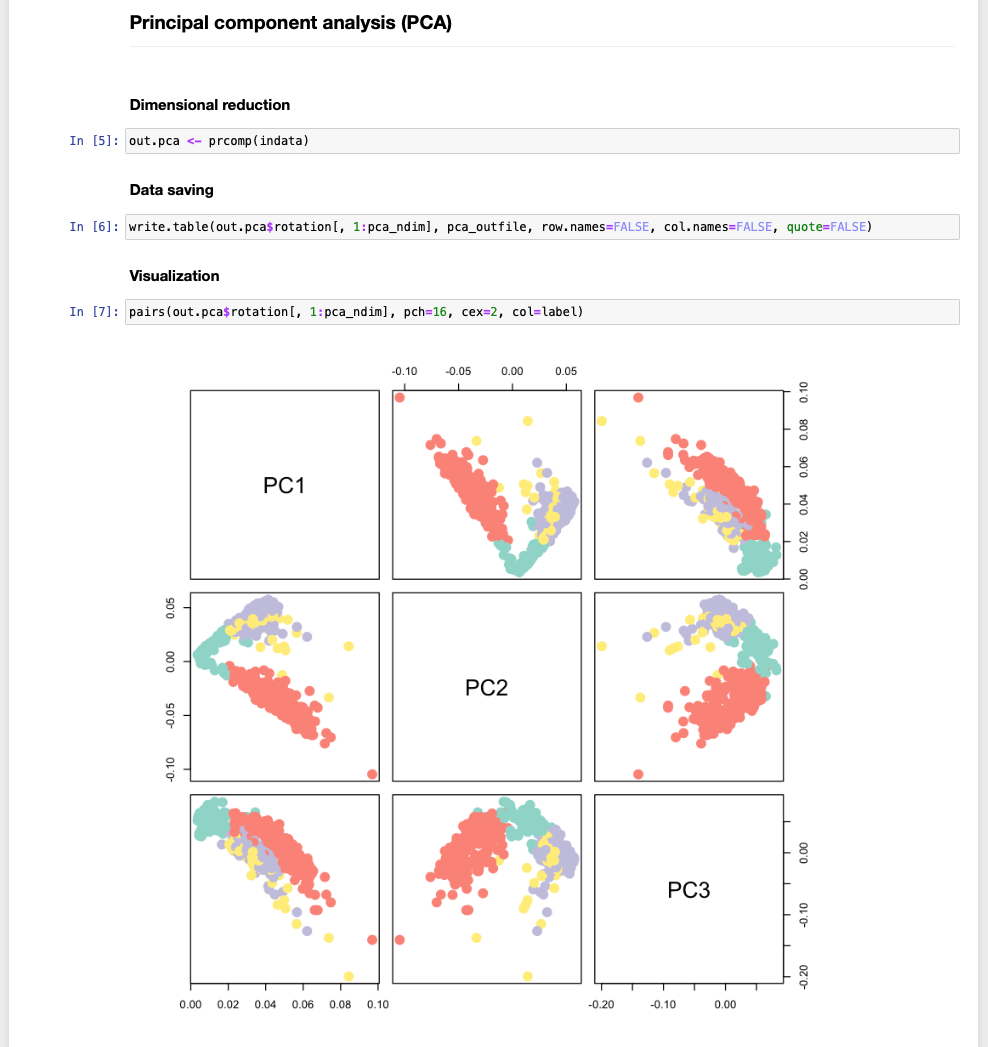
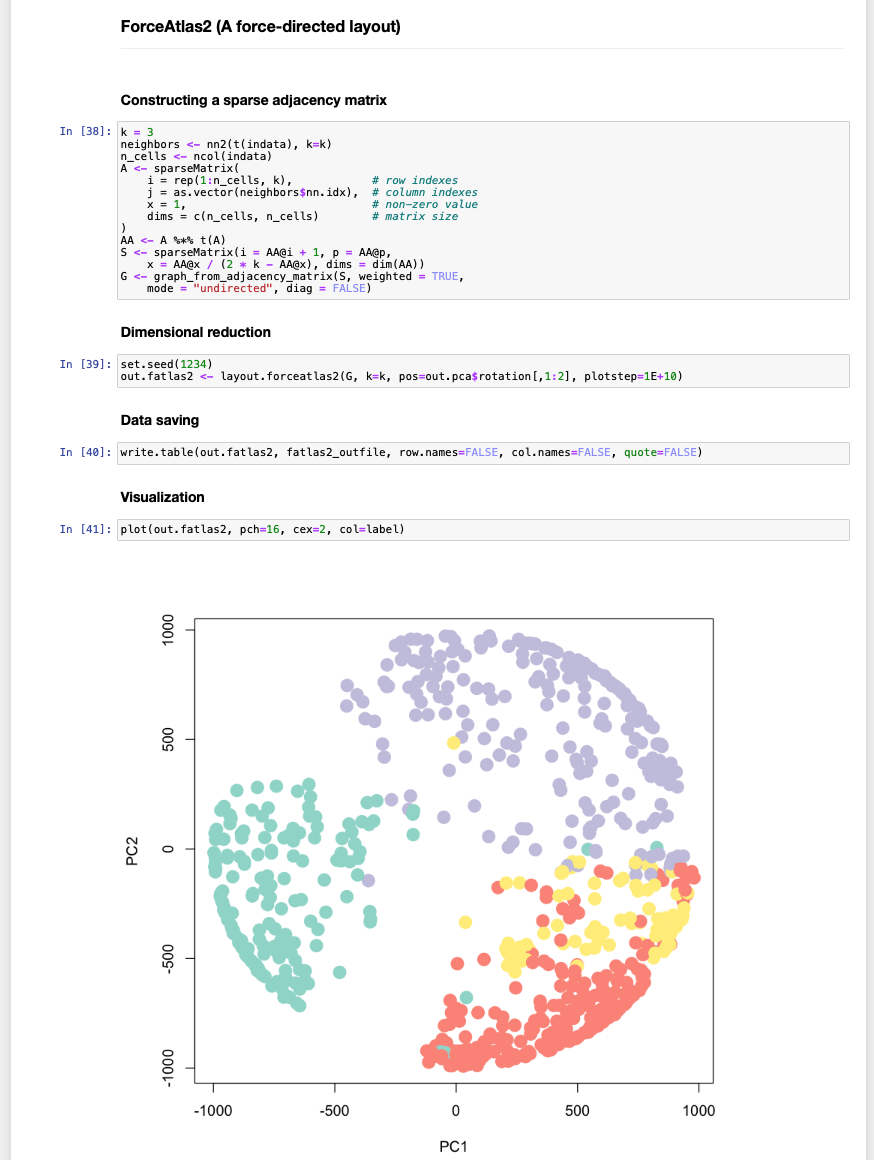
ここでは1細胞RNA-Seqのデータdata/input.csvと、ラベル情報data/label.csvというファイルに対して、複数の次元圧縮を試すというタスクを一度に行った。
なお、RをJupyter notebookで使えるようにするために、IRkernelのインストールと、.ipynbファイルのkernelspecのところを以下のように書き換えた。
"kernelspec": {
"display_name": "R",
"language": "R",
"name": "ir"
},
あとは、
jupyter notebook workflow.ipynb
で、ノートブックを起動して、上から順にひたすら次元圧縮を試すノートブックを作り込んだ後に、入力、出力ファイルの場所や、圧縮次元数を後からPapermillの引数として渡せるように書いたら、以下のようにPapermillを実行した。
papermill workflow.ipynb output/papermill/output.ipynb \
-p infile data/input.csv -p infile2 data/label.csv -p \
outdir output/papermill -p pca_ndim 3 -p nmf_ndim 4 \
-p mds_ndim 4 -p lda_ndim 3 -p som_ndim 2 -p isomap_ndim 2 \
-p lle_ndim 2 -p dmap_ndim 4 -p tsne_ndim 2 -p umap_ndim 2 \
-p tumap_ndim 2 -p fatlas2_ndim 2
これにより、output/papermillのところに、次元圧縮の結果ファイルやoutput.ipynbというノートブックが出力されるようになる。papermillで指定されたパラメーターはノートブックに埋め込まれているのがわかる。
https://github.com/kokitsuyuzaki/workflow_benchmark/blob/master/output/papermill/output.ipynb
https://colab.research.google.com/github/kokitsuyuzaki/workflow_benchmark/blob/master/output/papermill/output.ipynb
以下のように出力されたほうのノートブックを起動しておくと、実行途中の様子を確認できるのも便利。
jupyter notebook output/papermill/output.ipynb
ただし、昨日の記事でも書いたように、実データではパラメーターチューニングしたいのと、パラメーターの値次第ではコケるため、この書き方だと全計算をやり直す必要が出てくるので、各次元圧縮手法ごとにノートブック化&Dockerコンテナ化して、複数のパラメーターごとに分散処理するような使い方のほうがいいかもしれない。
参考
https://qiita.com/hrappuccino/items/d4961e1e42c82c52ef64
https://qiita.com/noko_qii/items/62a7f70fceb6153620f4