バイオインフォマティクスの研究をしていて、解析手法やデータベースを開発した際、R/Bioconductorパッケージとしてそれらを公開することは、以下のような点で良いことだと思う。
- 使いやすさ : パッケージ名(例 : snpStats ⇆ GOStats)、関数名(例 : snpStats ⇆ GOStats)、引数の与え方など、似たもの同士は似たように動作するようにしている
- 再現性 : 毎日全パッケージに対してビルドチェック( https://www.bioconductor.org/checkResults/ )が走っているので、動かなくなるとコアチームからメールが飛んでくる
- 持続性 : 少なくともある環境ではきちんと動作し続けている事が保証されている
BioconductorはCRANと比べると、かなりレビューが厳しめらしく、他のパッケージとの連携や使いやすさについてコアチームから色々注文を受ける。それら解析手法やデータを、二次利用するユーザーからすると、大変素晴らしいことだと思うが、パッケージ開発者側からすると、論文で方法論の新しさや、生物学的な面白さを問われている中、書いたコードにまで、色々言われるのは正直めんどくさい。**書いたコードを綺麗にしたり、使いやすくしても、誰も褒めてくれないし、論文にはならない。**そのため、RパッケージをGitHubに置いておくだけの"野良"パッケージや、コードを論文に添付しただけでソフトウェアと言っているものもかなり多い。自分としては、ソフトウェアや実装自体に対して、もう少し評価するものがあれば、Bioconductor投稿のモチベーションが上がるのにと思っていたが、2018年にF1000Reseach誌で、BiocWorkflowToolsというR/Bioconductorパッケージの論文が投稿され、このツールがそこらへんの問題を解決するかもしれないと思ったので、ここで紹介する。
BiocWorkflowToolsとは
このR/Bioconductorパッケージの目的は、Rマークダウンファイル(.Rmd)をF1000Reseach誌が指定する形式のPDFに変換し、F1000Reseach誌への投稿を促す事である。このドキュメント(Rではvignetteという)の作成は、Bioconductorでは義務なので、基本的に全開発者が行う作業である。なので、「Bioconductorにパッケージを投稿、ついでに論文にも投稿」というような事が原理的には可能になる。うまくいけば、査読付き論文としてカウントされるので、これならパッケージを作ってみようという気になりそう。
使い方
BiocWorkflowToolsのvignette( http://bioconductor.org/packages/release/bioc/vignettes/BiocWorkflowTools/inst/doc/Generate_F1000_Latex.html )に従い、試しに使ってみた。
準備
まず、BiocWorkflowToolsをダウンロードする。
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install(version="3.10")
BiocManager::install("BiocWorkflowTools")
Rコマンドで使う場合
Rを起動後、以下のようにrmarkdown::draft関数を使って、ドキュメントの雛形を出力する
rmarkdown::draft(file = "MyArticle.Rmd",
template = "f1000_article",
package = "BiocWorkflowTools",
edit = FALSE)
system("tree MyArticle")
作成されたディレクトリの中を見てみると、以下のようなファイルができていることがわかる
MyArticle
├──── F1000header.png : F1000誌フォーマットにする際のヘッダーの画像(いじらない)
├──── MyArticle.Rmd : Rマークダウンファイル、これから書き込んでいき、vignette&F1000論文にする
├──── f1000_styles.sty : Latexのスタイルファイル(いじらない)
├──── frog.jpg : 論文に挿入される画像ファイル
└──── sample.bib : 論文の引用文献をまとめたLatexのbibファイル(適宜編集)
.Rmdをのぞいてみると、
**Package**: `r packageVersion("ExampleWorkflow")`
と書かれている箇所があり、ここに自分が開発した、今動作しているマシンにインストールされているパッケージの名前にする
試しに、自分のmeshrパッケージに書き換えてみた
**Package**: `r packageVersion("meshr")`
次に、Rマークダウンを、rmarkdown::render関数を使って、以下のように.texファイル、.pdfファイルに変換する
rmd_file <- "MyArticle/MyArticle.Rmd"
rmarkdown::render(input = rmd_file)
RStudioを使う場合
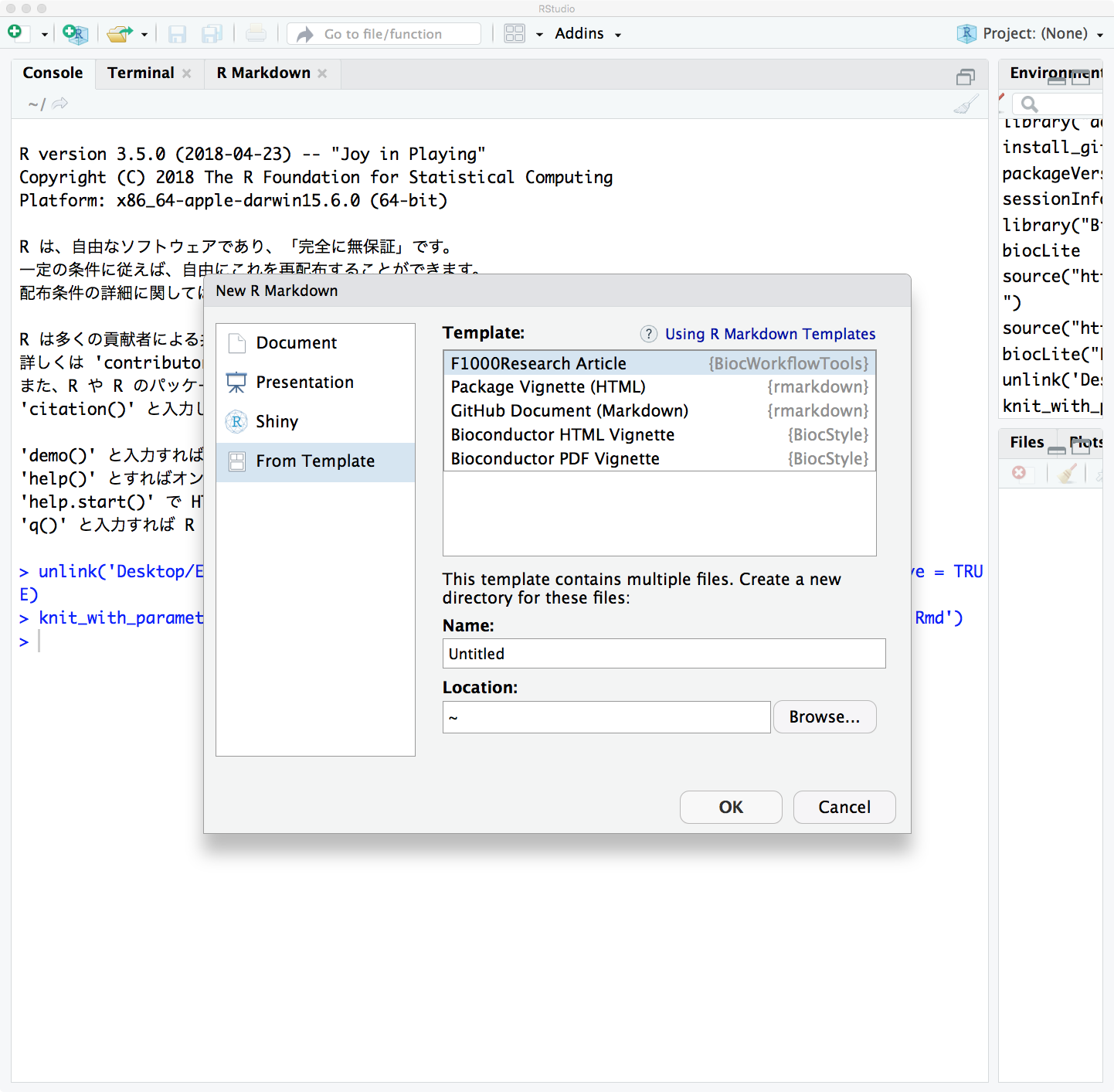
RStudio起動後、File > New File > R markdown... > Documentという順ですすむと、BiocWorkflowToolsがインストールされている場合、From Templateのところに、F1000Research Articleという雛形が選択できるようになる
この雛形を、自分がパッケージ開発しているディレクトリ(Location)に出力すると、RmdがRStudioのエディタ画面に表示されるので、上記と同様、
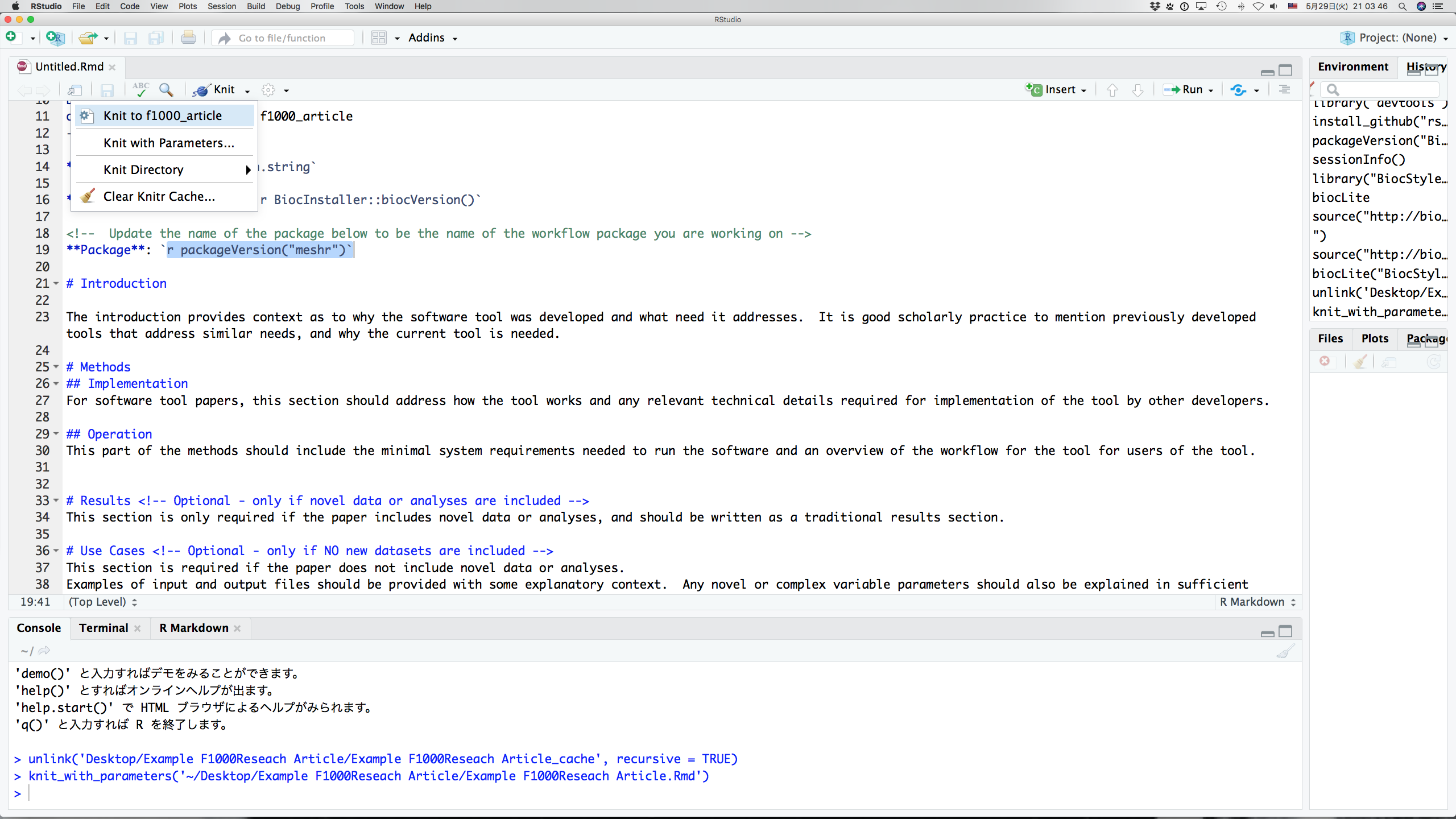
**Package**: `r packageVersion("ExampleWorkflow")`
のところを書き換えて、Knit > Knit to f1000_articleのボタンを押す
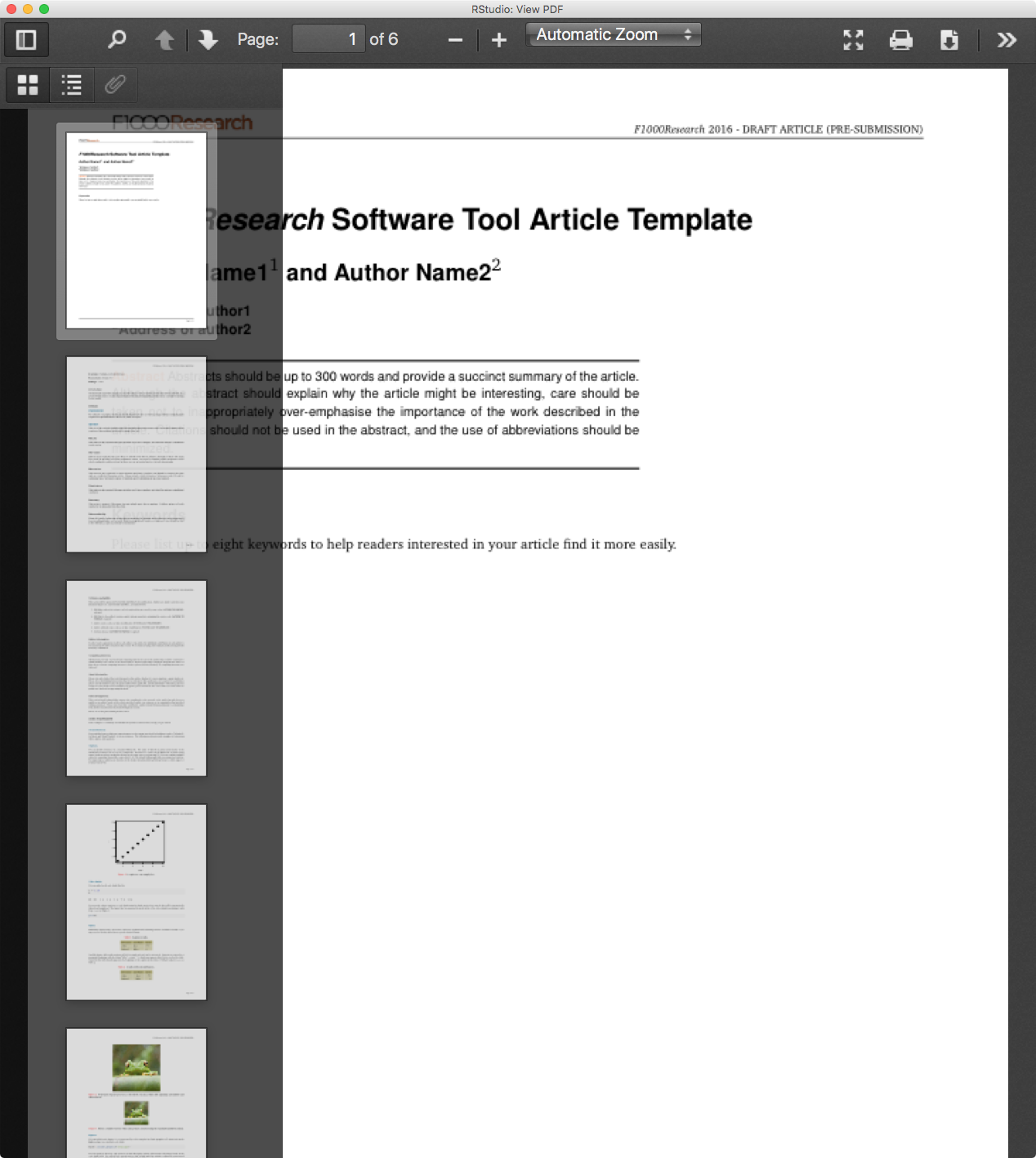
これにより、RmdがLatexとpdfに変換される
pdfはこのようにF1000の形式になっている
まとめ
BiocWorkflowToolsによって、F1000Reseach誌のフォーマットで、Rのドキュメントが作れるようになった。
また、
https://support.bioconductor.org/p/109535/
で言われているように、Bioconductorのvignetteとして、F1000Reseach誌のフォーマットをそのまま使っても良いし、HTMLを別途吐き出しして、そっちをvignetteにしても良い。なので、原理的には、vignette作成 = 論文執筆という感じにできそうだが、実際には、ツールのドキュメントと、論文はもとめられているものが違うので、全く同じ内容を書いていたものは見当たらなかった。
また以下のように、BioconductorのWorkflowという種類のパッケージと、F1000にWorkflow論文を投稿することは、1対1対応しているわけではないことに注意。
-
BioconductorでWorkflowパッケージになっていて、F1000でWorkflow論文になっている
- RNAseq123: https://f1000research.com/articles/5-1408/v3
- TCGAWorkflow: https://f1000research.com/articles/5-1542/v2
- RnaSeqGeneEdgeRQL: https://f1000research.com/articles/5-1438
- rnaseqDTU: https://f1000research.com/articles/7-952/v3
- chipseqDB: https://doi.org/10.12688/f1000research.7016.2
- CAGEWorkflow: https://doi.org/10.12688/f1000research.18456.1
- methylationArrayAnalysis: https://f1000research.com/articles/5-1281
- maEndToEnd: https://f1000research.com/articles/5-1384
- cytofWorkflow: https://f1000research.com/articles/6-748
-
BioconductorではWorkflowパッケージ扱いではないが、F1000にWorkflow論文を投稿している
-
複数のBioconductorパッケージを使って、F1000にWorkflow論文を投稿している
- single-cell関係のパッケージ群:
- Microbiome関係のパッケージ群: https://f1000research.com/articles/5-1492
- RNA-Seq関係のパッケージ群: https://f1000research.com/articles/4-1070
- Spatial Proteome関係のパッケージ群:
-
Bioconductorには登録していないが、F1000にWorkflow論文を投稿している
- singlecellworkflow: https://github.com/fperraudeau/singlecellworkflow