学習済みのLightGBMの中身をさっと見たいときに出来ることとして、以下の2点を紹介します
- 個々の木の描写
- 木々の構造をデータフレームに変換
準備
import lightgbm
import pandas as pd
import palmerpenguins
# version >= 3 が必要
print(lightgbm.__version__)
# dataの読み込み
df = palmerpenguins.load_penguins()
df.shape # (344, 8)
# 見やすいように若干使用データを修正する
X = df.drop(['species', 'island', 'flipper_length_mm'], axis=1)
X['sex'] = X['sex'].astype('category')
y = df['flipper_length_mm']
# 学習の実行
lgb = lightgbm.LGBMRegressor(n_estimators=2, num_leaves=8)
lgb.fit(X, y)
環境
lightgbmのバージョン3以上が必要です。Google colabではpip install lightgbm -Uで新しいバージョンのものを入れる必要がありました(2021年10月現在)。
また、後述のグラフの可視化の際にはgraphvizが必要です。
データ
今回はpalmerpenguinsのデータを使います。ペンギンかわいい。
pip install palmerpenguinsでインストールできます。

全部で344行8列のデータセットです。各列の詳細などは以下を参照ください。
学習
flipper_length_mm(羽の長さ)をtargetにした回帰を、sklearn APIを使って行います。
どんな感じで可視化できるのかイメージしやすいよう、木の数を2つ、木あたりの葉の数を8つに設定して学習します。
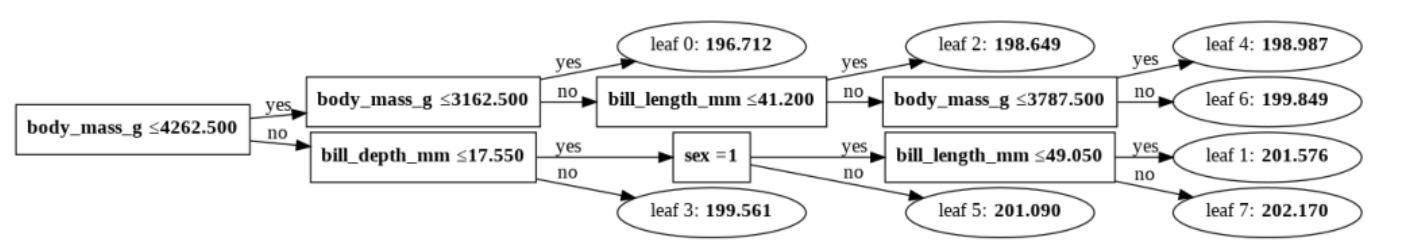
1. 木の描写
lightgbm.plot_tree(lgb, tree_index=0);
lightgbm.plot_tree(booster, tree_index)で結果を図示できます。tree_indexを省略すると一つ目の木が描写されます。
木が深いと見づらそうですが、自分で中身を確認するための可視化としては十分に思えます。人に見せる用のグラフを作成する場合は以下などを参考に作りたいところです。
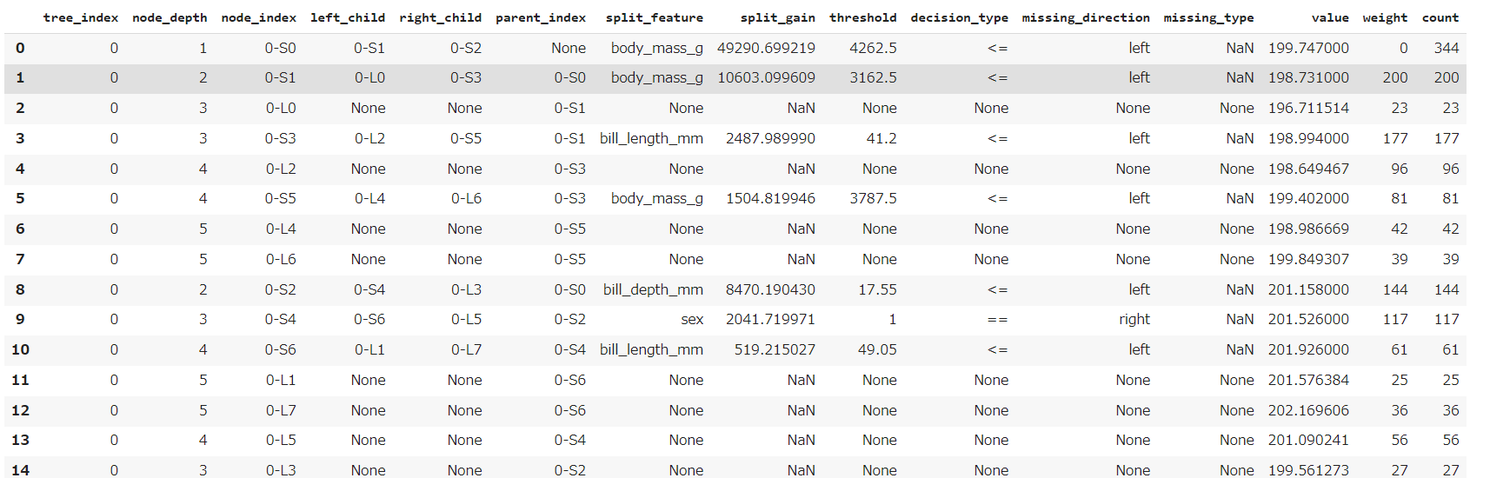
2. データフレームに変換
lgb.booster_.trees_to_dataframe()
trees_to_dataframe()で、木の構造をデータフレームにまとめてくれます。各列の持っている情報は以下の通りです。
- tree_index: 何本目の木か
-
node_depth ~ decision_type: 見たまんま
- node_indexはSがsplit、Lはleafを意味する(たぶん)。先頭の数字はtree_indexと同じ
- 特徴量がcategoryだとdecision_typeが
==になるっぽい(9行目) 。threshholdは数字のままだった。カテゴリ名を表示してくれまいかと期待したけど。残念。
-
missing_direction, missing_type: 欠損値について
- lightgbmだと
zero_as_missing=Trueで0を欠損値に指定することができて、その情報がmissing_typeには入ると思われ
- lightgbmだと
- value: そのノードでのpredicted valueに学習率を掛けたもの
-
count: そのノードに入るレコードの数
- 根ノードには全部入るので344だし、0-S1+0-S2も344になる
ぱっと思いつく使い方としては、
LGBMは最初の方の木に入っている特徴量が重要なので、関心がある特徴量が最初に出てくるのはどの木か見る、とか。
2つの特徴量が同じ木にばかり出てくるなら相互作用ありそうだな、とか。
これを使って特徴量重要度がどう計算されているのか理解したり、とか。
色々使えそうな気はします。使い慣れたpandasで返してくれるのがありがたいです。
参考