この記事は創薬 (dry) Advent Calendar 2019(#souyaku2019)の12/1分の記事です。
はじめに
インシリコ創薬やAI創薬と言う名前で情報科学的な手法が創薬に使われるようになって久しい。伝統的なインシリコ創薬はリガンドベース(Ligand Base Drug Design;LBDD)と構造ベース(Structure Base Drug Design;SBDD)のものに大別されるとされてきた。LBDDは既知の薬と「似たような」化合物をさがすことで新規化合物を探索する一方で、SBDDは標的となるタンパクと化合物の結合度を推定することで新規化合物の探索を行う。LBDDは計算量的にも負荷が軽く、また、ヒット率が高いという利点を持つが、既存薬が無い疾患に関しては無力なことと、まさに「似たような」化合物しか見つからないため、化学的な構造を異にする化合物は、仮に有効なものでも探索できないという欠点があった。一方、SBDDにはこのような欠点が無い一方で、計算量的な負荷が某大であること、また、標的遺伝子のタンパクとしての立体構造が既知でなくてはならないこと(正確には既知でない場合は立体構造の予測から情報科学的に行わなくてはならないため、そこで確実性がさがってしまう)などの欠点もあった。
この様な欠点を補うため、モデル動物や、培養細胞に化合物を投与した場合の遺伝子発現プロファイルを指標にして類似化合物を探すという試みが提案されている。この方法は化学的構造を異にする新規化合物を探せる上に計算量的な負荷も軽いというLBDDとSBDDのいいとこ取りのような手法ではあるが、依然として既知の有効な薬が無い場合には無力であるという問題があった。この様な観点から、田口は既知化合物が無くても遺伝子発現プロファイルから新規化合物を探索できる方法を提案し、昨年、一昨年のこのアドベントカレンダーで報告してきた。しかし、化合物を投与した場合の遺伝子発現プロファイルを用いる、と書くとドラッグリポジショニングにしか使えないのではという揶揄のような意見を、特に国内の創薬専門家からうけることもままあった。
そのような中で今年(2019年)10月にキューバであったBioinfoMics2019というイベントに プレナリーレクチャラーとして招かれた際、4人いるプレナリーレクチャラーの僕以外の3人のうち二人までが、化合物を投与した場合の遺伝子発現プロファイルを視野に入れた大規模な化合物データベースを作ったという報告であることを知り、彼我の差に衝撃を受けた。そこで今年の創薬アドベントカレンダーでそのような試みを紹介し、少しでも創薬関係者に興味を持ってもらおうと考えた次第である。以下、代表的(?)な2つの化合物データベースについて紹介する。
ChemicalChecker
ChemicalCheckerと通常の化合物データベースとの違いは、ChemicalCheckerは化合物間の類似性に基づいた化合物データベースである、ということである。通常の化合物データベースというと化合物に対して多くの性質が列挙されている化合物×属性の表をイメージすると思うが、ChemicalCheckerのデータ構造はその様な物ではなく、化合物と化合物の類似度の表記になっている(下図は類似度の計算のついての模式図)。
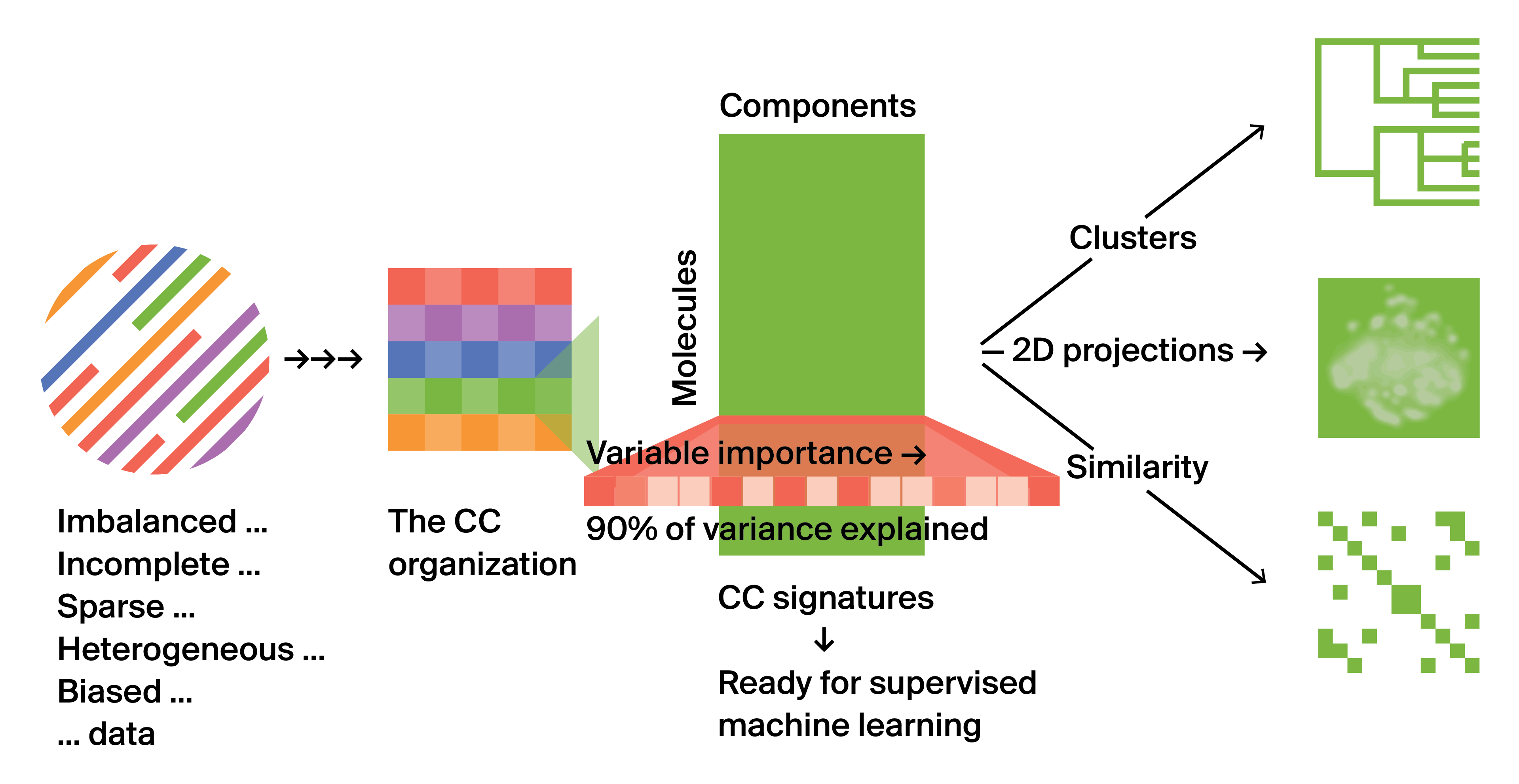
このため、表構造ではなく化合物×化合物×類似度(属性)という三階のテンソルの構造になっている。勿論、二次元平面であるディスプレイ上に三階のテンソルをそのまま表示することはできないので、化合物ごとに化合物×属性の表が表示される。ここではAcetaminophenを例にとって説明しよう。この画面では行に他の化合物、列に類似度を計算するのに用いた属性が並んでいる。属性は、Chemistry,Targets,Networks,Cells,Clinicsの5つに大きく分類されている。
Chemistry
Chemistryは、2D fingerprints, 3D fingerPrints, Scaffolds, Structural Keys, Physicohemistry,の5つから構成されている。Acetaminophenと行の化合物がこれらの5つのうちのどれかを考慮した類似度で類似性があるとみなされた場合にはオレンジの丸が描かれており、マウスオーバーすることで有意確率を知ることができる。これらはあくまで物質として化合物を記述する指標である。
Targets
Targetsはその名の通り、各化合物が標的とするタンパクの情報であり、同じタンパクを標的としていれば類似度が高い、とみなされる。TargetsはMechanisms of Action, Metabolic Genes, Crystals, Binding, HTS bioassaysの5つからなる。Mechanisms of Actionは標的に対する阻害剤か促進剤かで標的を分け、これが一致していれば類似度が高いとするものである。Metabolic Genesは薬剤代謝に関する遺伝子に対してどれくら共通の標的を共有しているか、Crystalは共結晶が取られてるタンパクの進化的な類似度(共通祖先タンパクをもっているか)、Bindingはアッセイ実験を元にした、実際の結合度評価(いわゆるK値)で考えた時、どれくらいの共通の標的を持っているか、HTS bioassaysはタンパクではなく機能的なバイオアッセイでの活性に基づいてどれくらい共通度があるか(なのでこの項目はタンパクに基づく指標ではない)という類似度評価である。
Networks
ここまでは或る意味、LBDDやSBDDで考慮されてきた性質だがこれ以降はやや様相が異なる。NetworksはSmall molecule roles, small molecule pathways, Signalling pathways, Biological Processes, Interactomeからなっている。Small molecule rolesは化合物の生物学的な機能、small molecule pathwaysはいわゆる代謝パスウェイにどれくらい共通して所属しているか、SIGNALING PATHWAYSはどれだけ共通に Signalling pathwaysに属しているか、BIOLOGICAL PROCESSESは標的としているタンパクが所属しているBIOLOGICAL PROCESSES(GO Term)の共通度、INTERACTOMESに至っては標的のタンパクのタンパクータンパク相互作用の共通度から計算した類似度である。一般に対象としている疾患以外の活性は共通していても意味がないと思われていると思うがこのデータベースではそういう立場をとらないのである。
Cells
Cellsは、GENE EXPRESSION,CANCER CELL LINES,CHEMICAL GENETICS,MORPHOLOGY, CELL BIOASSAYSの5つのカテゴリーからなっている。 GENE EXPRESSIONは各化合物を培養細胞に投与した時の発現プロファイルに基づく類似度、CANCER CELL LINESはやはり化合物を投与した場合の遺伝子発現プロファイルだが、癌細胞由来の培養細胞に限った場合、CHEMICAL GENETICSは酵母変異体に投与した場合の発現プロファイル、MORPHOLOGYは化合物投与後の細胞の画像情報、そして、CELL BIOASSAYSはその名の通り、化合物投与後の細胞活性である。いずれにせよ、細胞に化合物を投与した時の反応に基づく類似度である。
Clinics
Clinicsは、THERAPEUTIC AREAS, INDICATIONS, SIDE EFFECTS, DISEASE PHENOTYPES, DRUG-DRUG INTERACTIONSの5つのカテゴリーからなる。THERAPEUTIC AREASは、対象とする疾患の種類に基づく類似度、INDICATIONSは似たような意味だが適応症状、 SIDE EFFECTSは薬の副作用、 DISEASE PHENOTYPESは(疾患種類ではなく)症状別、DRUG-DRUG INTERACTIONSは薬物の相互作用に基づく類似度となっている。
まとめ
いずれにせよ、非常の広範な化合物に対する類似度を収集して創薬に役立てようとしていることがよく解る。計算された類似度の生データは公開されており、自由に研究に使うことができるのもおいしいだろう。詳しいことはプレプリントを参照されたい。
PhemacoDB
PhemacoDBもChemicalChekerと同じような問題意識で作られたデータベースで、化合物の構造や標的タンパクとのアフィニティを超えた広範な情報を化合物に対して収集することを目的とする。ただし、ChemicalCheckerとは異なり、主にDose dependenceに特化したデータ収集を行っており(下図は培養細胞MCF7のpaclitaxelという薬剤に対するdose dependenceの表示例である)、APIやRパッケージを提供することでよりユーザ側でのデータ利用を意識したつくりになっている。このビデオを見るのが全体像をつかむのには手っ取り早い。
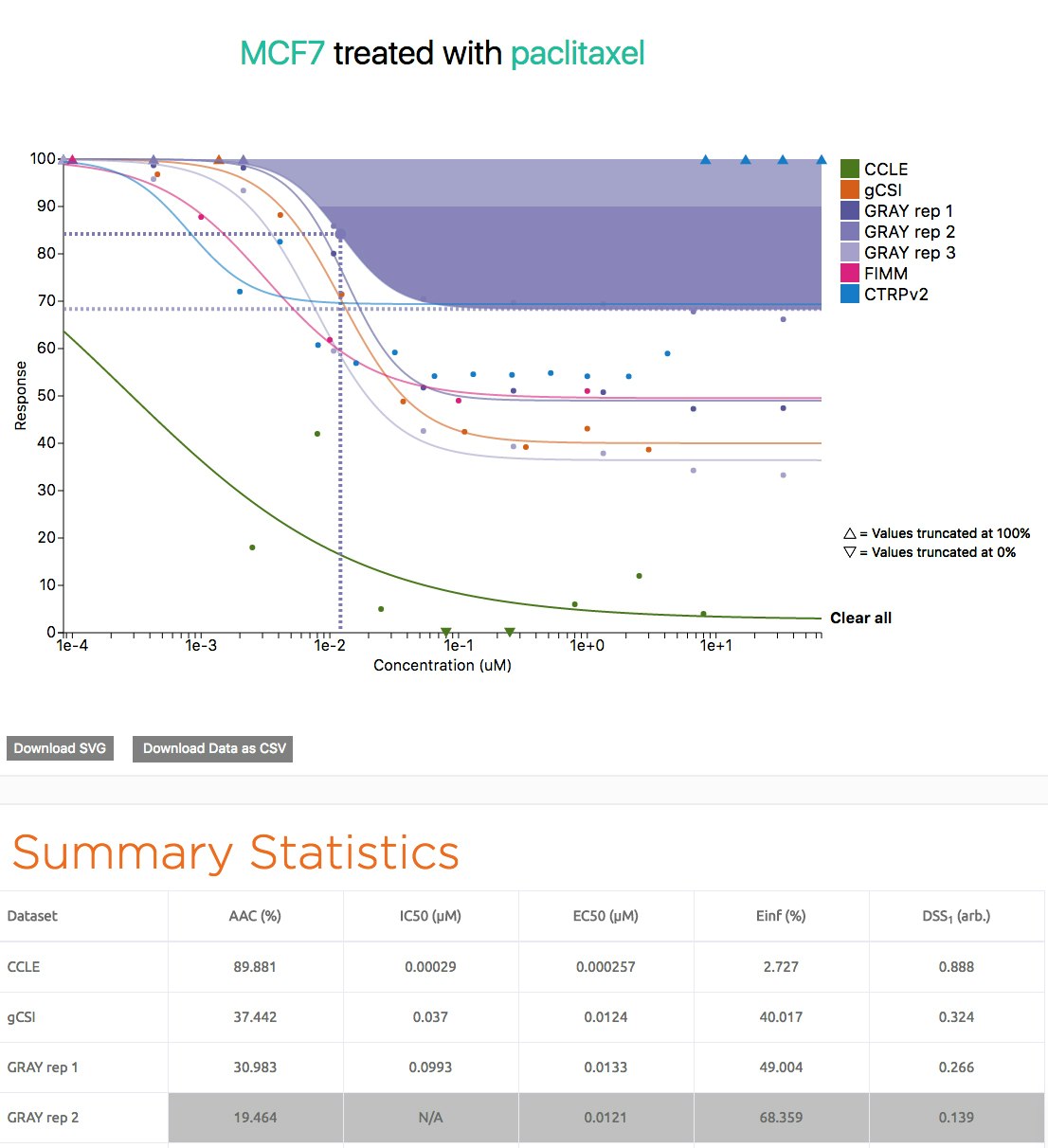
ビデオを視聴し終わったらEXPLOREをクリックしてみると全体像が分かりやすい。まず「臓器」と「遺伝子」を選んだあとNEXTをクリックするとその遺伝子を標的とする「培養細胞」と「薬」ペアが表記されるのでその中から組み合わせを選んでNEXTをクリックするとDose dependenceが表示される。
各化合物にはDose Dependenceの実験リストの他に既知の標的遺伝子の情報も併記されている。基本、Dose dependenceの実験を検索するというスタンスなので、実験が、培養細胞捌、臓器別、化合物別に検索出来て、表示ができるという使い勝手になっている。遺伝子からの検索も可能であるが、殆どの遺伝子は化合物の標的になってないので「その遺伝子を標的にしている化合物はありません」という答えが返ってくることが多い。詳しくは原著論文を参照されたい。
おわりに
インシリコ創薬において従来注目されていた化合物の構造情報や、標的タンパクとのアフィニティ以外の情報を広範に取得してデータベース化し、積極的に役立てようという欧米の動きについて紹介するために代表的な研究を2つ紹介した。日本でもインシリコ創薬は盛んだが、ここで上げたような化合物を培養細胞やモデル生物に投与した時の反応やゲノムとの関連を積極的にインシリコ創薬に用いようという動きはまだ少ないように思われる。この方向の研究が盛んになることを祈ってやまない。