RAD-seqやMIG-seq、GBSなどのゲノム縮約法による多型検出にほぼ必須のソフトとなったStacksの使い方をざっくり紹介します。
バージョン2以降、ペアエンドのデータにも対応しており、機能も豊富なので大変重宝するソフトです。
慣れれば非常に使いやすいです。
解説はMacOSの場合で進めていきますが、Linuxでも動かせます。
PCスペックの要求はそんなに高くなく、Core i5程度であれば普通に動きます。
インストール方法はこちらを参照ください。
環境
macOS 11.1
Stacks 2.54
間違えがあればコメント等でご指摘下さい!
Stacksとは
Stacksは、Illuminaシーケンサーで読まれたショートリード配列から遺伝子座を構築するためのソフトウェアパイプラインです。Stacksは、RAD-seqなどの制限酵素ベースのデータを用いて、遺伝地図を構築し、集団ゲノミクスや系統解析を行うために開発されました。
Stacksトップページより
Stacksで出来ること
- プーリングサンプルのインデックス配列ごと仕分け
- リードのクオリティコントロール(別のソフトでやった方が良い)
- リードを整列し、カタログ化する
- リファレンス配列へのマッピング、アライメント
- SNP検出と解析用ファイルの書き出し
- いくつか統計量の算出
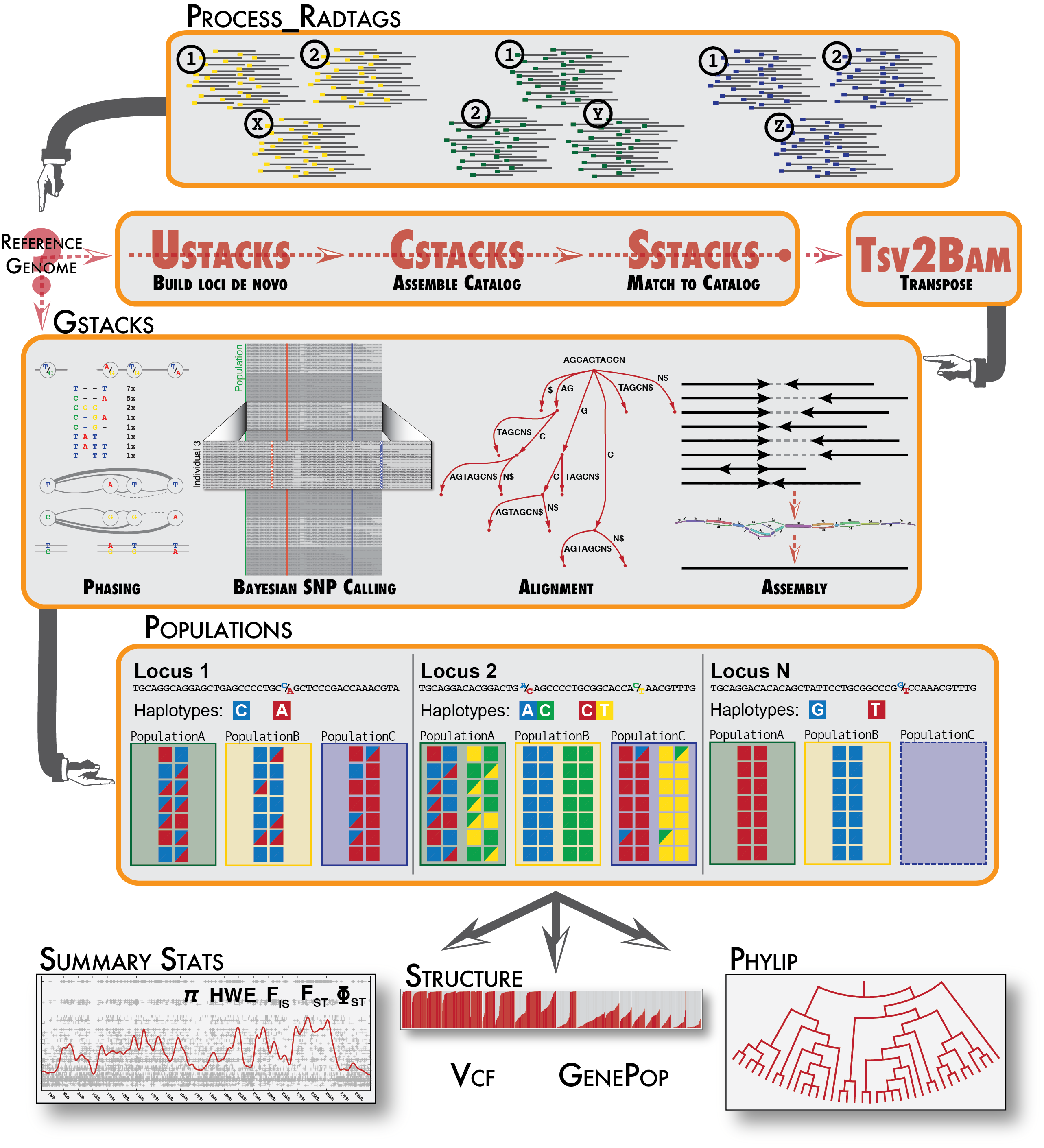
公式サイトより
データの準備
Stacksのすべてのプロセスでは、Fastqファイルをgz形式で圧縮したまま扱えます。
最初のプロセスは手持ちのファイルの状態によって変わります。
- fastq(.gz)ファイルは個体ごとに仕分けられている → クオリティコントロールへ
- fastq(.gz)ファイルはプールされた状態でRAD-seqのデータ → process_radtagsへ
- fastq(.gz)ファイルはプールされた状態でRAD以外のデータ → process_shortreadsへ
サンプルがプールされた状態から始める場合は、使用したインデックス配列とサンプル名を対応させるテキストファイルを用意しておきます。
AACCC Danio_rerio_Male_1
AAGGG Danio_rerio_Male_2
ACCAT Danio_rerio_Male_3
ACGTA Danio_rerio_Female_1
ACTGC Danio_rerio_Female_2
AGAGT Danio_rerio_Female_3
例えばこんな感じです。
process_radtagsの使い方
process_radtagsは制限酵素配列とインデックス配列を参照してリードをサンプルごとに振り分けます。
RAD-seqライブラリをHiSeq Xなどのイルミナシーケンサーで読んだデータがまるまるあるけど、デマルチプレックスをしていないデータを抱えている方はここから始めます。
クオリティコントロールも出来ますが、あまり高機能ではないのでそこは他のソフトですることを推奨します。
process_radtags -P -1 ./fPooled_sample_Read1.fq.gz -2 ./Pooled_sample_Read2.fq.gz
-o ./samples/ -b ./barcodes/ --inline_null -e sbfI -y gzfastq -i gzfastq
#ペアエンドの場合は-Pを指定
#-1と-2でペアエンドのインプットデータのディレクトリを指定
#-oで出力先のフォルダを指定
#-bでインデックス配列とサンプル名の書かれたファイルを指定
#-eで使用した制限酵素の種類を指定
#-yと-iでインプットおよびアウトプットのファイル拡張子を指定。ここではfastq.gz
クオリティコントロールに進みます。
process_shortreadsの使い方
process_shortreadsはインデックス配列を参照してリードをサンプルごとに振り分けます。
MIG-seqライブラリをNovaSeqなどのイルミナシーケンサーで読んだデータがまるまるあるけど、デマルチプレックスをしていないデータを抱えている方はここから始めます。
MiSeqでシーケンスした場合は既に振り分けが終わっているはずです。
クオリティコントロールも出来ますが、あまり高機能ではないのでそこは他のソフトですることを推奨します。
process_shortreads -1 ./rawdata/Pooled_sample_Read1.fq.gz -2 ./rawdata/Pooled_sample_Read2.fq.gz
-o ./samples/ -b ./barcodes.txt -y gzfastq -i gzfastq
#-1と-2でペアエンドのインプットデータのディレクトリを指定
#-oで出力先のフォルダを指定
#-bでインデックス配列とサンプル名の書かれたファイルを指定
#-yと-iでインプットおよびアウトプットのファイル拡張子を指定。ここではfastq.gz
クオリティコントロールに進みます。
クオリティコントロール
リードにはシーケンスエラーやアダプター配列が含まれている場合があります。
それらは解析のノイズになり、誤った結果を導く原因になるので最初に除去しておきます。
本ページでは省略しますが、fastp(おすすめ)、FastQC、Trimmomatic等で簡単に処理できます(いずれもHomebrewで導入可)。スクリプトを書いてループさせると楽です。
異なるリード長が混在しているとStacksがエラーを吐くので、長さは統一しておきます。
参照サイト
高速なfastqの前処理パイプライン fastp | macでインフォマティクス
FastQC | bioinformatics
Trimmomatic | bioinformatics
ペアエンドの場合、ファイル名は手直しが必要な場合があります。
以下のように、拡張子の前にリード1の場合はサンプル名.1、リード2の場合はサンプル名.2としておくと、
Stacksが勝手にリードをペアエンドとして認識してくれます。
$ ls ./samples
Danio_rerio_Male_1.1.fasta.gz
Danio_rerio_Male_1.2.fasta.gz
Danio_rerio_Male_2.1.fasta.gz
Danio_rerio_Male_2.2.fasta.gz
Danio_rerio_Male_3.1.fasta.gz
Danio_rerio_Male_3.2.fasta.gz
Danio_rerio_Female_1.1.fasta.gz
Danio_rerio_Female_1.2.fasta.gz
Danio_rerio_Female_2.1.fasta.gz
Danio_rerio_Female_2.2.fasta.gz
Danio_rerio_Female_3.1.fasta.gz
Danio_rerio_Female_3.2.fasta.gz
リードのスタック化から解析ファイルの出力まで
ちょっと拍子抜けですが、前準備が終われば、後はパイプラインを走らせるだけで終わります。
パイプラインは以下のStacksの6つのコアプログラムを自動で走らせてくれます。
ustacks → cstacks → sstacks → tsv2bam → gstacks → populations
このうち、ustacksとpopulationsではパラメータ設定によってリードのスタック化やSNP出力に関わるフィルタリングの強さを調整できます。
パイプラインで一通り走らせてから、populationsだけパラメーターを変えながら何度もやり直したりします。
popmapの準備
解析に使用するサンプルのリストをStacksに与えるために、popmap.txtを用意します。
同時に、サンプルのPop情報も付与できます。
Popmapファイルの中身は以下のような感じです。
サンプル名<タブ区切り>Pop情報
サンプル名<タブ区切り>Pop情報
サンプル名<タブ区切り>Pop情報
アウトプットファイルのサンプルの順番はPopmapの順になります。
ペアエンドモードの場合、サンプル名からリード番号以下をカットします。
シングルエンドの場合、サンプル名にはリード番号までを含めます。
ペアエンドの場合はこんな感じです。
Danio_rerio_Male_1 India
Danio_rerio_Male_2 Bangladesh
Danio_rerio_Male_3 Bangladesh
Danio_rerio_Female_1 India
Danio_rerio_Female_2 India
Danio_rerio_Female_3 Bangladesh
denovo_map.pl の使い方
基本的には、
- QC済みのファイルが格納されたフォルダ
- 書き出し先のフォルダ
- 各個体とそのPop情報が書かれたテキストファイル
の3つを指定するだけです。
denovo_map.plの実行は以下のようにします。
denovo_map.pl -M 5 -T 16 -o ./stacks --popmap ./popmap.txt --samples ./samples --paired
-X "ustacks:-m 5" -X "populations:--write-single-snp -R 0.7 --vcf --min-mac 2 --max-obs-het 0.75"
#-Mはustacksにおけるスタック間の最大塩基置換数の許容値
#-Tでスレッド数を指定
#-oで出力先のフォルダを指定
#--popmapでPopmapファイルのディレクトリを指定
#--sampleでインプットデータの場所を指定。Read1も2も同じフォルダに格納しておく
#--pairedはペアエンドモードで実行する場合に付ける
#-X "プログラム名: オプション"で各プログラムのオプションを設定できる
#ここではustacksでDepth数(-m)とpopulationsでフィルタリングと書き出し形式の設定をしている
サンプル数が多いと結構時間がかかります。
PCのスペックによりますが、MIG-seq程度のデータ量でも数時間〜半日かかります(192サンプル)。
人の作ったパイプラインを使うのは最高だなぁ。
ustacksの設定について
ustacksはStacksパイプラインの一番最初のプログラムです。
各個体において、読み込んだリードから同じ遺伝子座同士をまとめてくれます(スタック)。
このとき、同じ遺伝子座と判断する基準やその数に関する3つのパラメーター設定ができます。
denovo_map.plで勝手にやってくれますが、パラメーターは設定できるので解説します。
誤訳、誤解が怖いので原文も載せておきます。
-M — Maximum distance (in nucleotides) allowed between stacks (default 2).
-m — Minimum depth of coverage required to create a stack (default 3).
-N — Maximum distance allowed to align secondary reads to primary stacks (default: M + 2).
-
-Mは同じ遺伝子座のリードをまとめる際に、どのぐらいの塩基置換まで許容するかの値で、一般的には2〜5あたりがよく使われていると思われます。許容値を上げるほど最終的に得られる座数が減ります。 -
-mは各遺伝子座にリードが張り付いてスタックを形成しますが、その時の張り付いたリード数が一定値以下の遺伝子座を除外します。一般的には3〜30ぐらいがよく使われている気がします。より厚く設定した方が、エラーやコンタミ由来の変異を取り除けると思われますが、下限値を上げるほど最終的に得られる座数が減ります。 -
-NはM+2の値がデフォルト設定となっており、それでいいと思います(脳死)。
処理が終わると、ログに各個体の平均Depthが表示されます。
Danio_rerio_Male_1: 31.20x
Danio_rerio_Male_2: 37.56x
Danio_rerio_Male_3: 36.57x
Danio_rerio_Female_1: 37.15x
Danio_rerio_Female_2: 28.59x
Danio_rerio_Female_3: 37.42x
populationsの設定について
populationsはStacksパイプラインの一番最後のプログラムです。
カタログ化された遺伝子座やSNPを一定の基準に基づいて、解析用ファイルとして出力してくれます。
denovo_map.plで勝手にやってくれますが、ここだけ何度もやり直すと思われるので解説します。
populationsでは一部の出力形式だと集団ごとに個体がマージされる場合があります(例えば、Phylip形式)。その場合は1列目も2列目もサンプルIDを書いたpopmapを使います。
フィルタリングの設定
誤訳、誤解が怖いので原文も載せておきます。
#変異サイトを持つ遺伝子座の集団や個体間における共有率でのフィルタリング
-p,--min-populations [int] — minimum number of populations a locus must be present in to process a locus.
-r,--min-samples-per-pop [float] — minimum percentage of individuals in a population required to process a locus for that population.
-R,--min-samples-overall [float] — minimum percentage of individuals across populations required to process a locus.
-H,--filter-haplotype-wise — apply the above filters haplotype wise (unshared SNPs will be pruned to reduce haplotype-wise missing data).
#マイナーアリルの頻度や数、ヘテロ接合度によるフィルタリング
--min-maf [float] — specify a minimum minor allele frequency required to process a nucleotide site at a locus (0 < min_maf < 0.5).
--min-mac [int] — specify a minimum minor allele count required to process a SNP.
--max-obs-het [float] — specify a maximum observed heterozygosity required to process a nucleotide site at a locus.
#出力するSNPを各遺伝子座につき一つにする場合の設定
--write-single-snp — restrict data analysis to only the first SNP per locus.
--write-random-snp — restrict data analysis to one random SNP per locus.
#出力する遺伝子座の指定と除外
-B,--blacklist — path to a file containing Blacklisted markers to be excluded from the export.
-W,--whitelist — path to a file containing Whitelisted markers to include in the export.
よく使うものだけ解説します。
-
-Rは変異サイトのある遺伝子座およびSNPが全サンプル内でどれだけ共有されているかによってフィルターをかけます。例えば、0.5とすると、その遺伝子座が対象のサンプル内の50%以上の個体が持っていない場合、除外されます。-rは各集団内における割合、-pはSNPを共有する集団数でフィルターします。 -
--min-macは全サンプル内で同じ変異を持つ個体の数に下限を設け、それ以下の変異(マイナーアリル)を除外します。--min-mafはマイナーアリルの頻度でフィルターします。 -
--write-single-snpはひとつの遺伝子座に変異サイトが複数乗っている場合に、1番目のSNPのみを出力するオプションです。同じ遺伝子座に乗っている変異は互いに連鎖していると考えられるので、その影響を排除したい場合に指定します。--write-random-snpは遺伝子座から1つのSNPを選ぶ時に、1番目ではなくランダムに選びます。
出力ファイルの種類
よりどりみどりですが、結構手直しが必要になったりします。
--ordered-export — if data is reference aligned, exports will be ordered; only a single representative of each overlapping site.
--fasta-loci — output locus consensus sequences in FASTA format..
--fasta-samples — output the sequences of the two haplotypes of each (diploid) sample, for each locus, in FASTA format.
--vcf — output SNPs and haplotypes in Variant Call Format (VCF).
--genepop — output results in GenePop format.
--structure — output results in Structure format.
--radpainter — output results in fineRADstructure/RADpainter format.
--plink — output genotypes in PLINK format.
--hzar — output genotypes in Hybrid Zone Analysis using R (HZAR) format.
--phylip — output nucleotides that are fixed-within, and variant among populations in Phylip format for phylogenetic tree construction.
--phylip-var — include variable sites in the phylip output encoded using IUPAC notation.
--phylip-var-all — include all sequence as well as variable sites in the phylip output encoded using IUPAC notation.
--treemix — output SNPs in a format useable for the TreeMix program (Pickrell and Pritchard).
--no-hap-exports — omit haplotype outputs.
--fasta-samples-raw — output all haplotypes observed in each sample, for each locus, in FASTA format.
よく使うものだけ解説します。
-
--vcfはとりあえず書き出しておくのがいいです。なんにでも使えますが、vcftoolsやDsuiteなどで集団遺伝学的パラメーターや各種統計量の算出によく使います。 -
--genepopもとりあえず書き出しておく形式です。GenoDiveのようなソフトでそのまま解析できます。 -
--structureはSTRUCTUREやintrogressでの解析に使えます。 -
--plinkはPLINKでの解析に使えます。ADMIXTUREで解析する際はPLINKを通してインプットファイルを作ります。 -
--phylip-varはフィルタリングを通過した遺伝子座のSNPだけを繋げたPhylipファイルを出力します。RAxML-NGやIQ-TREE、Splitstreeなどでの系統解析に便利です(解析の前にファイルの最後のコメントアウトを消す必要があります)。 -
--phylip-var-allはフィルタリングを通過した遺伝子座の全長配列を繋げたPhylipファイルを出力します。パーティションファイルも一緒に吐き出します。
座数やミッシング(欠損値)の具合を確認しながら、パラメーターを調整したりサンプルの選定をします。
後は解析するだけだ・・・!
トラブル
解析が回らない
うまくいかない時はたいていディレクトリの指定が間違っているか、データ量のないサンプルが混ざっているか、実際のサンプル名とpopmapに書いてある名前の不一致があるかだと思います。
解析が途中で止まる
1回だけ、一度に解析するサンプル数を増やした時(250〜)、UNIXのエラーによってgstacksで止まったことがありました。
ログを見ると以下のようになっていました。
gstacks
==========
/usr/local/bin/gstacks -P ./stacks -t 16 -M ./popmap.txt
[E::hts_open_format] Failed to open file ./stacks/sample-name.matches.bam
Error: Failed to open BAM file './stacks/sample-name.matches.bam'.
Error: You might need to increase your system's max-open-files limit, see https://groups.google.com/d/msg/stacks-users/GZqJM_WkMhI/m9Hreg4oBgAJ
Aborted.
denovo_map.pl: Aborted because the last command failed (1).
これはユーザーが一度に開けるファイル数の上限に達してしまったときに生じるエラーです。
上限値の設定をulmitコマンドで確認してみます。
$ ulimit -a
core file size (blocks, -c) 0
data seg size (kbytes, -d) unlimited
file size (blocks, -f) unlimited
max locked memory (kbytes, -l) unlimited
max memory size (kbytes, -m) unlimited
open files (-n) 256
pipe size (512 bytes, -p) 1
stack size (kbytes, -s) 8192
cpu time (seconds, -t) unlimited
max user processes (-u) 8352
virtual memory (kbytes, -v) unlimited
一度に開けるファイル数(open files)が256になっており、Stacksはこれを超えてしまったようです。
(止まったサンプルは254番目でした)
ここでは上限値を4倍に設定します。
$ ulimit -n 1024
これで動きました。ヨシ!
マルチスレッドで動いていないとき
Stacksでスレッド数指定のオプションを入れても、マルチスレッドで動いていないケースが時々あります。
大抵はOpenMP関係のトラブルが原因だと思われます。
インストール時にOpenMPに対応しているgccとg++を指定してあげる必要があります。
以下を参照してください。
引用文献
Rochette, N., A. Rivera‐Colón, and J. Catchen. 2019. Stacks 2: Analytical methods for paired‐end sequencing improve RADseq‐based population genomics. Molecular Ecology 28: 4737-4754. https://doi.org/10.1111/mec.15253