お前だれ
都内?某T大学部4年、機械学習・統計手法でバイオインフォマ、ケモインフォマでケモケモしたりしようとしているのドメイン知識まだまだのRic.です。
今回やったこと
すでに釈迦に説法な内容かもしれないが.txtファイルから構造データ(SMILES)を読み込み、化合物間の類似度をRDKitによって計算、一定の水位以上の類似度でフィルタリングを行い.csvファイルに書き出しを行った。
(これは今回自分が卒論において学習データをフィルタリングする用に備忘録として書いています。)
何故やるのか
- SDFやMOLファイルでは無くテキストファイルでデータを渡される事もままある(自分だけ?)
- データベースの類似度フィルタリングは大体Tanimoto係数(DiceとFraggleも使いたい)
(東京化成が採用しているのはTanimoto係数だそう。)
データを学習にかけたりする際にcsvの方が便利であろうという事で軽く作って見ました。
どうやるのか
今回採用したfingerprintの種別は,MACCSkeys
先述しましたが比較手法として以下の三つ
- Fraggle係数
- Tanimoto係数
- Dice係数
今回はRDKitとPythonでどう実装するかがメインとして書くので、それぞれ詳細に知りたい場合は
このリンク
Fraggleについて
Tanimotoについて
Diceについて
を参照すると良いかと思います。(お世話になりました)
何をやったのか
HIVに活性であった化合物(1000前後)とGefitinibとの類似性を以上の三つの類似度指標で検証した。
実際のコードはこちら、急いで書いたので拙い部分が多いですがご容赦ください。
from rdkit import rdBase, Chem, DataStructs
from rdkit.Chem import AllChem, Draw, rdMolDescriptors
from rdkit.Chem.Fraggle import FraggleSim
import pandas as pd
from rdkit.Chem.Draw import IPythonConsole
from rdkit.Chem.Fingerprints import FingerprintMols
from rdkit.Avalon import pyAvalonTools
from rdkit.Chem.AtomPairs import Pairs, Torsions
from rdkit.Chem import MACCSkeys
df1 = pd.DataFrame({'Compound': [],
'Similarity': [],
},
)
df2 = pd.DataFrame({'Compound': [],
'Similarity': [],
},
)
df3 = pd.DataFrame({'Compound': [],
'Similarity': [],
},
)
# 各種データフレームの用意
mol1 = Chem.MolFromSmiles('CCc(c1)ccc2[n+]1ccc3c2Nc4c3cccc4')
# 比較したい化合物のSMILESを入力
fp1 = MACCSkeys.GenMACCSKeys(mol1)
# fingerprintの生成
f = open('HIV_smiles_property_1.txt')
# ここで比較対象にしたい化合物群のファイルを読み込み
lines = f.readlines()
f.close()
i = 0
j = 0
n = 0
for line in lines:
mol2 = Chem.MolFromSmiles(line)
fp2 = MACCSkeys.GenMACCSKeys(mol2)
Fraggle = FraggleSim.GetFraggleSimilarity(mol1, mol2, tverskyThresh=0.8)
Tanimoto = DataStructs.TanimotoSimilarity(fp1, fp2)
Dice = DataStructs.DiceSimilarity(fp1, fp2)
if Fraggle[0] > 0.8:
print("Fraggle: "+line+":"+str(Fraggle[0]))
s = pd.Series([str(line), str(Fraggle[0])], index=df1.columns, name=str(i))
df1 = df1.append(s)
i += 1
if Tanimoto > 0.6:
print("Tanimoto: "+line+":"+str(Tanimoto))
s = pd.Series([str(line), str(Tanimoto)], index=df2.columns, name=str(j))
df2 = df2.append(s)
j += 1
if Dice > 0.7:
print("Dice: "+line+":"+str(Dice))
s = pd.Series([str(line), str(Dice)], index=df3.columns, name=str(n))
df3 = df3.append(s)
n += 1
print(df1)
print(df2)
print(df3)
df1.to_csv("Fraggle.csv")
df2.to_csv("Tanimoto.csv")
df3.to_csv("Dice.csv")
print('done!')
Fraggle,Tanimoto,Diceの三つのデータフレームを作りある一定の水位以上の類似を持つ者はcsvファイルへ書き込む
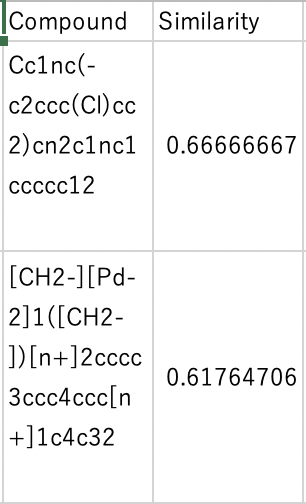 Tanimoto
Tanimoto
 Dice
Dice
終わりに
今回は類似度の計算の仕方にフォーカスしましたがfingerprintにも種類があり、それによっても類似度は変動するためその辺りも検証してみようと思う。
また深層学習の実装の話まで持っていくつもりだったが実装が間に合わなかったので概要だけ触れておくと
dcGAN(deep convolutional GAN)が畳み込みのネットワークで学習精度を保ちつつ生成を行ったのと同様、GNNをGANに噛ませる事で化合物を精度を保ちつつ活性しそう・しなさそうな、それっぽい構造を持つ化合物を生成するモデルを考えている。
名付けてdgGAN?
お楽しみにしてください。
