要点
- Google Cloud Platform演習の備忘録。
- オープンデータを使ってデータ取り込み・前処理・可視化までをやってみました。
- 長くなるので3部構成です。今回はデータ取り込みまで記載します。
目的
- BigQueryとデータポータルを用いたデータ整理〜可視化の練習
- ついでに、オープンデータを使ったデータ前処理も実施
全体の流れ
- データの取り込み
- 前処理
- 処理データを格納 👈今回はここまで
- BigQueryへのデータ読み込み
- 可視化と考察
データをPythonで前処理し、BigQueryに読み込ませてデータポータルでの可視化を行います。
BigQuery上でSQLによる前処理も行えるのですが、今回はPythonを使いました。使用するオープンデータを探していたとき、BigQueryで直接読み込めないExcelファイルを多く見かけたためです (今回のデータはcsvでしたが)。
BigQueryに取り込み後の整形や計算はSQLで処理することにしました。
前処理後のデータはGoogle Cloud Storage (GCS) に格納してからBigQueryに読み込ませました。BigQueryにはローカルやGoogleドライブから直接ファイルをアップロードできるのですが、今回は練習のためにGCSを使っています。
メソッド
データ
-
厚労省: 新型コロナウイルス感染症について > オープンデータ
(2021/1/12時点)
ツール
- Google ドライブ (以下gドライブ)
- Colaboratory (以下colab)
- Google Cloud Platform
- Google Cloud Storage (以下GCS)
- BigQuery
- データポータル
環境
- 端末: iPad Pro 第3世代
- ブラウザ: Google Chrome Ver 87.0.4280.77
実施手順
データの読み込み
データをgドライブに格納
gドライブに10個のファイルをアップロードしました。
今回はドライブ直下に「covid」フォルダを作成してファイルを格納しています。
colabをgドライブに接続
colab上でノートを新規作成します (ハンバーガーアイコン¥ファイル¥ノートブックを新規作成)
モジュールのインポート
ファイルの入出力のためにosを、データをデータフレームとして操作するためにpandasをインポートしました。
import os
import pandas as pd
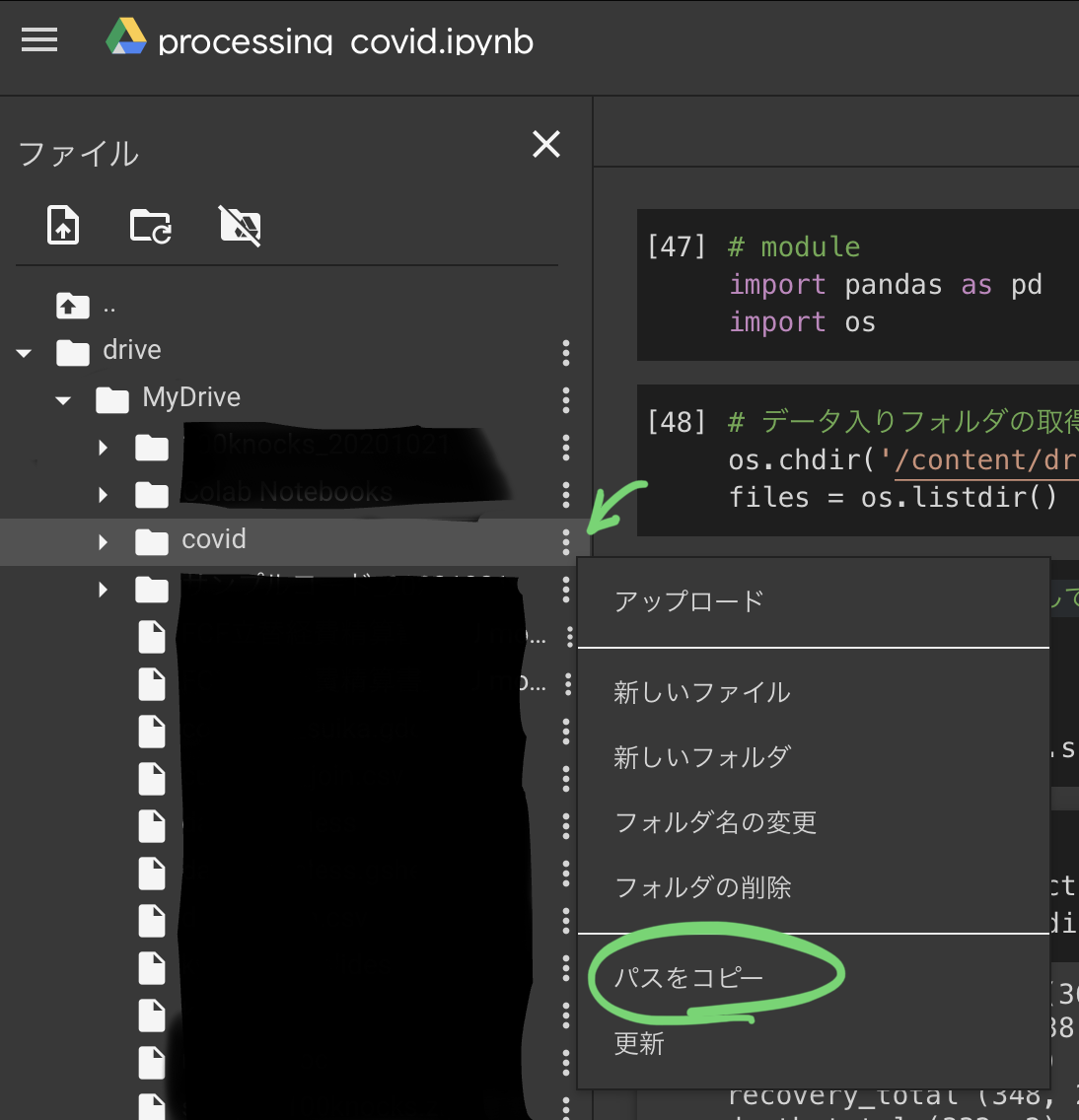
gドライブへのマウント
ファイルを読み込むため、カレントディレクトリにデータを格納したcovidフォルダを指定します。
os.chdir('/content/drive/MyDrive/covid') # コピーしたパスを貼り付ける
files = os.listdir() # データをdict型に取り込む準備
ファイルをまとめて処理することを想定し、データをdict型に格納します。
keyはファイル名、valueはファイル内のdataframeとします。
# 各データを格納するdict型を生成
data_dict = {}
for file in files:
data_dict[os.file.splitext(file)[0]] = pd.read_csv(file)
os.file.splitext()はファイル名の拡張子以外の部分と拡張子をtupple型で返します。
os.file.splitext(file)[0]はfileの拡張子以外の部分を取得していることになります。
これで、data_dictにフォルダ内の全てのファイルについて名前とデータが格納されました。
ここで各データの行列数を確認します。
for data in data_dict:
print(data, data_dict[data].shape)
pcr_positive_daily (361, 2)
pcr_tested_daily (338, 2)
cases_total (342, 2)
recovery_total (348, 2)
death_total (332, 2)
pcr_case_daily (327, 7)
current_situation (4, 8)
employment_subsidy_week (37, 7)
life_welfare_small_fund (3, 7)
severe_daily (341, 2)
300個台の行からなる7ファイルと、より少ない行数からなる3ファイルがあるようです。
ファイルの説明を読むと、前者の7ファイルは「年月日」と「ファイル名が示す特徴量」からなるようです。
たとえばpcr_positive_dailyの中身を見てみると...
data_dict['pcr_positive_daily'].head()
| 日付 | PCR 検査陽性者数(単日) | |
|---|---|---|
| 0 | 2020/1/16 | 1 |
| 1 | 2020/1/17 | 0 |
| 2 | 2020/1/18 | 0 |
| 3 | 2020/1/19 | 0 |
| 4 | 2020/1/20 | 0 |
多くのデータが年月日ごとの時系列データであるため、これらを年月日をキーにして結合したテーブルを作っておけば、観察できる幅が広がりそうです。
時系列データのなかで最も行数が少ない (期間が短い) のはpcr_case_dailyのようなので、このファイルに記載された年月日の範囲内で他の6データを結合していきます。
前処理
データの結合
pandasでデータフレームを横方向に結合する関数にはmergeとconcatがあり、それぞれ結合できるデータフレームの条件が異なるようです。
| 3個以上のデータフレーム | 行数の異なるデータフレーム | |
|---|---|---|
| merge | × | ○ |
| concat | ○ | × |
今回は結合したいデータの列数が異なるので、concatの使用は難しそうです。
forループにmergeを組み込んで、時系列ファイルを結合することにしました。
# 時系列データでサイズが最も小さいpcr_case_dailyを主データとしてleft_joinしていく
df = data_dict['pcr_case_daily']
# 行数がpcr_case_dailyより多いファイルだけ結合する
for data in data_dict:
if len(data_dict[data]) > len(data_dict['pcr_case_daily']:
df = pd.merge(df, data_dict[data], on='日付')
print(df.shape)
df.head()
(324, 13)
| 日付 | 国立感染症研究所 | 検疫所 | 地方衛生研究所・保健所 | 民間検査会社 | 大学等 | 医療機関 | PCR 検査陽性者数(単日) | PCR 検査実施件数(単日) | 入院治療を要する者 | 退院、療養解除となった者 | 死亡者数 | 重症者数 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 2020/2/18 | 472 | 75 | 398 | 0.0 | 79.0 | NaN | 7 | 9 | 31 | 14 | 1 | 6 |
| 1 | 2020/2/19 | 15 | 68 | 609 | 0.0 | 0.0 | NaN | 10 | 71 | 35 | 16 | 1 | 7 |
| 2 | 2020/2/20 | 20 | 15 | 758 | 0.0 | 0.0 | NaN | 9 | 90 | 52 | 16 | 1 | 9 |
| 3 | 2020/2/21 | 261 | 188 | 902 | 132.0 | 108.0 | NaN | 11 | 85 | 62 | 16 | 1 | 10 |
| 4 | 2020/2/22 | 341 | 127 | 677 | 2.0 | 19.0 | NaN | 27 | 96 | 96 | 17 | 1 | 11 |
df.tail()
| 日付 | 国立感染症研究所 | 検疫所 | 地方衛生研究所・保健所 | 民間検査会社 | 大学等 | 医療機関 | PCR 検査陽性者数(単日) | PCR 検査実施件数(単日) | 入院治療を要する者 | 退院、療養解除となった者 | 死亡者数 | 重症者数 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 319 | 2021/1/5 | 0 | 0 | 5831 | 34704 | 4726 | 16521 | 4885 | 88446 | 40908 | 205212 | 3718 | 784 |
| 320 | 2021/1/6 | 0 | 0 | 7333 | 31352 | 4701 | 14939 | 5946 | 73967 | 43423 | 208621 | 3790 | 796 |
| 321 | 2021/1/7 | 0 | 0 | 7539 | 29584 | 3157 | 11848 | 7537 | 81440 | 46780 | 211900 | 3856 | 826 |
| 322 | 2021/1/8 | 0 | 0 | 5619 | NaN | NaN | NaN | 7844 | 63373 | 51125 | 215527 | 3931 | 827 |
| 323 | 2021/1/9 | 0 | 0 | 3570 | NaN | NaN | NaN | 7278 | 35730 | 55238 | 218676 | 3995 | 852 |
特徴数は日付を含んだ13個。結合したい列数と一致しています。
問題なくデータの結合が行えたように見えます。
欠損値の対応
データを見ると、"国立感染症研究所"~"医療機関"の列はpcr_case_daily由来で、施設ごとのPCR検査数を表しているようです。このファイル由来の列にNaNが入っているのが見て取れました。
そこで、各特徴量について欠損値がないかチェックしてみます。
df.isnull().sum()
日付 0
国立感染症研究所 0
検疫所 0
地方衛生研究所・保健所 0
民間検査会社 2
大学等 2
医療機関 19
PCR 検査陽性者数(単日) 0
PCR 検査実施件数(単日) 0
入院治療を要する者 0
退院、療養解除となった者 0
死亡者数 0
重症者数 0
dtype: int64
"民間検査会社""大学等"に2個、"医療機関"に19個の欠損値がありました。
df.tail()の結果から判るように、前者の欠損値は最後の2行にあるようです。
ファイルのダウンロード時点では集計が終わっていなかったものと思われます。
おそらく、最終2行のデータは集計が完了していなかったのでしょう。
今回は行ごと除くことにしました。
一方、後者の欠損値は最終2行と最初の17行に見つかりました。
(df.head(30)のようなコマンドで確認しています)
こちらは集計日が古いことから、単に集計していなかったものと思われます。
最終2行と同様に除いても良いのですが、こちらは0で置換することにしました。
実際の処理に移ります。
まずは最終2行の除去から。
df_dropped = df.dropna(subset=['民間検査会社']) # "民間検査会社"の値が欠損している行をdropnaで除く
df_dropped.tail()
||日付|国立感染症研究所|検疫所|地方衛生研究所・保健所|民間検査会社|大学等|医療機関|PCR 検査陽性者数(単日)|PCR 検査実施件数(単日)|入院治療を要する者|退院、療養解除となった者|死亡者数|重症者数|
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|317|2021/1/3|0|0|3592|7940|1790|8149|3127|20291|38729|198874|3598|731|
|318|2021/1/4|0|0|4433|16740|5624|19251|3302|84338|39905|201606|3654|771|
|319|2021/1/5|0|0|5831|34704|4726|16521|4885|88446|40908|205212|3718|784|
|320|2021/1/6|0|0|7333|31352|4701|14939|5946|73967|43423|208621|3790|796|
|321|2021/1/7|0|0|7539|29584|3157|11848|7537|81440|46780|211900|3856|826|
無事削除できました。
次に、残った欠損値を0で置換します。
df_dropped = df_dropped.fillna(0)
df_dropped.head()
||日付|国立感染症研究所|検疫所|地方衛生研究所・保健所|民間検査会社|大学等|医療機関|PCR 検査陽性者数(単日)|PCR 検査実施件数(単日)|入院治療を要する者|退院、療養解除となった者|死亡者数|重症者数|
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|0|2020/2/18|472|75|398|0|79|0|7|9|31|14|1|6|
|1|2020/2/19|15|68|609|0|0|0|10|71|35|16|1|7|
|2|2020/2/20|20|15|758|0|0|0|9|90|52|16|1|9|
|3|2020/2/21|261|188|902|132|108|0|11|85|62|16|1|10|
|4|2020/2/22|341|127|677|2|19|0|27|96|96|17|1|11|
念のため、改めて欠損値チェックを行います。
df_dropped.isnull().sum()
日付 0
国立感染症研究所 0
検疫所 0
地方衛生研究所・保健所 0
民間検査会社 0
大学等 0
医療機関 0
PCR 検査陽性者数(単日) 0
PCR 検査実施件数(単日) 0
入院治療を要する者 0
退院、療養解除となった者 0
死亡者数 0
重症者数 0
dtype: int64
すべての欠損値について対応を完了しました。
(任意) 列順の変更
必須の行程ではありませんが、自分の好みに列の順番を変更してみました。
未検証ですが、ここをスキップしても次の行程以降でエラーは出ないはずです。
# 列の順番をカスタマイズ
df_dropped = df_dropped[['日付',
'PCR 検査実施件数(単日)',
'PCR 検査陽性者数(単日)',
'入院治療を要する者',
'重症者数',
'死亡者数',
'退院、療養解除となった者',
'国立感染症研究所',
'検疫所',
'地方衛生研究所・保健所',
'民間検査会社',
'大学等',
'医療機関'
]]
df_dropped.head()
||日付|PCR 検査実施件数(単日)|PCR 検査陽性者数(単日)|入院治療を要する者|重症者数|死亡者数|退院、療養解除となった者|国立感染症研究所|検疫所|地方衛生研究所・保健所|民間検査会社|大学等|医療機関|
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|0|2020/2/18|9|7|31|6|1|14|472|75|398|0|79|0|
|1|2020/2/19|71|10|35|7|1|16|15|68|609|0|0|0|
|2|2020/2/20|90|9|52|9|1|16|20|15|758|0|0|0|
|3|2020/2/21|85|11|62|10|1|16|261|188|902|132|108|0|
|4|2020/2/22|96|27|96|11|1|17|341|127|677|2|19|0|
BigQuery読み込み用ファイルの作成
最後に、列名を英語に変換します。
BigQueryでは日本語を正常に読み込めないためです。
# key = 日本語の列名, value = 英語の列名 のdict型を作る
name_column = {
'日付': 'Date',
'国立感染症研究所': 'Inspect_NIID',
'検疫所': 'Inspect_QS',
'地方衛生研究所・保健所': 'Inspect_LIH_HC',
'民間検査会社': 'Inspect_Company',
'大学等': 'Inspect_Univ',
'医療機関': 'Inspect_MI',
'PCR 検査陽性者数(単日)': 'Positive',
'PCR 検査実施件数(単日)': 'PCR',
'入院治療を要する者': 'Treated',
'退院、療養解除となった者': 'Recovered',
'死亡者数': 'Death',
'重症者数': 'Severe'
}
df_new = df_dropped.rename(columns=name_column)
df_new.head()
||Date|PCR|Positive|Treated|Severe|Death|Recovered|Inspect_NIID|Inspect_Quarantine|Inspect_LHRI_HealthCenter|Inspect_Company|Inspect_Univ|Inspect_Medical|
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|0|2020/2/18|9|7|31|6|1|14|472|75|398|0|79|0|
|1|2020/2/19|71|10|35|7|1|16|15|68|609|0|0|0|
|2|2020/2/20|90|9|52|9|1|16|20|15|758|0|0|0|
|3|2020/2/21|85|11|62|10|1|16|261|188|902|132|108|0|
|4|2020/2/22|96|27|96|11|1|17|341|127|677|2|19|0|
(英語表記がおかしかったらごめんなさい...)
データの保存
作成したデータフレームは、"covid.csv"としてカレントディレクトリ (gドライブのcovidフォルダ)に保存します。
df_new.to_csv('covid.csv', index=False)
これでBigQueryに取り込める処理済みのデータができました。
次はこのデータをGCSを介してBigQueryに取り込み、データの可視化・考察を行っていきます。