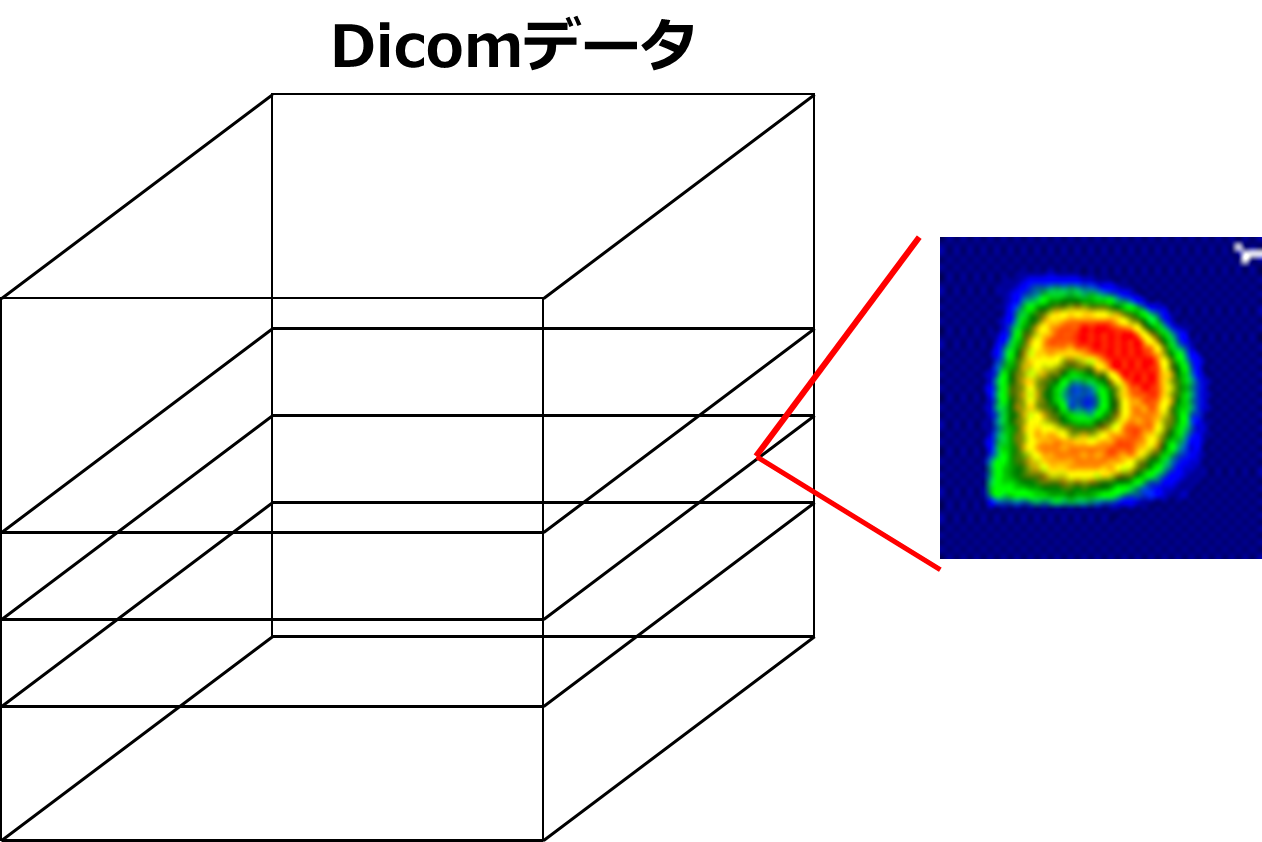
タイトルの通りですね。心臓の輪切りデータから、その中心を求めて可視化したいです。
ただ、公開できそうなdicomデータの持ち合わせがないので、dicomのいじるところはコード上でのみ解説、
その後の動作はこちらの画像データを一部利用させていただいてます。
Dicom画像の読み込み
そもそもdicomデータがどんな構造になってるかですが、上記画像のように複数枚の画像が層のように重なってるイメージでいいかなと思います。
配列で表すと3次元配列の構造になっていることになります。
一つ一つの画像に対してアプローチしたい場合は、まず何層目のデータを利用したいかを指定してあげることで、その層の画像をいじることができます。
それでは読み込んでみましょう。今回は37層目のデータを触りたいので固定で37と入れています。
path = './data/test_dicom.dcm'
dicom_data=pydicom.read_file(path)
dicom_list_37 = dicom_data.pixel_array[37]
データの前処理
続いてデータの前処理を行います。
今回は心臓と思われる点群のみ抽出し、だいたいの中心を求めたいです。なのでノイズとなるデータを消去したいです。
今回はそもそも0のデータを削除し、その後閾値を決めてそれ以上のデータのみ抽出していきます。
閾値にはパーセンタイルを用い、この数値は運用時にパラメータとして適宜変更したいと考えています。
今回は97%にしています。
最後にmedian_filterをかけてノイズ除去を行います。
これに利用するカーネルサイズもパラメータとして適宜変更するものとします。
percentile_threshold = 95
k_siz = 5
l_dicom=dicom_data.pixel_array[37][dicom_data.pixel_array[37]>0]
threshold=np.percentile(l_dicom, percentile_threshold )
x_list = []
y_list = []
for y in range(len(dicom_list_37)):
for x in range(len(dicom_list_37[y])):
if dicom_list_37[y][x] < threshold:
dicom_list_37[y][x] = 0
dicom_list_37=median_filter(dicom_list_37,k_siz)
def median_filter(src, ksize):
d = int((ksize-1)/2)
h, w = src.shape[0], src.shape[1]
dst = src.copy()
for y in range(d, h - d):
for x in range(d, w - d):
# 近傍にある画素値の中央値を出力画像の画素値に設定
dst[y][x] = np.median(src[y-d:y+d+1, x-d:x+d+1])
return dst
また、今回のテストに使う画像ではRGBをグレースケールに変更後に上記処理をかけています。
点群の取得
前処理ができたので、基準値以上の値を持つデータの座標をxとyごとにlistに保存していきたいと思います。
x_list = []
y_list = []
for yi, y in enumerate(dicom_list_37):
for xi, x in enumerate(y):
if x != 0:
y_list.append(yi)
x_list.append(xi)
点群の中心座標、半径の取得
本題の部分ですが、最小二乗法で円をカーブフィッティングという方法があるらしく、こちらを利用させていただきます。
def circle_fitting(x, y):
sumx = sum(x)
sumy = sum(y)
sumx2 = sum([ix ** 2 for ix in x])
sumy2 = sum([iy ** 2 for iy in y])
sumxy = sum([ix * iy for (ix,iy) in zip(x,y)])
M = np.array([[sumx2,sumxy,sumx],
[sumxy,sumy2,sumy],
[sumx,sumy,len(x)]])
Y = np.array([[-sum([ix ** 3 + ix*iy **2 for (ix,iy) in zip(x,y)])],
[-sum([ix ** 2 *iy + iy **3 for (ix,iy) in zip(x,y)])],
[-sum([ix ** 2 + iy **2 for (ix,iy) in zip(x,y)])]])
M_inv = np.linalg.inv(M)
X = np.dot(M_inv, Y)
a = - X[0] / 2
b = - X[1] / 2
r = np.sqrt((a ** 2) + (b ** 2) - X[2])
return a, b, r
画像内に円を描画して可視化
最後に中心が正しいかを可視化するために、画像上に円を描画してみたいと思います。
円の描画は先ほど出した座標と半径これにΘを利用して描画していきます。
Θを用いた円の座標をx,yごとにlistに保存し、その後該当セルを上書きすることで描画します。
xe=[]
ye=[]
theta=np.arange(0,2*math.pi,0.1)
for itheta in theta:
xe.append(int(np.round(r*math.cos(itheta)+a)))
ye.append(int(np.round(r*math.sin(itheta)+b)))
for y, x in zip(ye, xe):
dicom_list_37[y][x] = 0
動作確認
それでは最後に動作確認をしてみましょう。
表示は以下のコマンドで行います。
im=Image.fromarray(dicom_list_37)
plt.imshow(im)
plt.show()
黒い円が描画した円になります。
だいたい中央が取れているのではないでしょうか。
ノイズ除去しないと右上の点群によってずれが生じるので、前処理が大切になりますね。
まとめ
今回はdicom画像から心臓の中心点を求め、それを可視化してみました。
点群から正円を出す。結構面白いですね。フリーハンドで書いた円をきれいにしてくれたりできそうなので、これを使ってきれいな魔方陣かけるアプリとか作ってみようかなと思う今日この頃でした。