概要
時系列データのクラスタリングにtslearnという強力なパケージがあり、動作確認したときの備忘録
仕事で使えそうなのでちょっとつまみ食い
- 実施期間: 2020年10月, 2023年2月
- 環境:Ubuntu18.04LTS, Ubuntu22.04LTS
動作確認用Conda仮想環境作成
Miniconda Install 備忘録の手順で新しい動作確認用仮想環境を作成しておく
その上で必要な下記パケージをインストールする
conda install -c conda-forge tslearn
conda install -c conda-forge h5py
tslearnの手順にも書かれているとおり、他に下記が必要とのこと。
scikit-learn, numpy, scipy
準備
tslearnのクラスタリングは下記の3手法が実装されている。
- tslearn.clustering.KernelKMeans
- tslearn.clustering.KShape
- tslearn.clustering.TimeSeriesKMeans
今回はK-Shapeという手法を使用してみる。K-Shapeの概要はこのブログに書かれており、そこからオリジナルの論文も参照できる。
今回はtslearnのオフィシャルサンプルコードをベースに、不整脈波形のクラスタリングを試す。
データはKaggleからECG Heartbeat Categorization Datasetを使用した。
DNNではないので、mitbih_train.csvだけダウンロードしておく。
125Hzの波形データ187点と最後の列が0~4のラベルになっている。'0'が正常な波形で、それ以外は症状ごとに異なる波形らしい。
全87554件あるが、シャッフルして1000点で性能評価、100点を描画させ目視確認する。
コード(Multilabel classification)
kkk_oooさんに識別後の波形に色付け部を書いていただいたので、3年越しにコードと考察の見直しを行う。
旧コードは末尾の折りたたみ部へ移動
import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
from tslearn.clustering import KShape
from tslearn.datasets import CachedDatasets
from tslearn.preprocessing import TimeSeriesScalerMeanVariance
データを整形し、最後にスケーリングする。
0~4の5ラベルに分けるクラスタリングモデルと捉え、Confusion matrixで性能を確認する。
先頭1000個のsampleを使用する。
train_df=pd.read_csv('/home/hoge/mitbih_train.csv',header=None)
trainArr = []
trainArr = train_df.values
np.random.seed(seed = 42)
np.random.shuffle(trainArr)
trainArr_X = []
trainArr_y = []
trainArr_X = trainArr[:1000, :187]
trainArr_y = trainArr[:1000, -1:]
trainArr_X = trainArr_X.reshape([1000, 187, 1])
print(trainArr_X.shape) # (1000, 187, 1)
print(trainArr_y.shape) # (1000, 1)
sz = trainArr_X.shape[1]
# For this method to operate properly, prior scaling is required
trainArr_X = TimeSeriesScalerMeanVariance().fit_transform(trainArr_X)
tslearnのKShapeモデルを使用する。
from sklearn.metrics import multilabel_confusion_matrix
# kShape clustering
n_clusters = 5
ks = KShape(n_clusters=n_clusters, verbose=True, random_state=42)
y_pred = ks.fit_predict(trainArr_X)
trainArr_y = trainArr_y.reshape([1, 1000])
np_trainArr_y = trainArr_y.astype(np.int64)
multilabel_confusion_matrix(np_trainArr_y[0], y_pred)
array([[[107, 62],
[680, 151]],
[[928, 51],
[ 20, 1]],
[[718, 211],
[ 52, 19]],
[[764, 231],
[ 3, 2]],
[[656, 272],
[ 72, 0]]])
multilabel_confusion_matrixはone-vs-restに変換され作成される。
つまり、各matrixの1行目がsampleのrest、2行目がsampleのoneにあたり、1列目がpredictionのrest、2列目がpredictionのoneにあたる。
lable2について評価している3番目のMatrixを例に取ると、
19sampleについてlable2を正しく予測し、
718sampleについてそれ以外(lable0,1,3,4)を正しく予測し、
211sampleについてそれ以外のlabel0,1,3,4をlabel2と誤って予測し、
52sampleについてlabel2をそれ以外と誤って予測した、
と読む。
今回は医療データなので異常波形を正常波形と予測することが忌避されるが、それ以外(rest)に正常と異常が混ざってしまうためmultilabel_confusion_matrixでは忌避できたかどうかは評価できない。
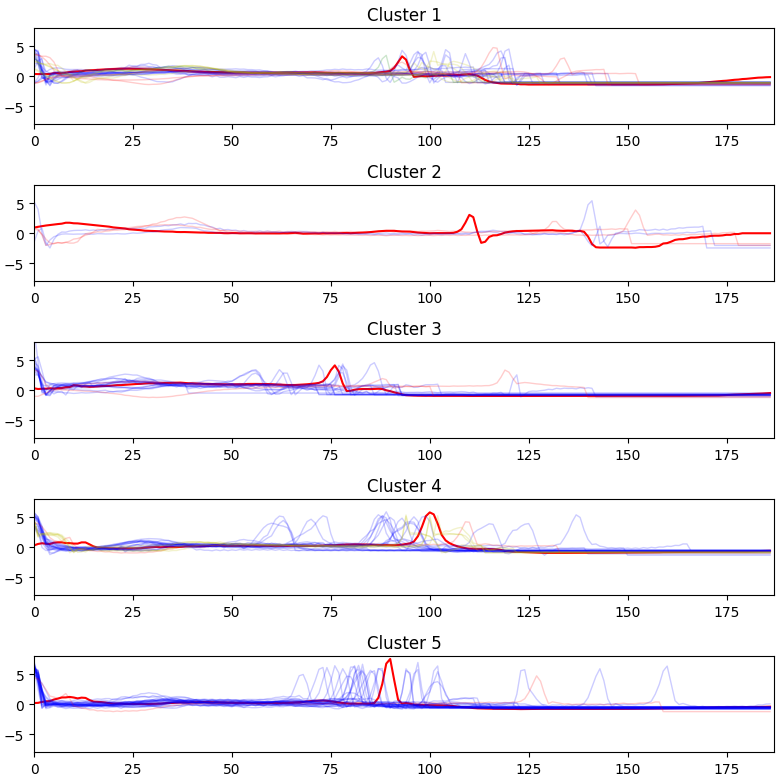
色分けできるようにコードを頂いたので、改造し描画させる。
まず、1000個中の先頭の100個について抜き取る。
trainArr_X = trainArr_X[:100, :, :]
trainArr_y = trainArr[:, :100]
np_trainArr_y = np_trainArr_y[:, :100]
y_pred = y_pred[:100]
で、描画部
# 先頭100個の描画部
class_lst = list(range(5)) # original data中の固定クラス(n_clustersとは異なる)
color_dict = {0: 'b-', 1: 'g-', 2: 'r-', 3: 'k-', 4: 'y-', }
fig, ax = plt.subplots(n_clusters, 1, figsize=(8, 8), tight_layout=True)
for yi in range(n_clusters):
trainArr_X_i = trainArr_X[y_pred == yi]
trainArr_y_i = np_trainArr_y[0][y_pred == yi]
ax[yi].plot(ks.cluster_centers_[yi].ravel(), "r-")
for lbl in class_lst:
for xx in trainArr_X_i[trainArr_y_i == lbl]:
ax[yi].set_xlim(0, sz)
ax[yi].set_ylim(-8, 8)
ax[yi].plot(xx.ravel(), color_dict[lbl], linewidth=1, alpha=.2)
ax[yi].title.set_text('Cluster %d' % (yi + 1))
理想は上から、青線のみ、緑線のみ、赤線のみ、黒線のみ、黄色線のみ、だが、混在していることがわかる。
コード(Binary classification)
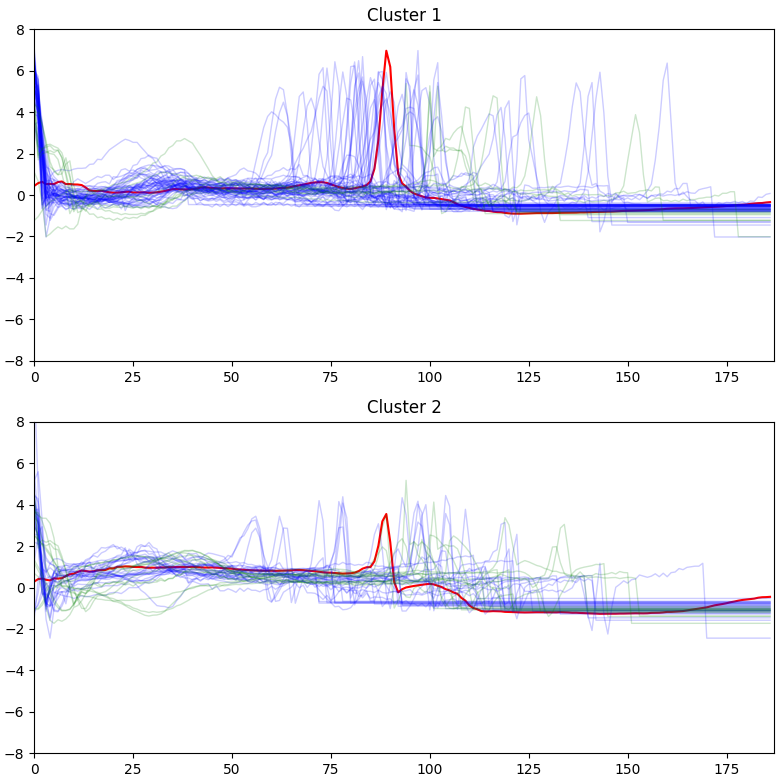
正常な波形と異常な波形の二値問題にしたときの精度も見てみる。
label0はそのまま、それ以外のlabelをlabel1とする。
変更は下記2行。
# csv.read()直後に下記実行
train_df.loc[train_df[187] >= 1.0, 187] = 1.0 # 0.0以外のラベルは1.0に置き換え、binary classification 問題とする
# kShape clustering
n_clusters = 2
array([[[105, 64],
[326, 505]],
[[505, 326],
[ 64, 105]]])
異常波形を示すlabel1に関する2番めのmatrixから、異常波形を正常波形と予測することが忌避されたか確認できる。
64sampleについて異常波形を正常波形と予測していることがわかる。つまり、
recall = 105/(105+505) = 17.21%
これが良いのか悪いのかは本投稿の範疇外
また、326sampleの正常波形を異常と予測し「要精密検査」判定の手紙が送られてしまうことになった。
波形は下図となる。
confusion matrixでは性能は見劣りしたが、波形的には波高値方向に分類できているっぽいように見える。
Kshape()に調整できるパラメータが殆どないので、これが限界と思われる。
クラス数が少ない問題に適しているのかもしれない。
以上
旧コード
コード
必要ライブラリをインポートする。
import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
from tslearn.clustering import KShape
from tslearn.datasets import CachedDatasets
from tslearn.preprocessing import TimeSeriesScalerMeanVariance
データの整形し、最後にスケーリングする。
train_df=pd.read_csv('/home/hoge/mitbih_train.csv',header=None)
trainArr = []
trainArr = train_df.values
np.random.shuffle(trainArr)
trainArr_X = []
trainArr_y = []
trainArr_X = trainArr[:100, :187]
trainArr_y = trainArr[:100, -1:]
trainArr_X = trainArr_X.reshape([100, 187, 1])
print(trainArr_X.shape) # (100, 187, 1)
print(trainArr_y.shape) # (100, 1)
sz = trainArr_X.shape[1]
# For this method to operate properly, prior scaling is required
trainArr_X = TimeSeriesScalerMeanVariance().fit_transform(trainArr_X)
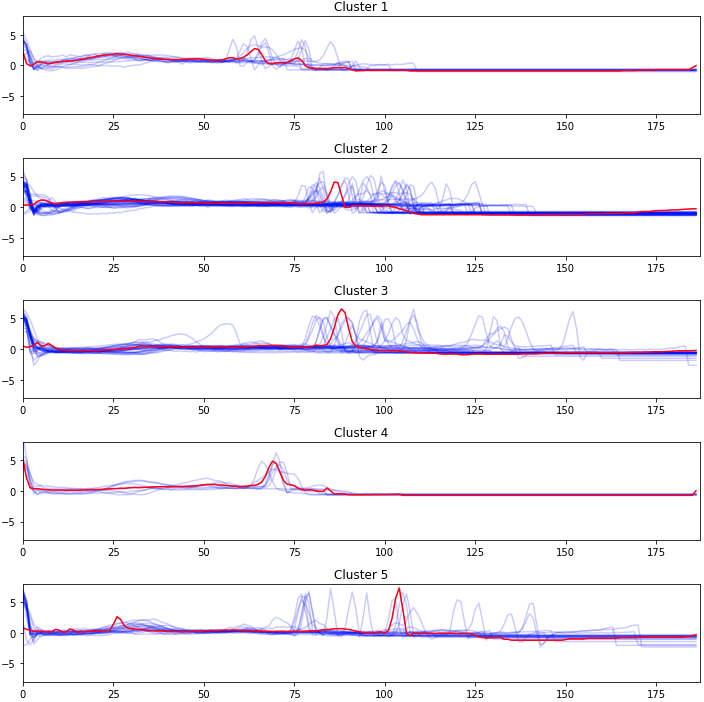
クラスタリングの実行と結果の表示する。クラスは5個とした。
# kShape clustering
ks = KShape(n_clusters=5, verbose=True, random_state=seed)
y_pred = ks.fit_predict(trainArr_X)
plt.figure(figsize=(10, 10), tight_layout = True)
for yi in range(5):
plt.subplot(5, 1, 1 + yi)
for xx in trainArr_X[y_pred == yi]:
plt.plot(xx.ravel(), "b-", alpha=.2)
plt.plot(ks.cluster_centers_[yi].ravel(), "r-")
plt.xlim(0, sz)
plt.ylim(-8, 8)
plt.title("Cluster %d" % (yi + 1))
plt.show()
結果
下図を得る。赤線は各クラスタの中心に一番近い波形と思われる。
(ホントは100本の線を病名ごとにtrainArr_yで色分けしたかったけど、Python力不足で直ぐに書けなかったので、後日対応)
ここで使用した100個の元データのラベル数(上図のCluster番号とは無関係)は下記の通り。
'0': 86個、 '1': 2個、 '2': 4個、 '3': 2個、 '4': 6個
ラベルの数と各クラスの数が明らかに異なっているので、何も調整せずに使用するわけには行かない模様。どのようなパラメータがあるのか要調査。
なお、生データは187点中、後半はゼロでパディングされていた。そのまま突っ込んだがこれは問題ではなかったようにみえる。
このデータセットをCNNでClassificationするコードもKaggleにあり、やはり精度は抜群に良い。