はじめに##
交差検証は「ものさし」のようなものです。いくら良い結果が出たところでその「ものさし」の目盛りがズレていては全ての長さが間違っていたことになります。交差検証も同様です。交差検証の方法が間違っていた場合いくら良いモデルが出来たと思っても間違っていることになります。全ての努力は水の泡です。ですから交差検証は何かの問題を解くときに一番最初に取り組むものですし、機械学習の中で一番重要と言っても過言ではないです。この記事ではそんな交差検証を紹介し、良い「ものさし」が作れるデータサイエンティストを目指します。
目次##
基礎から発展の順で書きます。基礎では主にsklearnのドキュメントを参考にし、発展のところではkaggleなどで実際に使われたCVの手法を書きます。発展は面白いCVが多いですので是非参考に。
なお筆者は専門家ではないので(一応確認はしているが)間違いもあるかと思われます。その時は下のコメントか編集リクエストを頂けると有難いです。
基礎
- KFold
- Stratified KFold
- Leave One Group Out (LOGO)
- Repeated KFold
- Shuffle Split
- Stratified Shuffle Split
- Leave One Out (LOO)
- Leave P out
- Group KFold
- Leave P Groups Out
- Time Series Split
- Pre-defined Fold-Splits
発展
- Nested Cross Validation
- CV For Blending
- CV① For Stacking
- CV② For Stacking
- CV③ For Stacking
- gbm.cv()
- Adversarial Validation
良く使うCV#
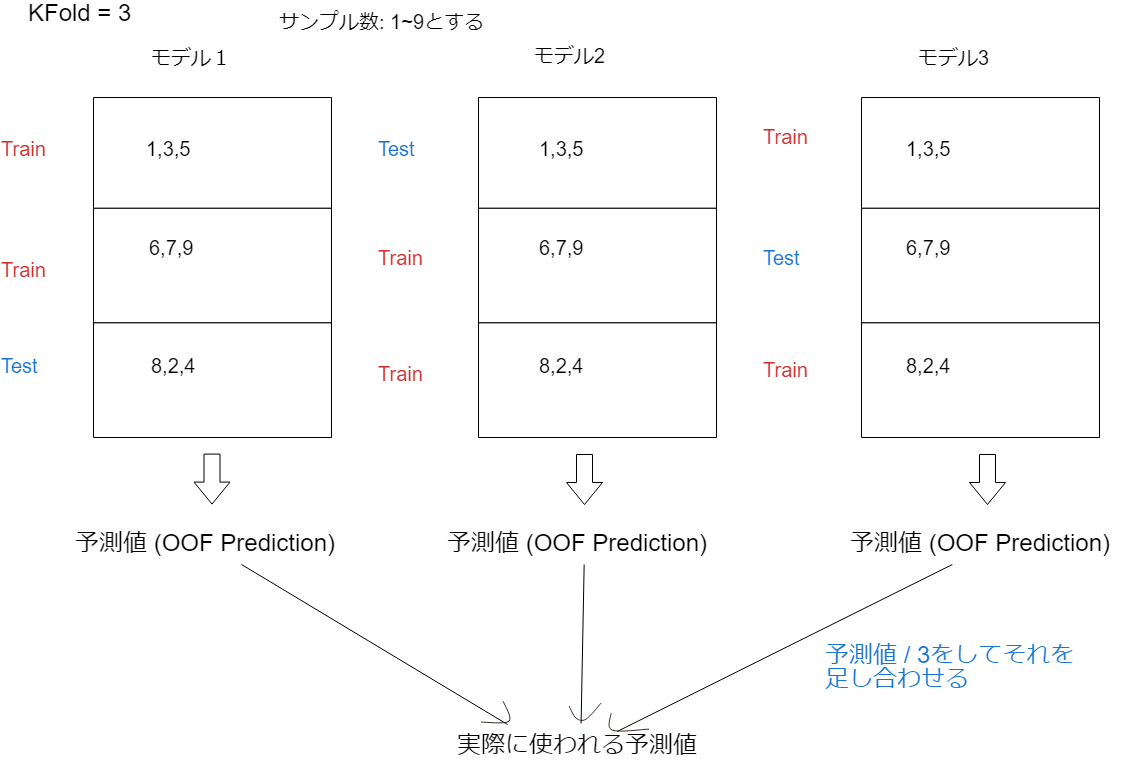
KFold##
KFoldとはデータセットをKグループ(フォールド)に等しく分割します。そのうちの1グループ(フォールド)をテストデータ、他のグループ(フォールド)を訓練データとし一つのモデルを構築します。これをK回グループ間でテストデータをローテーションすることでK個のモデルを作ります。最終的な予測値は殆どの場合K個のモデルの予測値の平均で決めます。
またOOF予測値とはOut Of Fold (Prediction)から来ており、文字通りフォールドから出てきた予測値という意味で頻繁に使われます。またどのグループにどのサンプルを置くかはランダムに決められ、random_stateというパラメータでランダム性を設定可能です。
import numpy as np
from sklearn.model_selection import KFold
X = ["a", "b", "c", "d"]
kf = KFold(n_splits=2)
for train, test in kf.split(X):
print("%s %s" % (train, test)) #train,testはサンプルのインデックス
# n_splits=2なので2グループ
[2 3] [0 1] #訓練データ=c,d テストデータ=a,b
[0 1] [2 3] #訓練データ=a,b テストデータ=c,d
基礎の実装は全てsklearnのドキュメントから引用。
Stratified KFold##
Stratified KFoldとは基本構造はKFoldです。しかしグループを分割するときにある一定の規則性を与えます。具体的にはラベルのクラスの分布がテストデータと訓練データに出来るだけ均等に渡るようにグループが分けられます。例えばラベルが0と1の分類問題があったとしましょう。ラベル1の数が10個でラベル0の数が100個だとします。このときKFoldで分けてしまうとテストデータにラベル1の数が無かったりと不均等になります。これはCVが誤った精度を出力してしまう可能性があり危険です。Stratified KFoldを使えば、このようなラベルのクラスの分布が偏っているデータ(不均衡データ)に対処できます。
from sklearn.model_selection import StratifiedKFold
X = np.ones(10)
y = [0, 0, 0, 0, 1, 1, 1, 1, 1, 1]
skf = StratifiedKFold(n_splits=3)
for train, test in skf.split(X, y):
print("%s %s" % (train, test)) #train,testはサンプルのインデックス
# 全体のラベル数は0が4つ、1が6つ。
# テストと訓練で不均等にならないようにサンプルを分ける。
[2 3 6 7 8 9] [0 1 4 5]
[0 1 3 4 5 8 9] [2 6 7]
[0 1 2 4 5 6 7] [3 8 9]
Leave One Group Out (LOGO)##
LOGOとは予め分けるグループ(フォールド)を決めておいて1グループをテストデータとし他のグループを訓練データとする分割方法です。ちなみにgroups[]の中に各サンプルのインデックスに応じてグループナンバーを指定します。
from sklearn.model_selection import LeaveOneGroupOut
X = [1, 5, 10, 50, 60, 70, 80]
y = [0, 1, 1, 2, 2, 2, 2]
groups = [1, 1, 2, 2, 3, 3, 3]
logo = LeaveOneGroupOut()
for train, test in logo.split(X, y, groups=groups):
print("%s %s" % (train, test)) #train,testはサンプルのインデックス
[2 3 4 5 6] [0 1] #テストデータはグループ1
[0 1 4 5 6] [2 3] #テストデータはグループ2
[0 1 2 3] [4 5 6] #テストデータはグループ3
その他のCV#
Repeated KFold##
これはKFoldを指定された回数分リピートする分割法です。なおKFoldのサンプルの分割法は1リピートにつきランダムで変わりますので毎回のリピートでサンプルが同じようなグループになることはありません。
import numpy as np
from sklearn.model_selection import RepeatedKFold
X = np.array([[1, 2], [3, 4], [5, 6], [7, 8]])
random_state = 12883823
rkf = RepeatedKFold(n_splits=2, n_repeats=2, random_state=random_state)
for train, test in rkf.split(X):
print("%s %s" % (train, test)) #train,testはサンプルのインデックス
[2 3] [0 1] #訓練データ=[5, 6],[7, 8] テストデータ=[1, 2],[3, 4]
[0 1] [2 3]
[0 2] [1 3] #ここから2回目
[1 3] [0 2]
Shuffle Split##
KFoldと違ってフォールド数以外にもテストサイズを指定できます。KFoldを緩めたバージョンと考えて良いでしょう。
from sklearn.model_selection import ShuffleSplit
X = np.arange(10)
ss = ShuffleSplit(n_splits=5, test_size=0.25,
random_state=0)
for train_index, test_index in ss.split(X):
print("%s %s" % (train_index, test_index)) #train,testはサンプルのインデックス
[9 1 6 7 3 0 5] [2 8 4]
[2 9 8 0 6 7 4] [3 5 1]
[4 5 1 0 6 9 7] [2 3 8]
[2 7 5 8 0 3 4] [6 1 9]
[4 1 0 6 8 9 3] [5 2 7]
Stratified Shuffle Split##
StratifiedKFoldと違ってフォールド数以外にもテストサイズを指定できる。StratifiedKFoldを緩めたバージョンと考えて良い。
Leave One Out (LOO)##
これはLeave One Outというより、Leave One Sample Outと読むと分かりやすいでしょう。文字通り一つのサンプル(データフレームの中の一行だけ)をテストセットとし、他を訓練データとすることです。あとはKFoldのようにテストセットをサンプル間でローテーションします。よってフォールド数=モデル数=サンプル数となります。
from sklearn.model_selection import LeaveOneOut
X = [1, 2, 3, 4]
loo = LeaveOneOut()
for train, test in loo.split(X):
print("%s %s" % (train, test)) #train,testはサンプルのインデックス
[1 2 3] [0]
[0 2 3] [1]
[0 1 3] [2]
[0 1 2] [3]
Leave P out##
これはLeave P Samples Outです。Leave One Sample OutのOneがPに置き換わった分割法です。毎回のフォールドでPサンプル数をテストセットとするという意味です。
from sklearn.model_selection import LeavePOut
X = np.ones(4)
lpo = LeavePOut(p=2)
for train, test in lpo.split(X):
print("%s %s" % (train, test)) #train,testはサンプルのインデックス
# テストのサンプル数が2つなのに注目。Leave P=2 Samples Out
[2 3] [0 1]
[1 3] [0 2]
[1 2] [0 3]
[0 3] [1 2]
[0 2] [1 3]
[0 1] [2 3]
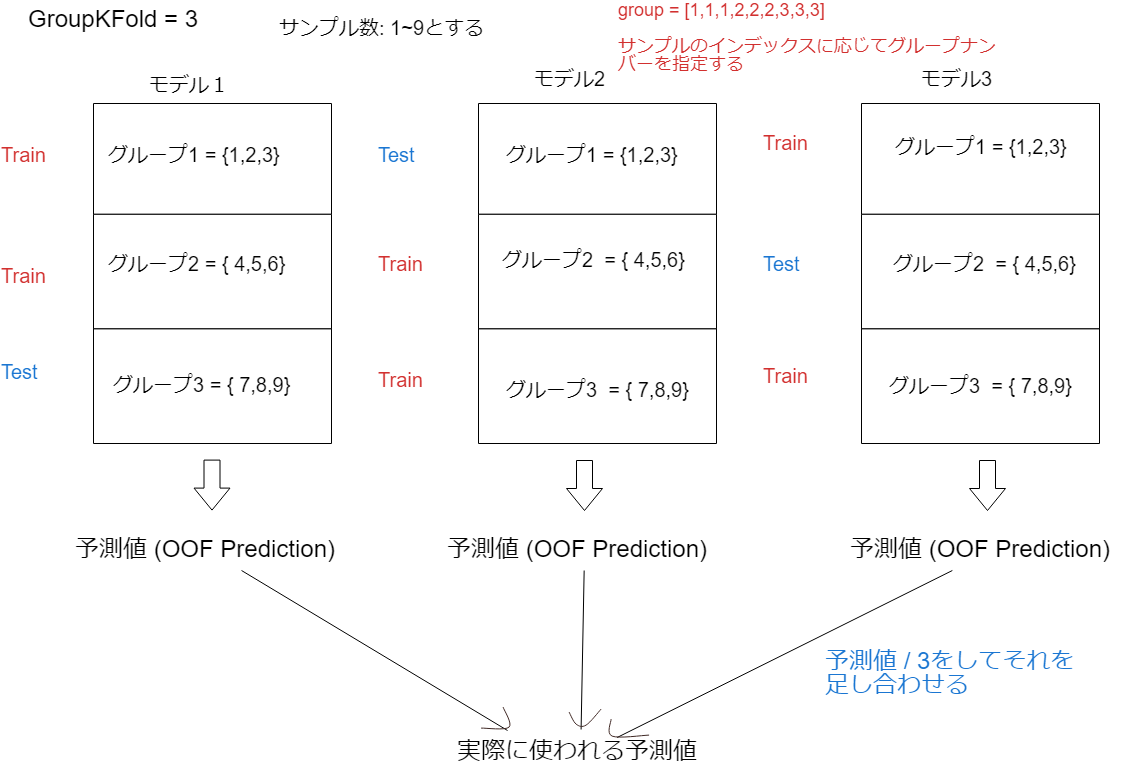
Group KFold##
Group KFoldとはグループを作ってグループ単位で訓練とテストデータへ分割します。そのグループ内のサンプルが訓練とテストデータの両方に置かれることを防ぎます。Leave One Group Outと同じです。ただしGroup KFoldではsplitsパラメータによってはテストデータが2グループになることもあります。
from sklearn.model_selection import GroupKFold
X = [0.1, 0.2, 2.2, 2.4, 2.3, 4.55, 5.8, 8.8, 9, 10]
y = ["a", "b", "b", "b", "c", "c", "c", "d", "d", "d"]
groups = [1, 1, 1, 2, 2, 2, 3, 3, 3, 3]
gkf = GroupKFold(n_splits=3)
for train, test in gkf.split(X, y, groups=groups):
print("%s %s" % (train, test)) #train,testはサンプルのインデックス
[0 1 2 3 4 5] [6 7 8 9]
[0 1 2 6 7 8 9] [3 4 5]
[3 4 5 6 7 8 9] [0 1 2]
Stratified Group KFold##
訓練と検証用フォールドのグループの分布が偏らないように分割します。sklearnには実装されてないので以下を参考にしてください。
Leave P Groups Out##
これはPグループ数をテストセットにするという意味です。Leave One Group Outではテストセットは1グループだけでしたがLeave P Groups OutではテストセットはPグループ数あります。そして他のグループは全て訓練データとします。
from sklearn.model_selection import LeavePGroupsOut
X = np.arange(6)
y = [1, 1, 1, 2, 2, 2]
groups = [1, 1, 2, 2, 3, 3]
lpgo = LeavePGroupsOut(n_groups=2)
for train, test in lpgo.split(X, y, groups=groups):
print("%s %s" % (train, test)) #train,testはサンプルのインデックス
[4 5] [0 1 2 3]
[2 3] [0 1 4 5]
[0 1] [2 3 4 5]
Time Series Split##
Time Series Splitとは文字通り時系列データに使われる分割法です。これは時系列データを混ぜて学習させるがために起きるモデルへのデータリークを防ぎます。データリークとは何らかの方法で、その時点では知り得ない情報をモデルが学習してしまうことです。
例えば株予測モデルがあったとしてモデルが未来の値動きのパターンや値幅を学習してしまうことはデータリークに値します。データリークによって学習したモデルは通常より悪い意味で精度が良くなります。Time Series Splitを使うことでこれを防ぐことが可能です。
具体的には訓練データを時系列の古いデータから一つずつ付け足していくことでデータリークを防いでいます。
from sklearn.model_selection import TimeSeriesSplit
X = np.array([[1, 2], [3, 4], [1, 2], [3, 4], [1, 2], [3, 4]])
y = np.array([1, 2, 3, 4, 5, 6])
tscv = TimeSeriesSplit(n_splits=3)
print(tscv)
TimeSeriesSplit(max_train_size=None, n_splits=3)
for train, test in tscv.split(X):
print("%s %s" % (train, test)) #train,testはサンプルのインデックス
[0 1 2] [3]
[0 1 2 3] [4]
[0 1 2 3 4] [5]
追記:データリークを起こす可能性のある訓練データで良く見られるのはフォールド数を増やせば増やすほどCVの値が良くなっていくことです。これはフォールド数を増やすことで自然と訓練データの数が増えるがために起きます。訓練データが増えればモデルがより多くのリークデータを学習してしまうので精度が極端に良くなります。
ちなみにデータリークのない訓練データでもフォールド数を増やせばCVの値が良くなることはよくあります。訓練データの数を増やせばその分モデルの精度は良くなりますし、early_stoppingするときのラウンドは検証用データで判断されます。よって検証用データの都合の良いところで学習がストップするので、検証用データのエラー値(CV値)は概ね良いです(検証データのスコアが悪くても改善しなければ学習をストップするので勿論その限りではない)。しかし検証用データが少なくなればそれだけ学習をストップするか否かの判断がその検証用データに偏るので本命のテストデータでは精度が悪くなります。
Pre-defined Fold-Splits##
分割方法を予め設定できるのがPre-defined Fold-Splitsです。PredefinedSplit()にテストセットのインデックスをどう組み合わせるかを予め設定できます。
import numpy as np
from sklearn.model_selection import PredefinedSplit
X = np.array([[1, 2], [3, 4], [5, 6], [7, 8]])
y = np.array([0, 0, 1, 1])
# test_fold = [[1, 2]のアドレス, [3, 4]のアドレス, [5, 6]のアドレス, [7, 8]のアドレス]
test_fold = [0, 1, -1, 1]
ps = PredefinedSplit(test_fold)
for train_index, test_index in ps.split():
print("TRAIN:", train_index, "TEST:", test_index)
TRAIN: [1 2 3] TEST: [0]
TRAIN: [0 2] TEST: [1 3]
[1,2]のアドレスは0なので最初のテストセットとなる。
[3, 4]と[7, 8]のアドレスは1なのでその後のテストセットとなる。
なお[5, 6]は-1なのでテストセットから除外する。(-1はそのサンプルをテストセットに使わないという意味)
発展#
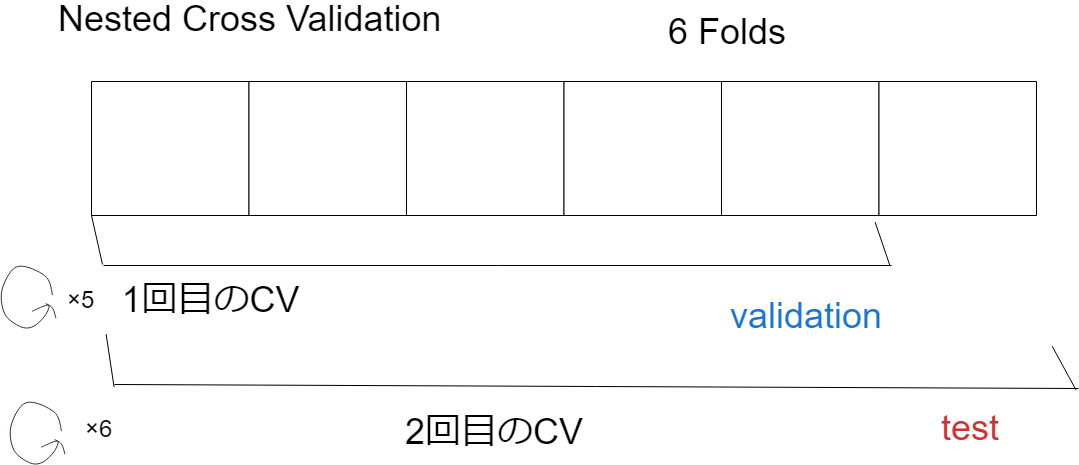
Nested Cross Validation##
Nested Cross Validationとは主にハイパーパラメーターチューニング(以下、長いのでハイパラチューニング)した後に、さらにモデル評価をしたいときに使います。具体的には予めフォールドには加えないデータセットを確保(ホールド)しておいて、その他のデータだけをフォールドに分割して交差検証します。その後訓練し終わったモデルに予め確保しておいたデータセットを予測させます。その予測値と実測値の違いをCVエラーとしてハイパーパラメータの評価に使います。
そもそもグリッドサーチやらOptunaでハイパラを探索しすぎるとデータを過学習してしまいます。特に多くの特徴量+少ないデータで長く探索すると過学習したモデルを容易に作れます。なぜならそのデータに合わせたハイパラになってしまうからです。これを防ぐためにはある程度重要な特徴量が出揃っているスタンダードなモデルが出た状態でハイパラチューニングしてそれを固定するか、Nested Cross Validationを使います。実際にkaggleでも使われています:Top 10 - Solution - Giba and Amjad
Nested Cross Validationは二重forループのようなものです。再三になりますが内側のループではテストセットだけ外しておいて(ホールドしておいて)普段通りモデルを作りハイパラチューニングします。そして外側のループでテストセットの精度を測ります。これを全フォールド回数(テストセットも回して)最終的なモデルの精度を測ります。
一つ注意点としてはデータ量が多い場合処理コストが、かなりかかることです。iフォールドならば内側ループにi-1フォールド*(外側で)iフォールドかかります。
ちなみにハイパラチューニングした後モデル評価としてNested Cross Validationを使う以外に普通にテストセットをホールドしておきたいときにも使えます。例えばこのkaggleソリューションではNestedCVの構造を使っています。NestedCVの使いどころは各フォールドの相関性が低いときです。各フォールドのデータの分布がバラバラだと正確に精度を測れなくなります。なぜなら検証用データ次第で反復数が異なりモデル自体の構造も違ってくるからです。場合によっては過学習/未学習します。またそれらの検証用データのスコアを平均化してCVとするのでデータ全体の精度としては正しくない可能性が高いです。逆に相関性が高い分布が似ているフォールド群の場合は検証用データと他のフォールドとで違いが出ずモデルも安定します。
相関が低いときは(訓練可能なデータ量にもよりますが)検証用データに惑わされないようにホールドセットを確保しておくのが良策です。
実装はこちらが参考になりました。最初にグリッドサーチでパラメータを探したあとにcross_val_score()で交差検証しています。
CV For Blending##
ブレンディングとはいくつかの既存のモデルの予測値を重みづけして足し合わせ最終の予測値を出すことです。例として分かりやすいのは全てのモデルの予測値を足してモデルの数で割ることで、全モデルの予測値の平均を出すことが挙げられます。しかしこれを平均でなく$M=W1M1+W2M2+W3*M3$としたいとき各々の重みをどう推測すべきでしょうか。
考えられるのは例えば予めデータセットの10%をホールドしておいて他の90%のデータでモデルに訓練させます。その後夫々のモデルに残りの10%を予測させ重みを自分で決めるか、ロジスティック回帰などのスタッキングモデルから学びます(OOF予測値1を特徴量としているのでFeature Importanceで分かるはず)。しかしこの手法は10%のデータセットで決めるのでデータセット自体が少ない場合はお勧めできません。またホールドするデータセットの分布も偏らないようにする必要があります。
補足:重みを決めるもう一つの手法として、ブレンディングに含めなかったが、かなり良く予測できるモデルを参考にする手法があります。具体的にはブレンディングしたモデルの予測値が、その含めなかったモデルの予測値に出来るだけ相関するように各々のモデルの重みを最適化します。これをすることで安定したモデルの精度を保ちながらも、モデルにさらなる多様性を与えることができます。
参考ディスカッション:
Strategy B https://www.kaggle.com/general/18793
参考コンペ:
https://www.kaggle.com/c/otto-group-product-classification-challenge/discussion/14335https://www.kaggle.com/c/otto-group-product-classification-challenge/discussion/14335
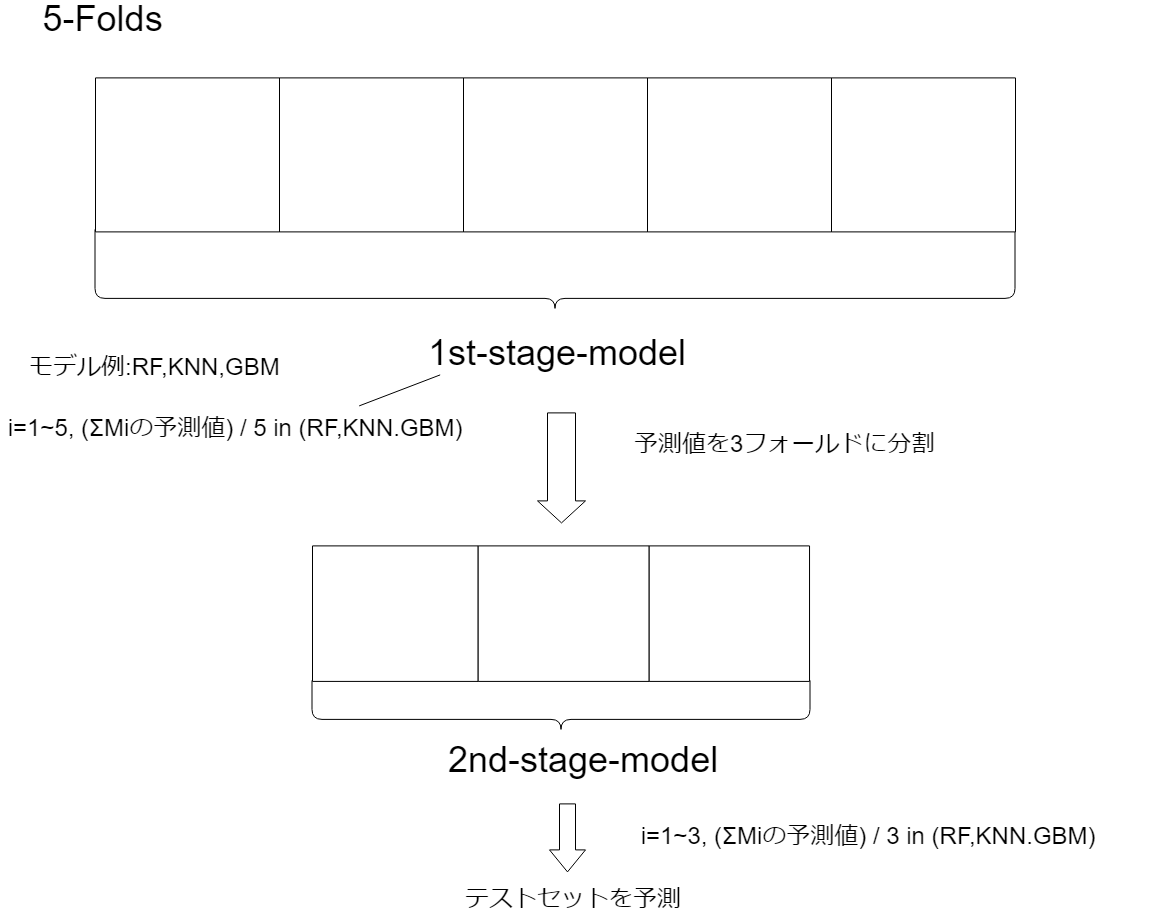
CV① For Stacking##
「スタッキング」とはいくつかの既存のモデルの予測値を特徴量として最終的なモデルに予測させることです。この「スタッキング」をするときに考えられるCV戦略があります。それは既存の1st-levelモデルにKフォールド分を学習させ、そのOOF予測値1を2nd-levelモデル(スタッキングモデル)にPフォールド分学習させることです。しかしデータリークする?可能性が挙げられています。
参考ディスカッション:
Strategy A https://www.kaggle.com/general/18793
参考元:
https://mlwave.com/kaggle-ensembling-guide/
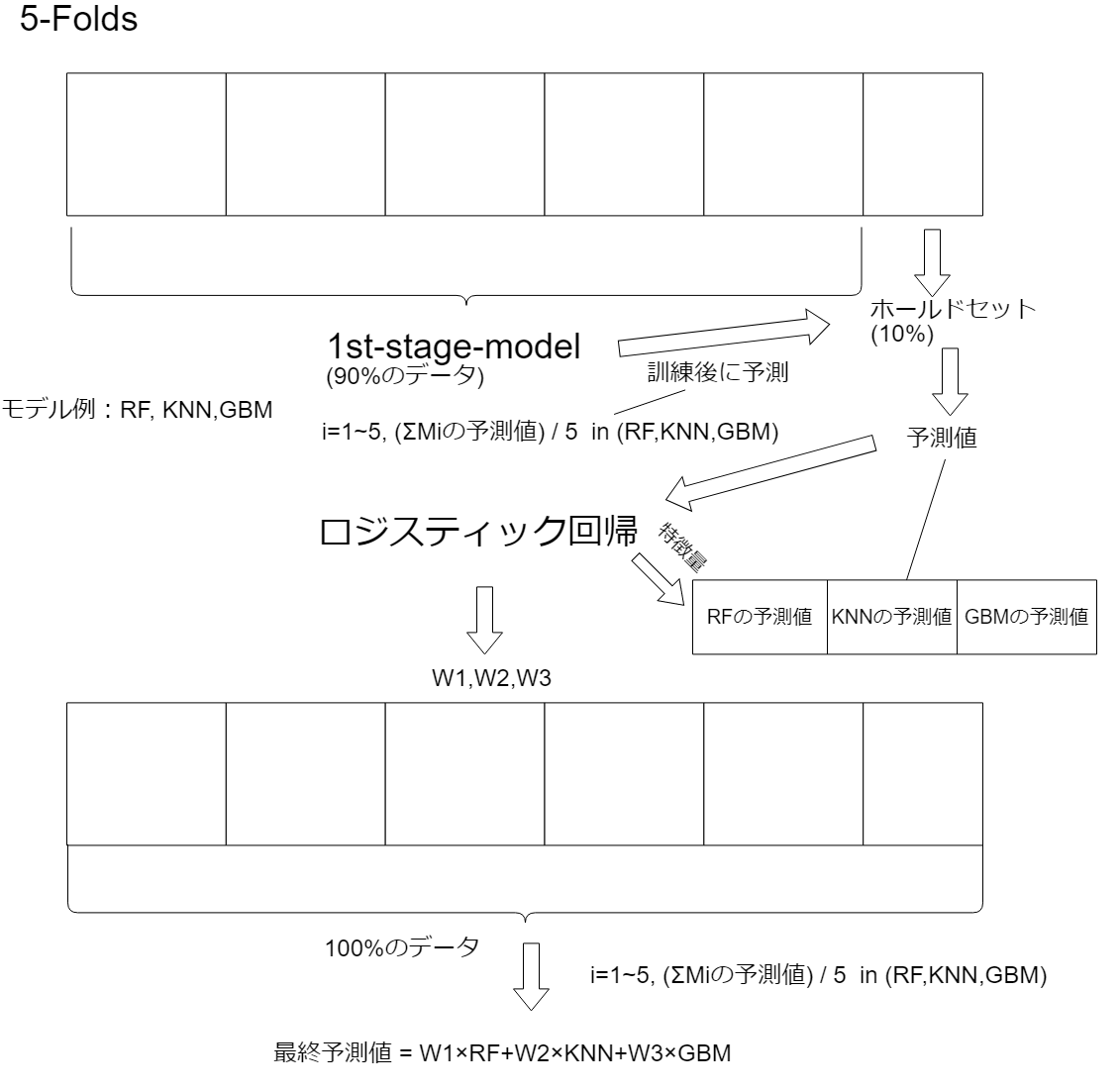
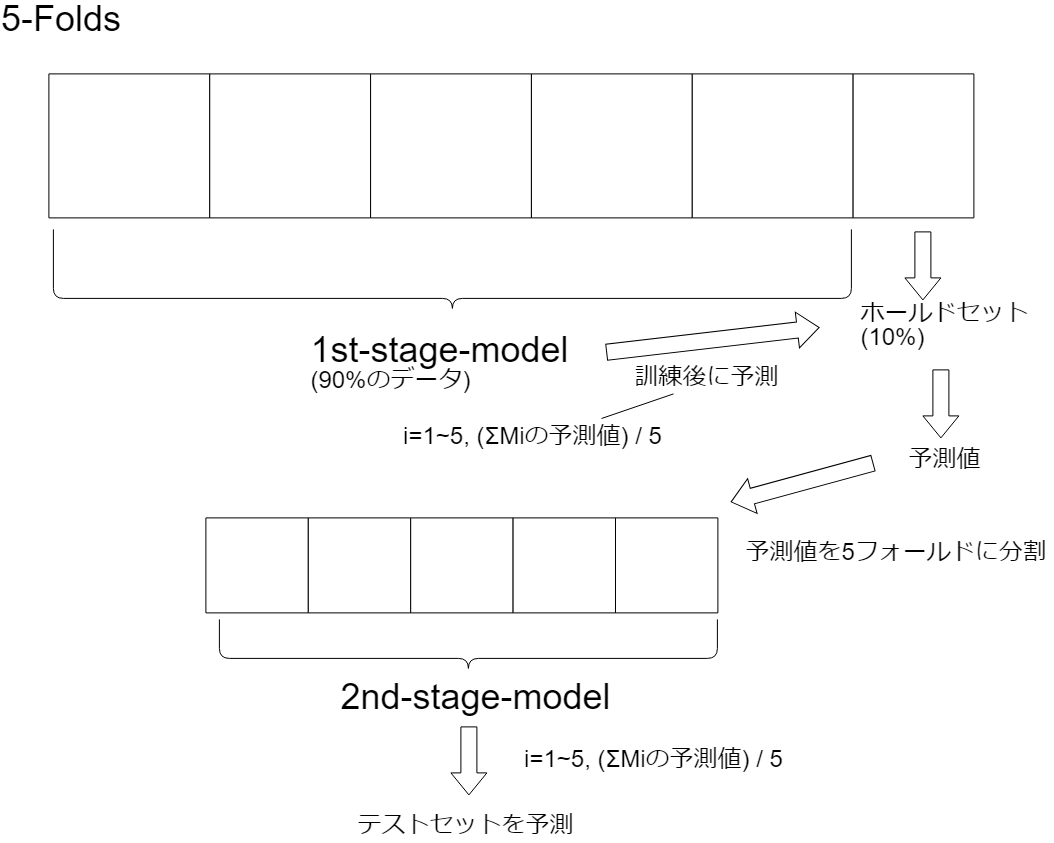
CV② For Stacking##
これはCV For Blendingの10%のホールドアウトをブレンディングの重み決めではなくそのままK-Foldするのに使います。まず訓練データの10%をホールドし、残りの90%でKフォールド1st-stageモデルを訓練させます。その次にその訓練したモデルで残りの10%の訓練データを予測します。そしてその予測値をPフォールドに分割して2nd-stageモデル(スタッキングモデル)にPフォールド分訓練させます。しかしこの手法は10%だと訓練データが極端に少ないので殆ど使われていません。
参考ディスカッション:
Strategy B https://www.kaggle.com/general/18793
参考元:
https://mlwave.com/kaggle-ensembling-guide/
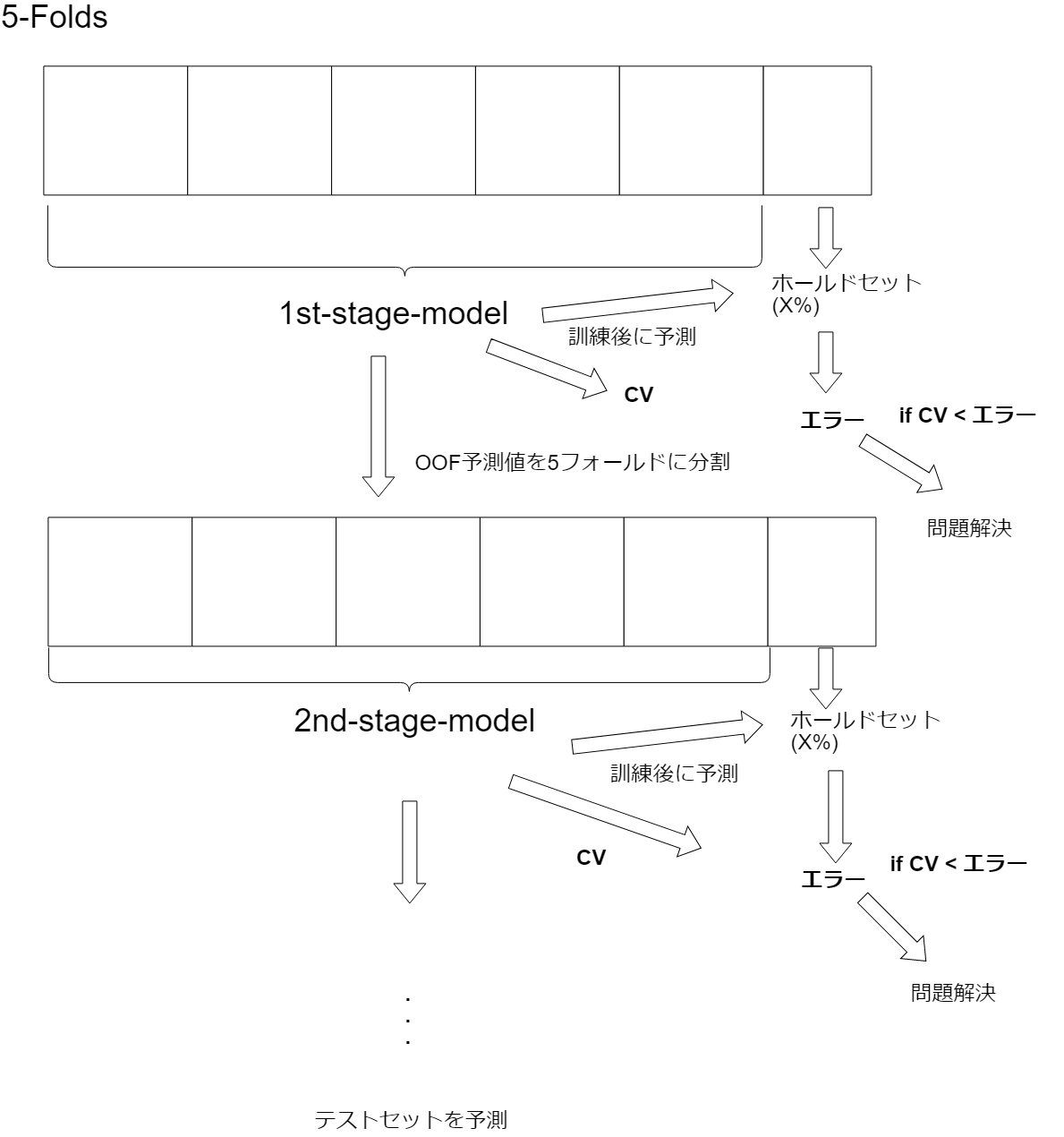
CV③ For Stacking##
この手法は最初に訓練データからx%のホールドを作ります。そして残りのホールド以外の訓練データでPホールド分訓練します。その後その訓練済みモデルでx%のホールドアウトを予測し、そのエラーとPフォールドで得たCVを比べます。もしCV < x%ホールドアウトエラーだったら、モデルがおかしい?のでそっちを解決します。もしもCV >= x%ホールドアウトエラーだったら、前のpフォールドモデルで得たOOF予測値[^1]を2nd-levelモデルの特徴量として訓練します。そして訓練後数%のホールドアウトを予測し、2nd-levelモデルのCVとx%のホールドアウトのエラーを比べます。CV < ホールドアウトのエラーなら加えたモデルが良くないと考えてそこを解決します。そうでないなら手順をリピートします。なぜCV < x%ホールドエラーならば手順を中断するのでしょうか。ディスカッションには詳しい理由は書かれていませんでしたが、恐らく過学習しているからだと考えられます(間違っていたら教えてください)。極端にCV < エラーとなればホールドしてないデータに過学習してしまっていることが想像できるでしょう。スタッキングは過学習しやすいので気を付ける必要があります。
当たり前ですがx%のホールドを選ぶときは、それぞれのデータの分布が異なるときは、その異なるデータを均等に配分し少ないデータならばxを小さくするか、この手法自体を使わないほうが良いかもしれません。またデータが大きいならばxを大きくすると安定したモデルを手にできるでしょう。
参考ディスカッション:
Strategy C https://www.kaggle.com/general/18793
gbm.cv()##
gbm.cv()はTraining API(gbm.train()などがあるところ)内に置いてある関数です。gbm.cv()はKFoldを使って同じイテレーション分(反復数分、ツリーを加えた数分)訓練されたモデルを抽出し一つの最適なCV値を出力します。例えばモデル1が1000反復、モデル2が400反復だとしてgbm.cv()は700反復だったときのCV値を使います。
これをなぜ使う必要があるかについて話します。まず基礎などで説明していたようにそのままKFoldを使うと各フォールドのモデルはそのフォールドに過学習します。(early_stopping_roundsを極端に低い値に設定すればその限りではありませんが。)例えばearly_stopping_rounds=100だったら各フォールドのモデルは100回モデルが改善しないところで学習をストップするためそのフォールドに過学習していることになります(early_stopping_roundsを設定しないときとほぼ変わらない)。これはスタッキングする以外の場合は問題ありません。なぜなら大抵の場合は全フォールドの予測値を足して平均化するからです。過学習した相関性の少ないモデル群を平均化すれば一般化されたモデルができあがります。ただしスタッキングをする場合は問題になります。
これを防ぐためにgbm.cv()を使います。gbm.cv()ではearly_stopping_roundsを使い各々のフォールドでearly_stopping_rounds分改善しないところで学習をストップします。しかし最終的なCVは同じ反復数(round数)訓練された一つのCVを出力します。gbm.cv()を使うことでテストセットに理想的なツリーの数(round数, 反復数)を知ることができます。
# lightgbmの他にもXGBoostやCatBoostにも実装されてます。
lgb.cv(params,
data,
num_boost_round=9999,
early_stopping_rounds=500,
nfold=5,
shuffle=True,
seed=0
)
Adversarial Validation##
Adversarial Validationは主にkaggleなどのコンペに使われます。実際の問題にはあまり使われませんが、考え自体は面白いので書いときます。
Adversarial Validationとは訓練データからテストデータに類似しているものだけを取り出して交差検証することです。
Kaggleでは訓練データでモデルに訓練させた後、そのモデルでテストセットを予測し結果をコンペに提出しますが、たまにテストセットの分布が訓練データと大きく異なっていることがあります。これでは訓練データから得たCVと実際に評価されるテストセットのスコアにズレが生じてしまいます。自分のCVとテストセットのスコアがズレているとCVが間違っている可能性が高くなります。するとハイパラチューニングや特徴量選択まで間違っていた可能性があります。これを防ぐために訓練データからテストセットに似たデータだけを取り出し交差検証します。
詳しい説明や実装はこちらが既に分かりやすかったので省きます。参考にしてください。
補足:Adversarial Validationの場合、テストセットによく似た訓練データを抽出して訓練する以外にも、単に訓練データとテストデータの分布の乖離を調べたいときにも使えます。(AUCが0.5から離れていれば訓練とテストの分布が異なると言えます。)この場合はAUCの乖離に大きく影響を及ぼしている特徴量を省くことなどが考えられます。テストデータによく似た訓練データだけを取り出すのはあくまでも選択肢の一つです。
またどの特徴量が影響を与えているか確かめる方法として特徴量一つ一つをAdversarial Validationで回す手もありますし、コルモゴロフ–スミルノフ検定(KS test)で訓練とテストのそれぞれの特徴量が同じ分布からかを確かめることもできます。(kaggleなどでも良く使われます。)
Adversarial Validationで分布を調べる実装の参考:https://www.kaggle.com/tunguz/lanl-adversarial-validation-shakeup-is-coming