三回目です。
今回のコンペはtestデータ自体が欠損したものであって、kaggleだけがprivateな欠損なしのデータを持っている。
このkaggleだけが持っているデータを推測しつつモデル作成をしなければならない。
この推測行為でコンペにアプローチすることをLeadearBoardProbingという。
kaggleのスコアが返ってくるleaderboardに表示されるスコア「publicScore」から本当のデータを推定すること。
早速
プライベートデータセットはtrainと構造が同じなのでしょうか?
答えはyesです。
通常のカグルコンペはtestとprivateが同じデータです。このコンペはprivate部分が含まれていません。
くわしくはこちら
リンクの中で書かれているのは、
以下リンクの内容
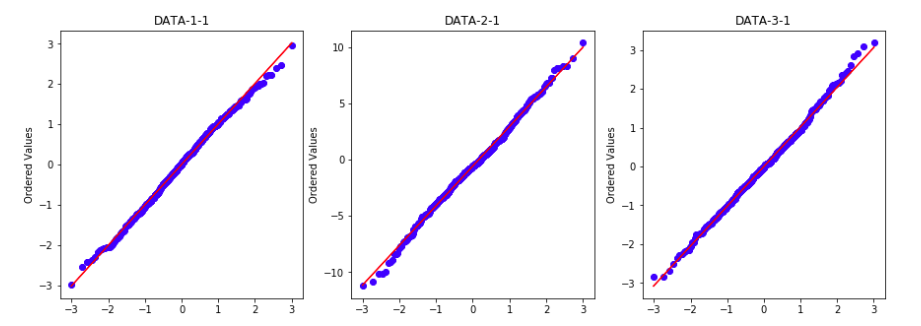
・データはwheezy-copper-turtle-magicのユニーク数512に対応した塊に分かれていて、正規分布していそうな形をしていること。
・細くとがったベル型の分布で一見正規分布ですが正確には正規分布ではありません。
・いくつかの変数は正規分布に従っています。
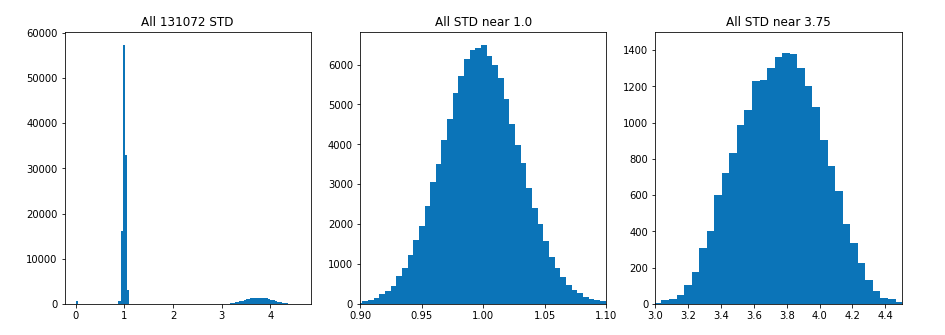
・512の変数に対して255個の変数があり、行の合計はだいたいtrainの全体(262144行)を512で割った数くらいです。
・前回SVMでやってみたところスコアは0.9くらいになりました。
・255個の変数を使ってしまうとオーバーフィッティングするので変数を減らすことが重要です。
・モデル構築の特徴量の選択はlassoやRFEで決めるといいのではないでしょうか。
・今回のデータは平均0,標準偏差1~3.75くらいのデータでした。
・標準偏差の大きいデータを選択してモデルを作成してみるとスコア0.9くらいでした。すべて標準偏差が1だったら特徴量選択は難しかったでしょう。
本文に戻る
プライベートデータセットについて
512のモデルを作る方法だけでなく単一のモデルを作成する方法も見てみます。
abhishekさんのカーネルとvladislavさんのneural netを使います。
import numpy as np, pandas as pd, os, gc
train = pd.read_csv('train.csv')
test = pd.read_csv('test.csv')
train.head()
使える特徴量を探して、使えないものを取り除きます。
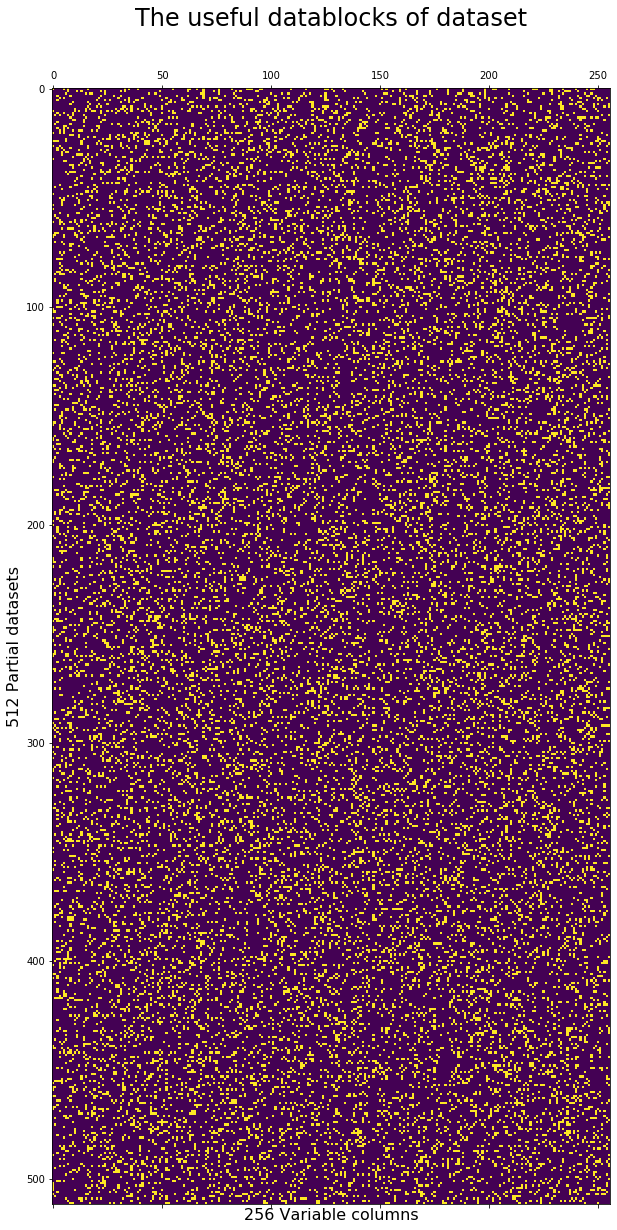
# FIND STANDARD DEVIATION OF ALL 512*256 BLOCKS
useful = np.zeros((256,512))
for i in range(512):
partial = train[ train['wheezy-copper-turtle-magic']==i ]
useful[:,i] = np.std(partial.iloc[:,1:-1], axis=0)
# CONVERT TO BOOLEANS IDENTIFYING USEFULNESS
useful = useful > 1.5
import matplotlib.pyplot as plt
# PLOT BOOLEANS OF USEFUL BLOCKS
plt.figure(figsize=(10,20))
plt.matshow(useful.transpose(),fignum=1)
plt.title('The useful datablocks of dataset', fontsize=24)
plt.xlabel('256 Variable columns', fontsize=16)
plt.ylabel('512 Partial datasets', fontsize=16)
plt.show()
標準偏差の判断基準値を1.5として、Trueになるものが黄色、Faulsが紫になる。
使えるもの(標準偏差1.5以上)が黄色であらわされている。
使えないデータ部分には0を入れましょう。
(pythonにはもっとうまいやり方があるとはおもいますが)
# SET MAGIC COLUMN AS ALL USEFUL
useful[146,:] = [True]*512
# REMOVE ALL USELESS BLOCKS
for i in range(512):
idx = train.columns[1:-1][ ~useful[:,i] ]
train.loc[ train.iloc[:,147]==i,idx ] = 0.0
test.loc[ test.iloc[:,147]==i,idx ] = 0.0
#if i%25==0: print(i)
使えないと認識された変数には0が入っている。
train.head()やtest.head()を確認するとわかる。
NNの構築
512のモデルに分けるのでなく、特徴量選択をした後、一つのモデルだけで結果を出してみる。
# SET MAGIC COLUMN AS ALL USEFUL
useful[146,:] = [True]*512
# REMOVE ALL USELESS BLOCKS
for i in range(512):
idx = train.columns[1:-1][ ~useful[:,i] ]
train.loc[ train.iloc[:,147]==i,idx ] = 0.0
test.loc[ test.iloc[:,147]==i,idx ] = 0.0
#if i%25==0: print(i)
# CUSTOM METRICS
def fallback_auc(y_true, y_pred):
try:
return metrics.roc_auc_score(y_true, y_pred)
except:
return 0.5
def auc(y_true, y_pred):
return tf.py_function(fallback_auc, (y_true, y_pred), tf.double)
この関数はあとでニューラルネットを実行するときに使われる。
ちなみにaucというのは
http://cookie-box.hatenablog.com/entry/2019/02/10/182619
このあたりを参考にして。
https://www.randpy.tokyo/entry/roc_auc
これがわかりやすかった True Positive(TP): 正解データ正であるものを、正しく正と予測できた数 False Positive(FP):正解データ負であるものを、間違って正と予測した数 Flase Negative(FN):正解データ正であるものを、間違って負と予測した数 True Negative(TN):正解データ負であるものを、正しく負と予測できた数
となります。 ※以降ですが、True PositiveはTP、False PositiveはFPといったように、表記を省略させて頂きますのでご了承ください。
さて、この表から正解率を求めるとすると、 正解率:(TP+TN)/(TP+FP+FN+TN) という式で計算することができます。(分母が全体の数、分子が正解した数)
正解率を評価指標として用いるのが直感的には良さそうではあるのですが、クラスに偏りがある場合、機能しなくなるという問題があります。
以上引用
予測値と正解クラスをひとつのデータとする。その予測値を閾値として全データを正負に振り分けたとき、TPFPTNFNがそれぞれ求まる。 これを全予測値に対して計算していく。 こうすることで、予測値と正解クラスの関係性がどれだけ正しいものなのかを確かめられる。 これをカーブにしたものがauc
2クラスを判断したいときに使える。 いろんなデータの特徴量をモデルにかけて一つの0~1の間の数字にする。 その数字と二つのクラスの分け目の閾値がうまく設定されていれば、良いモデルを使ったoutputであるといえる。
本文に戻って
# ONE-HOT-ENCODE THE MAGIC FEATURE
len_train = train.shape[0]
test['target'] = -1
data = pd.concat([train, test])
data = pd.concat([data, pd.get_dummies(data['wheezy-copper-turtle-magic'])], axis=1, sort=False)
train = data[:len_train]
test = data[len_train:]
trainとtestについてmagic変数に対してonehotencorderを行う。
ここではdummiesを使っているがやっていることはonehotと同じ。
# PREPARE DATA AND STANDARDIZE
y = train.target
# trainの正解一覧をyに入れる
ids = train.id.values
# idも別で保存
train = train.drop(['id', 'target'], axis=1)
# trainからidとtargetを取り除いておく
test_ids = test.id.values
# testのidは別保存
# testはtargetがない。
test = test[train.columns]
test_ids = test.id.values
# testのidは別保存
# testはtargetがない。
test = test[train.columns]
test_preds_NN = np.zeros((len(test)))
test_preds_NN
# テストの予測値をいれたい
scl = preprocessing.StandardScaler()
# 標準化(スケール)をsciに入れる
scl.fit(pd.concat([train, test]))
# trainとtestをくっつけたものを標準化している?r言語でいうrbind=concat
train = scl.transform(train)
test = scl.transform(test)
ニューラルネットの構築
NFOLDS = 15
RANDOM_STATE = 42
gc.collect()
# STRATIFIED K FOLD
folds = StratifiedKFold(n_splits=NFOLDS, shuffle=True, random_state=RANDOM_STATE)
for fold_, (trn_, val_) in enumerate(folds.split(y, y)):
#print("Current Fold: {}".format(fold_))
trn_x, trn_y = train[trn_, :], y.iloc[trn_]
val_x, val_y = train[val_, :], y.iloc[val_]
# BUILD MODEL
inp = Input(shape=(trn_x.shape[1],))
x = Dense(2000, activation="relu")(inp)
x = BatchNormalization()(x)
x = Dropout(0.3)(x)
x = Dense(1000, activation="relu")(x)
x = BatchNormalization()(x)
x = Dropout(0.3)(x)
x = Dense(500, activation="relu")(x)
x = BatchNormalization()(x)
x = Dropout(0.2)(x)
x = Dense(100, activation="relu")(x)
x = BatchNormalization()(x)
x = Dropout(0.2)(x)
out = Dense(1, activation="sigmoid")(x)
clf = Model(inputs=inp, outputs=out)
clf.compile(loss='binary_crossentropy', optimizer="adam", metrics=[auc])
# CALLBACKS
es = callbacks.EarlyStopping(monitor='val_auc', min_delta=0.001, patience=10,
verbose=0, mode='max', baseline=None, restore_best_weights=True)
rlr = callbacks.ReduceLROnPlateau(monitor='val_auc', factor=0.5,
patience=3, min_lr=1e-6, mode='max', verbose=0)
# TRAIN
clf.fit(trn_x, trn_y, validation_data=(val_x, val_y), callbacks=[es, rlr], epochs=100,
batch_size=1024, verbose=0)
# PREDICT TEST
test_fold_preds = clf.predict(test)
test_preds_NN += test_fold_preds.ravel() / NFOLDS
# PREDICT OOF
val_preds = clf.predict(val_x)
oof_preds_NN[val_] = val_preds.ravel()
# RECORD AUC
val_auc = round( metrics.roc_auc_score(val_y, val_preds),5 )
all_auc_NN.append(val_auc)
print('Fold',fold_,'has AUC =',val_auc)
K.clear_session()
gc.collect()
出力結果は
Fold 0 has AUC = 0.90394
Fold 1 has AUC = 0.90558
Fold 2 has AUC = 0.90671
Fold 3 has AUC = 0.9032
Fold 4 has AUC = 0.90656
Fold 5 has AUC = 0.90635
Fold 6 has AUC = 0.90499
Fold 7 has AUC = 0.90905
Fold 8 has AUC = 0.90222
Fold 9 has AUC = 0.90496
Fold 10 has AUC = 0.90572
Fold 11 has AUC = 0.90483
Fold 12 has AUC = 0.90806
Fold 13 has AUC = 0.90164
Fold 14 has AUC = 0.90284
この結果はカーネルからそのまま持ってきました。
(PCが重すぎてfold1までしか動かなかった・・・)
# DISPLAY NN VALIDATION AUC
val_auc = metrics.roc_auc_score(y, oof_preds_NN)
print('NN_CV = OOF_AUC =', round( val_auc,5) )
print('Mean_AUC =', round( np.mean(all_auc_NN),5) )
# PLOT NN TEST PREDICTIONS
plt.hist(test_preds_NN,bins=100)
plt.title('NN test.csv predictions')
plt.show()
SVMで512のモデルを作る
SVMだと0.928をだしてくれる。
train = pd.read_csv('train.csv')
test = pd.read_csv('test.csv')
cols = [c for c in train.columns if c not in ['id', 'target', 'wheezy-copper-turtle-magic']]
from sklearn.svm import SVC
from sklearn.feature_selection import VarianceThreshold
from sklearn.model_selection import StratifiedKFold
from sklearn.metrics import roc_auc_score
# INITIALIZE VARIABLES
oof_preds_SVM = np.zeros(len(train))
test_preds_SVM = np.zeros(len(test))
# BUILD 512 SEPARATE MODELS
for i in range(512):
# ONLY TRAIN/PREDICT WHERE WHEEZY-MAGIC EQUALS I
train2 = train[train['wheezy-copper-turtle-magic']==i]
test2 = test[test['wheezy-copper-turtle-magic']==i]
idx1 = train2.index; idx2 = test2.index
train2.reset_index(drop=True,inplace=True)
# FEATURE SELECTION (USE SUBSET OF 255 FEATURES)
sel = VarianceThreshold(threshold=1.5).fit(train2[cols])
train3 = sel.transform(train2[cols])
test3 = sel.transform(test2[cols])
# STRATIFIED K FOLD
skf = StratifiedKFold(n_splits=11, random_state=42)
for train_index, test_index in skf.split(train3, train2['target']):
# MODEL WITH SUPPORT VECTOR MACHINE
clf = SVC(probability=True,kernel='poly',degree=4,gamma='auto')
clf.fit(train3[train_index,:],train2.loc[train_index]['target'])
oof_preds_SVM[idx1[test_index]] = clf.predict_proba(train3[test_index,:])[:,1]
test_preds_SVM[idx2] += clf.predict_proba(test3)[:,1] / skf.n_splits
#if i%10==0: print(i)
# DISPLAY SVM VALIDATION AUC
val_auc = roc_auc_score(train['target'],oof_preds_SVM)
print('SVM_CV = OOF_AUC =',round(val_auc,5))
SVM_CV = OOF_AUC = 0.92624
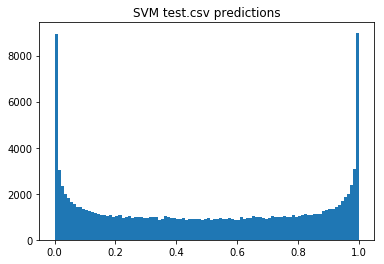
# PLOT SVM TEST PREDICTIONS
plt.hist(test_preds_SVM,bins=100)
plt.title('SVM test.csv predictions')
plt.show()
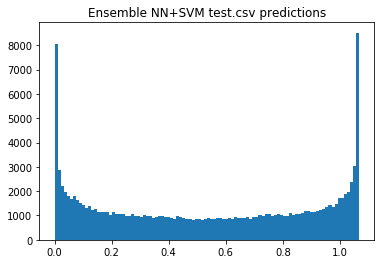
SVMとNNで計算した確率を足しあわせると
# DISPLAY ENSEMBLE VALIDATION AUC
val_auc = roc_auc_score(train['target'],oof_preds_SVM+oof_preds_NN)
print('Ensemble_NN+SVM_CV = OOF_AUC =',round(val_auc,5))
Ensemble_NN+SVM_CV = OOF_AUC = 0.91937
# PLOT ENSEMBLE TEST PREDICTIONS
plt.hist(test_preds_SVM+test_preds_NN,bins=100)
plt.title('Ensemble NN+SVM test.csv predictions')
plt.show()
より分離できていそう??
確率が1,0以外の部分が小さくなっている。
プライベートデータセットの推定
ダウンロードしたtest.csvはみんなに配られているものと同じ。
このコードをローカルで走らせたり、kaggleのカーネルで走らせたらtest.csvが読み込まれる。
256変数512種類のカテゴリカル変数、ラベル1行の合計(256512+1)行です。
kaggleにカーネルを提出したら、このコードは二回読み込まれるでしょう。
その時、データセットが512*512行のフルデータセットとなります。
我々のコードが二回目読み込まれたときには、新しいsubmission.csvの結果がリーダーボードに表示されることになります。
(前提として、最初のsubmission.csvはスコアになりません。二回目のsubmissionだけです。)
このコードは、フルデータセットを与えられたときにsubmission.csvを作ります??
我々はデータセットの構造を推定することができるでしょう。リーダーボードを通して、submission.csvが変化していることが分かるから。
さらに、testデータをプライベートのものとパブリックの配布されているものに分けることもできる。
idの列は偽物であることがここで話されている。
idはMD5でつくられた暗号である
リンク先のディスカッションを簡単に翻訳すると、
trainデータのidの一つ目は0train、二つ目は1train、三つめは2train
という文字をMD5で変換したものであるということ。
testも0testとかを変換しただけ。
IDがこれだけ適当に決められていることが分かれば、IDにあまり深い意味はないのかもしれないとわかりそう。
linerさんとyirunさんはよくこれを見つけられたなぁ
したがって、idのカラムをインデックスに入れて、プライベートとパブリックのデータがどう違うのかを推定することができます。
import hashlib
# CREATE LIST PUBLIC DATASET IDS
public_ids = []
for i in range(256*512+1):
st = str(i)+"test"
public_ids.append( hashlib.md5(st.encode()).hexdigest() )
# DISPLAY FIRST 5 GENERATED
public_ids[:5]
['1c13f2701648e0b0d46d8a2a5a131a53',
'ba88c155ba898fc8b5099893036ef205',
'7cbab5cea99169139e7e6d8ff74ebb77',
'ca820ad57809f62eb7b4d13f5d4371a0',
'7baaf361537fbd8a1aaa2c97a6d4ccc7']
testの中のidはMD5でハッシュ化されたものであることを確認
# DISPLAY FIRST 5 ACTUAL
test['id'].head()
0 1c13f2701648e0b0d46d8a2a5a131a53
1 ba88c155ba898fc8b5099893036ef205
2 7cbab5cea99169139e7e6d8ff74ebb77
3 ca820ad57809f62eb7b4d13f5d4371a0
4 7baaf361537fbd8a1aaa2c97a6d4ccc7
Name: id, dtype: object
testの本当のidと比較しても相違はないので、
やはりMD5で変換されていることが分かった。
# SEPARATE PUBLIC AND PRIVATE DATASETS
public = test[ test['id'].isin(public_ids) ].copy()
private = test[ ~test.index.isin(public.index) ].copy()
プライベートデータセットの構造を特定する
プライベートデータセットの構造を決める
このリンクで説明した通り、trainとtestには、512個のカテゴリカルデータの塊があるデータセットのように見えます。
各データには256個の変数があります。
つまり、データのブロックは512*216です(ヒートマップで見たやつ。ひとつのセルが131072個ある。これがtestもtrainもある。)
データには1~3.75の標準偏差があって、標準偏差1の群は役に立たないので捨てて、3.75が使えるものになります。
プライベートデータセットも同じ構造なのでしょうか?
# DETERMINE TRAIN DATASET STRUCTURE
useful_train = np.zeros((256,512))
for i in range(512):
partial = train[ train['wheezy-copper-turtle-magic']==i ]
useful_train[:,i] = np.std(partial.iloc[:,1:-1], axis=0)
useful_train = useful_train > 1.5
# DETERMINE PUBLIC TEST DATASET STRUCTURE
useful_public = np.zeros((256,512))
for i in range(512):
partial = public[ public['wheezy-copper-turtle-magic']==i ]
useful_public[:,i] = np.std(partial.iloc[:,1:], axis=0)
useful_public = useful_public > 1.5
# DETERMINE PRIVATE TEST DATASET STRUCTURE
useful_private = np.zeros((256,512))
for i in range(512):
partial = private[ private['wheezy-copper-turtle-magic']==i ]
useful_private[:,i] = np.std(partial.iloc[:,1:], axis=0)
useful_private = useful_private > 1.5
if np.allclose(useful_train,useful_public):
print('Public dataset has the SAME structure as train')
else:
print('Public dataset DOES NOT HAVE the same structure as train')
結果
Public dataset has the SAME structure as train
使える変数と使えない変数を1.5で分けてみたけど、
testも1.5で判定したら、配列が等価であった。
だからプライベートデータセットも構造としては同じものである。
リーダーボードを見てプライベートデータセットを考える
プライベートデータセットの構造が、trainやtestと異なる場合、すべての予測はゼロを入れることにします。
結果は0.5になるでしょう。
もしプライベートデータセットがtrainやtestと同じ構造であれば、0.95を提出できます
カーネルを実行すると、プライベートデータセットがないので、すべてゼロでsubmissionします。
でもこれをkaggleに送信すると、kaggleにはプライベートデータセットがあるので合格します。
sub = pd.read_csv('sample_submission.csv')
if np.allclose(useful_train,useful_public) & np.allclose(useful_train,useful_private):
print('We are submitting TRUE predictions for LB 0.950')
sub['target'] = (test_preds_NN + test_preds_SVM) / 2.0
else:
print('We are submitting ALL ZERO predictions for LB 0.500')
sub['target'] = np.zeros( len(sub) )
sub.to_csv('submission.csv',index=False)
結果
We are submitting ALL ZERO predictions for LB 0.500
これがリーダーボードでは0.5ではないのでプライベートデータセットが使われていることがわかる。
まとめ
これを提出するとカーネルが0.95を獲得したことがわかります。
kaggleの評価手順で二回目実行時にプライベートデータセットにアクセスして、
プライベートデータセットがトレーニングデータセットと同じデータ構造を持っていることを確認できます。
さらに、この構造を変更し、512のモデルでなく、単一の高スコアを出すNNをつくることもしました。
以上
次回へ続く