バイオ領域のトレンドを知りたい
初めに
大学にて生物学を専攻しており、データサイエンスは全くの無知からスタートしたのですが練習、備忘録として興味ある範囲からトレンドを分析しどのような結果が得られるのか知りたくなったので実際に行ってみました。
参考:https://qiita.com/takubb/items/e5578a8143a4f6b0f7fc
概要
この記事では、初めにGoogle Trendでの基本の検索データ取得、可視化を行う。
その後検索ワードを複数に増やし各検索ワードでの相関係数やトレンドを抽出します。
最後に国別でのトレンド比較も行います。
結果の見方や改善方法などについては、また別の記事にて記載する予定です(データに意味を持たせるのってむずかしい、、)
環境
私はバイオインフォマティクス関連の解析も扱うので
os :windows10
エディター:VScode
サーバー :Ubuntu
言語 :python(conda環境)
みたいな感じで行っていますが、今回の分析はgooglecolabで完結できるものなので特に必要なものはありません。
ただし、次の項目記載のモジュールのインポートまえに各種ダウンロードは必要です。(ほとんどpipで済んでしまうので省略)
モジュールのインポート
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
import statsmodels.api as sm
from pytrends.request import TrendReq
import japanize_matplotlib
Pytrendsについて
Pytrendsを利用して、Google Trend APIへアクセス
公式サイト
https://pypi.org/project/pytrends/
TrendReqで接続言語とタイムゾーンを指定して、API接続し検索キーワードをリストで渡す
timeframeに期間をし、geoにエリア(国名を英2字)を指定する。データはpandas.DataFrame形でリターンされる
データ取得の基本
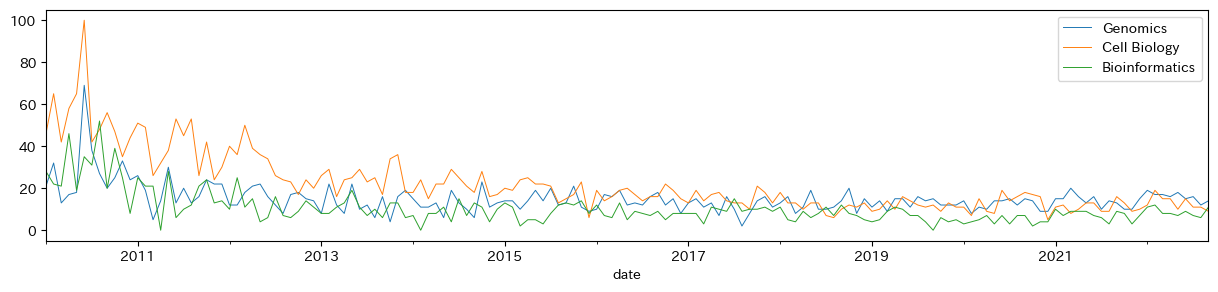
試しにゲノミクス、細胞生物学、生物情報学を検索ワードにデータを取得してみる。
(おすすめは最近流行ってるなーとか流行り終わったなーと感じる特定分野のキーワードだとよい結果が得られます)
# 検索する際の条件(国、タイムゾーン)を指定
pytrends = TrendReq(hl='ja-JP', tz=360)
kw_list = ["Genomics",'Cell Biology','Bioinformatics']
# リストに検索ワード、タイムフレームに時期、地域を指定
pytrends.build_payload(kw_list, timeframe='2010-01-01 2022-09-30', geo='JP')
# データフレームに格納しプロット
df = pytrends.interest_over_time()
df.plot(figsize=(15, 3), lw=.7)
ヒトゲノム計画(2010)のころに世間への認知からゲノミクスや生物情報学の関心が増えたのだろうか、、
ただ、最近の動向がどの分野もいまいちつかみずらい
検索ワードを増やし可視化
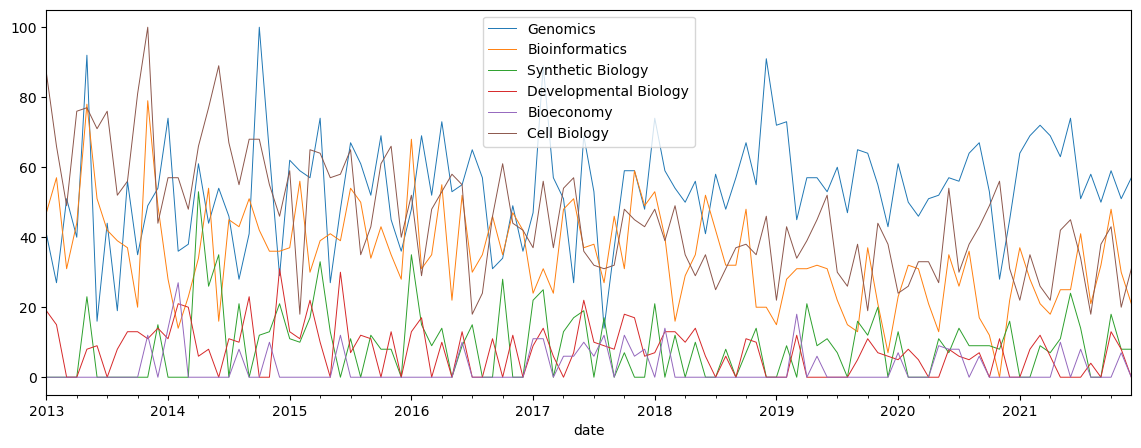
先ほど2010以降に全体として減少した結果であり、あまり有益なデータが得られなかったので、過去十年でワードを増やして可視化してみる。
# Pytrendsは三つ以上を同時に取得できないので分ける
kw_list1 = ["Genomics","Bioinformatics","Synthetic Biology"]
kw_list2 = ["Developmental Biology", "Bioeconomy", "Cell Biology"]
pytrends = TrendReq()
# 先ほどと同じように記述
pytrends.build_payload(kw_list1, timeframe='2013-01-01 2021-12-31', geo='JP')
# isPartialといういらないカラムができるので消しておく
df1 = pytrends.interest_over_time().drop('isPartial',axis=1)
pytrends.build_payload(kw_list2, timeframe='2013-01-01 2021-12-31', geo='JP')
df2 = pytrends.interest_over_time().drop('isPartial',axis=1)
# df1とdf2を結合して表示
df = pd.concat([df1, df2], axis=1)
df.plot(figsize=(14, 5), lw=.7)
データが多いと見にくくなってしまったので、アクセス数を算出し見てみる
グラフのコードについてはpandasのグラフ作成を自分で調べて理解するのをお勧めします。
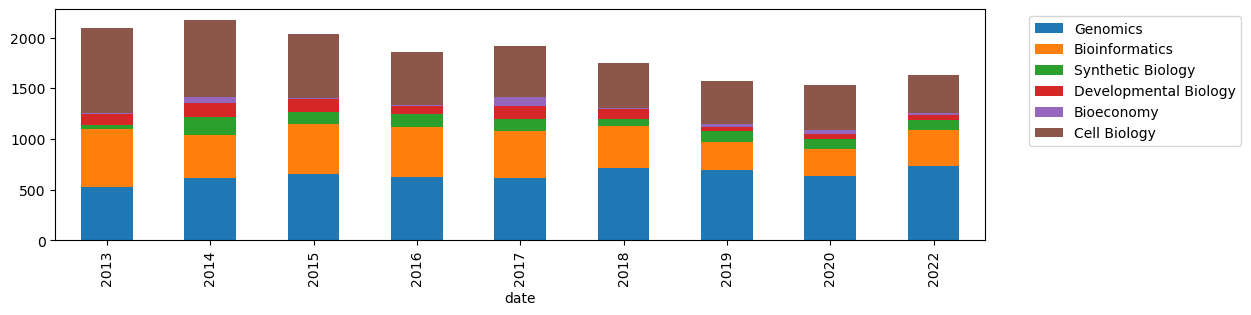
yearly = df.resample('Y').sum().plot.bar(stacked=True, figsize=(12, 3))
plt.xticks(np.arange(9), ('2013','2014','2015','2016','2017','2018','2019','2020','2022'))
plt.legend(bbox_to_anchor=(1.04,1), loc="upper left")
plt.show()
全体としてアクセス数が減っているのがわかったが、ほかの検索ワードもあまり変動がなかったので、より細かく解析してみる。
時系列データ分析
トレンド抽出
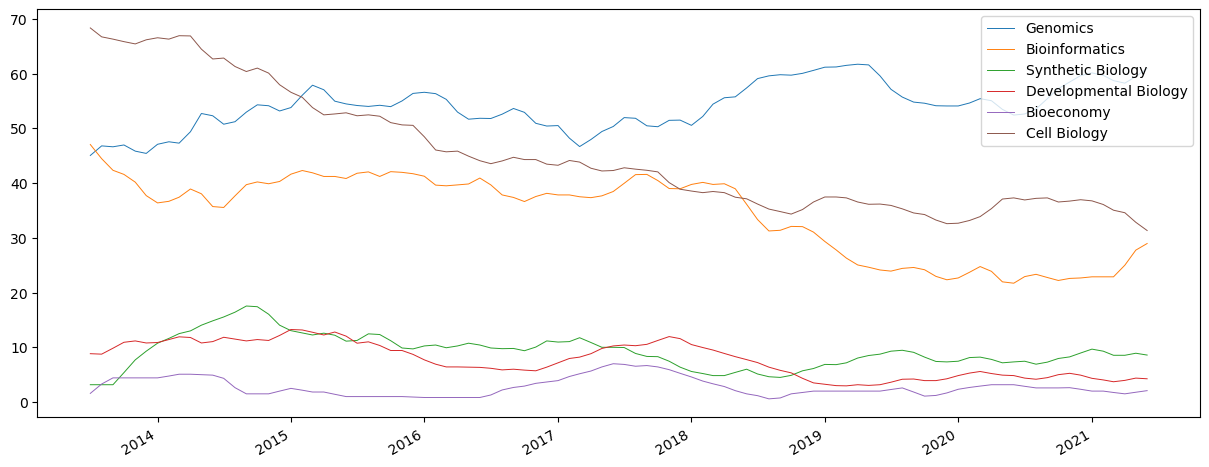
季節性と周期性があるので、Seasonal decompositionを施して、トレンドを出力してみる
seasonality = pd.DataFrame()
# 一列ごとにトレンドを抽出した値を入れていく
for i in df.columns:
res = sm.tsa.seasonal_decompose(df[i])
seasonality = pd.concat([seasonality, res.trend], axis=1)
seasonality.columns = ["Genomics","Bioinformatics","Synthetic Biology","Developmental Biology", "Bioeconomy", "Cell Biology"]
seasonality.plot(figsize=(15, 6), lw=.7)
相関係数
resid_list = []
for i in df.columns:
res = sm.tsa.seasonal_decompose(df[i])
resid_list.append(res.resid)
resid = pd.concat(resid_list, axis=1)
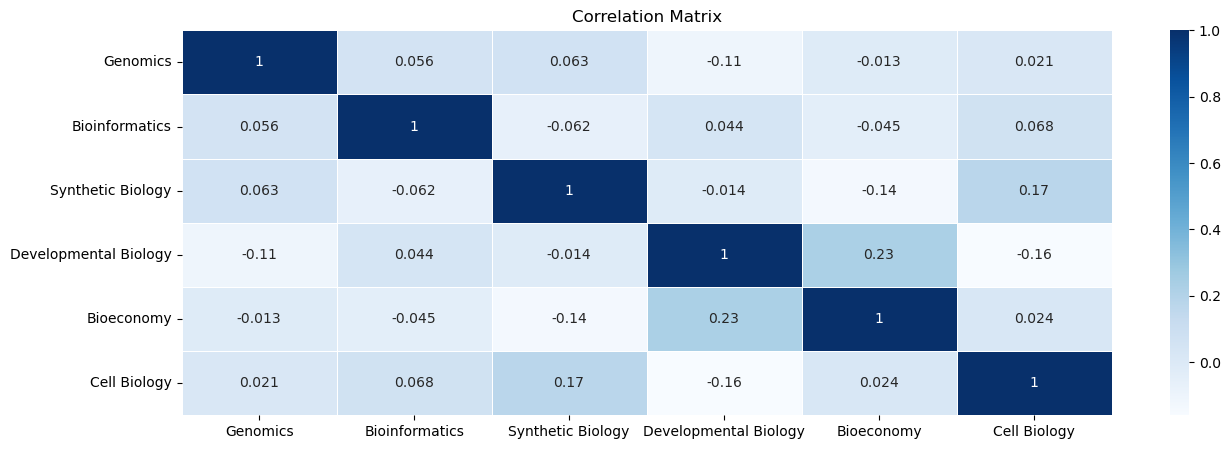
cor_matrix = resid.corr()
cor_matrix.columns = ["Genomics","Bioinformatics","Synthetic Biology","Developmental Biology", "Bioeconomy", "Cell Biology"]
cor_matrix.index = ["Genomics","Bioinformatics","Synthetic Biology","Developmental Biology", "Bioeconomy", "Cell Biology"]
fig = plt.figure(figsize=(15, 5))
ax = fig.add_subplot(111)
sns.heatmap(cor_matrix, annot=True, lw=0.7, cmap='Blues', ax=ax)
ax.set_title('Correlation Matrix')
plt.show()
相関はあまり見られなかったが、バイオインフォマティクスとバイオエコノミーが減少している中、ゲノミクスは大きく上昇しているのがわかった。
国別でのトレンド比較
最後に上昇しているゲノミクスと減少しているバイオインフォマティクスのキーワード検索傾向に関する国別の違い調べてみる
- 日本
- U.S
- イギリス
- シンガポール
- オーストラリア
# 先ほどまでのコードと違う点としては国コードをリスト化しループ処理によって処理しています。
data = pd.DataFrame()
trg_area = ['JP','US','GB','SG','AU']
pytrends = TrendReq()
for kw in kw_list:
df = pd.DataFrame()
for area in trg_area:
pytrends.build_payload([kw], timeframe='2018-01-01 2022-12-31', geo=area)
df_ = pytrends.interest_over_time()
df = pd.concat([df, df_], axis=1)
data = pd.concat([data, df], axis=1)
data.drop('isPartial', axis=1, inplace=True)
genomics = data[kw_list[0]]
bioinfo = data[kw_list[2]]
genomics.columns = trg_area
bioinfo.columns = trg_area
# 以下プロット用
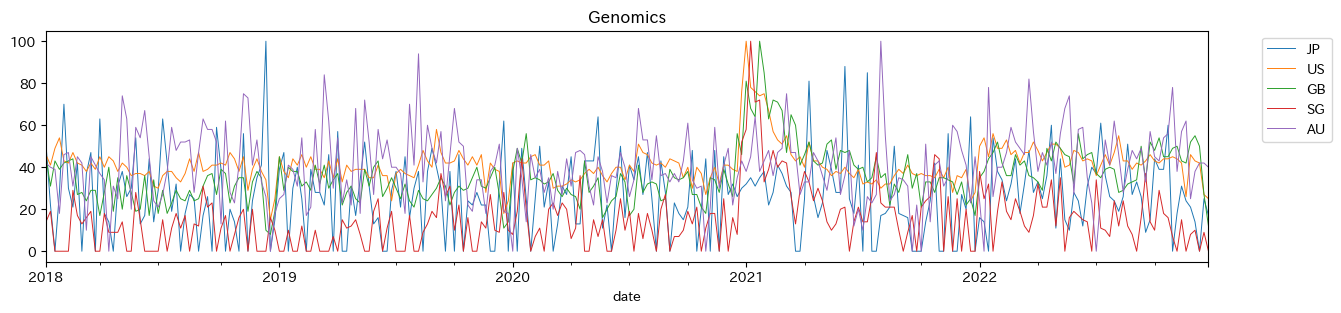
genomics.plot(figsize=(15, 3), lw=.7)
plt.legend(bbox_to_anchor=(1.04,1), loc="upper left")
plt.title('Genomics')
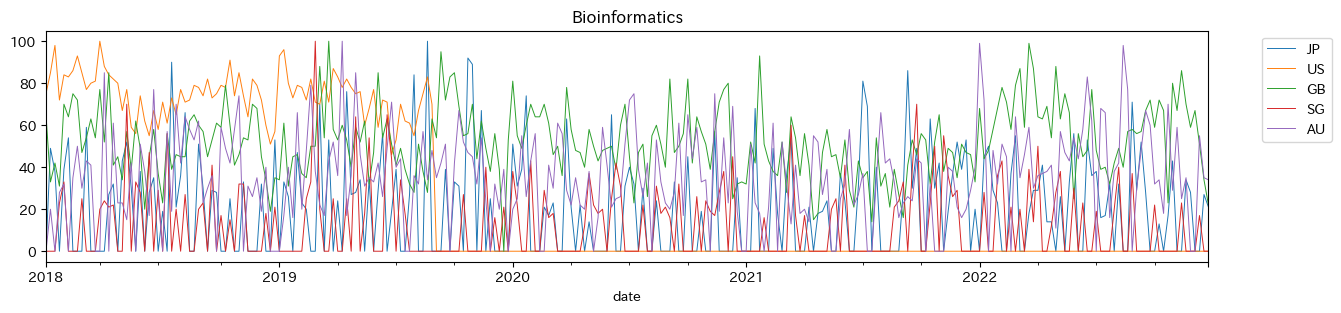
bioinfo.plot(figsize=(15, 3), lw=.7)
plt.legend(bbox_to_anchor=(1.04,1), loc="upper left")
plt.title('Bioinformatics')
もちろん検索数をただグラフにしただけでは傾向はつかめないので先ほど同様にトレンド抽出を行う。
アメリカのデータが取得できていないので原因と改善の必要がありますが、とりあえず練習用なのでそのままやっちゃいます
trend = pd.DataFrame()
for i in range(len(trg_area)):
res = sm.tsa.seasonal_decompose(genomics.iloc[:,i])
trend = pd.concat([trend, res.trend], axis=1)
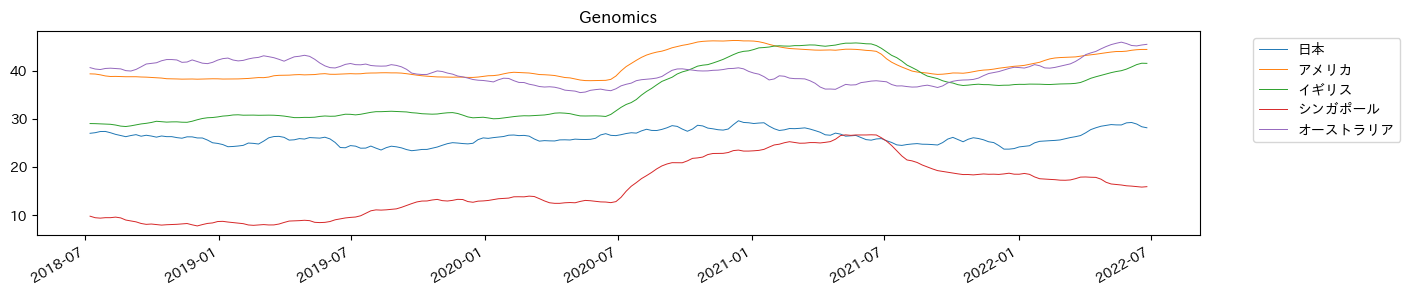
trend.columns = ['日本','アメリカ','イギリス','シンガポール','オーストラリア']
trend.plot(figsize=(15, 3), lw=.7)
plt.legend(bbox_to_anchor=(1.04,1), loc="upper left")
plt.title('Genomics')
2020年7月からシンガポール、イギリス、アメリカで上昇しているがわかる。
これは、この月にコロナウイルスが正式に分類・命名された論文の発表月であることもあり、コロナ関連のゲノミクスが注目を受けたと考えられた。
まとめ・考察
今回の分析結果は練習用であり、目的をそこまで明確に決めていなかったためトレンドから目新しい傾向変化などは分析できなかったが、
初めのアクセス数データだけでは見れない情報をつかむことができるトレンドの技術等を今後活用していけると感じた。