0. 初めに
1. 自己紹介
しがない大学生のあめふりてる*と申します。主に、HSP3と補助としてC/C++を使用しています。
競技プログラミングをほんの少しだけ嗜んでいます。現時点ではAtCoder緑です。楽しいので皆さんもやりましょう。
2. 今回の記事を書く経緯
今回は、こちらの記事に感銘を受け、私も「ムダにクリエイティブな"Hello World"」に挑戦してみようと思い、遺伝的アルゴリズムを使うことにしました。
もともと、HSP3で遺伝的アルゴリズムを使ってみたいという思いもあり、せっかくだから、ついでに記事にもしてみようと筆をとった次第です。
3. 記事を書くにあたり
せっかく記事にするのですから、コードだけをポンと載せて終わりというのはなんだか味気ないと思います。そこで、遺伝的アルゴリズムの概要についても触れてから本題に入ろうと思います。
ざっくりなんとなく伝わってくれるだけで私としては十分嬉しい限りです。よかったら、コメントやいいねをください。小躍りして喜びます。
1. 遺伝的アルゴリズム
遺伝的アルゴリズムについては、そんなに詳しい話は私もわからないので詳細はGoogle先生に聞くのが正確に情報を得られると思います。ここでは、本当にざっくり適当に説明していきたいと思います。
1. 遺伝的アルゴリズムとは
まず、遺伝的アルゴリズムとは何ぞやという話からしていきたいと思います。
遺伝的アルゴリズムとは、生物の進化を模倣した進化的計算手法の一つです。こう聞くととても難しく聞こえますが、考え方はとても身近なものです。
自然界において、生物はより強い子供を作ろうと日々努力しています。そのために、より強い個体同士が交配するような仕組みになっています。生物でいえば、戦いが強かったり、模様が綺麗だったたりする個体はモテます。皆さんも、美少年・美少女を見かけたり、年収が高い人が現れたりしたら好きになりますでしょ?そんな感じです。
この「強い個体同士が交配する」「強い子供を作る」という仕組みを真似したのが遺伝的アルゴリズムです。ここがわかれば、遺伝的アルゴリズムの概要の半分は分かったといっても過言でないように思います。
2. 遺伝的アルゴリズムの流れ
さて、遺伝的アルゴリズムの基本的な流れを見ていきましょう。以下の6つの手順に従って遺伝的アルゴリズムは構成されています。
- 初期遺伝子たちを作る
- それらを評価する
- 優れた個体を残し、劣った個体を捨てる
- 優れた個体同士を交配させる
- 出来た遺伝子の一部を突然変異させる
- 2へ戻る
これだけで、どんどん強い個体になっていくのは見ていて楽しいものがあります。以上の流れは、コードを見ていく際に実際に確認していきましょう。
3. 遺伝的アルゴリズムを使ってみよう(OneMax問題)
さて、ここまで遺伝的アルゴリズムの概要を説明してきましたが、実際どのようなコードで動くのかピンとこない方もいると思います。
なので、一問だけ遺伝的アルゴリズムを使ってとある問題を解いてみましょう。今回取り扱う問題は、「OneMax問題」です。この項で書くことが、本題の基本的なことになります。「そんなのより早く"Hello, World!"が見たい」という方は、飛ばして第2章に進んでください。
1. OneMax問題とは
OneMax問題とは何かというと、0と1のみからなる数列の和を最大化する問題のことです。言い換えると、数列の各要素が1にする問題のことです。
0と1からなる数列とは、例えば、[0, 1, 1, 0, 1]のような数列です。これを[1, 1, 1, 1, 1]にするのが最終目標です。
これを遺伝的アルゴリズムで解いていきましょう。
2. HSPによる実装
さて、早速ですがコードを見ていきましょう。
# module _GA_
//評価関数
#defcfunc ScoreCalc array gen, int iter, int len
ret = 0
i = 0
repeat len
ret += gen(iter, i);
i++
loop
return ret
//ソート
#deffunc SortScore array score, array gen, int gen_num, int len
//選択ソート
i = 0
repeat gen_num - 1
min = i
j = i + 1
repeat
if(j >= gen_num):break;
if(score(j) > score(min)):min = j;
j++
loopQ
tmp = score(i):score(i) = score(min):score(min) = tmp
j = 0
repeat len
tmp = gen(i, j):gen(i, j) = gen(min, j):gen(min, j) = tmp
j++
loop
i++
loop
return
# global
randomize
screen 0, 960, 540, 0, 0, 0
wait 500
//初期化
len = 100//遺伝子長
gen_num = 100//個体数
parents_num = gen_num / 2//残す親の個数
childlen_num = gen_num - parents_num//作る子供の個数
dim gen, gen_num, len//各遺伝子の配列
dim child_gen, gen_num, len//次世代の遺伝子の配列
dim score, gen_num//各遺伝子のスコアの配列
//初期遺伝子生成
i = 0
repeat gen_num
j = 0
repeat len
gen(i, j) = 0
j++
loop
i++
loop
//メイン
repeat
//スコア計算//個体評価
i = 0
repeat gen_num
score(i) = ScoreCalc(gen, i, len)
i++
loop
//ソート
SortScore score, gen, gen_num, len
//描画
redraw 0
color 255, 255, 255:boxf:color 0,0,0
pos ,0
i = 0
repeat gen_num
pos 0,
j = 0
repeat len
mes gen(i, j), 1
j++\
loop
mes " " + score(i)
i++
loop
redraw 1
//交叉 + 突然変異
//優れた親を半分保存
i = 0
repeat parents_num
j = 0
repeat len
child_gen(i, j) = gen(i, j)
j++
loop
i++
loop
//子を生成
k = 0
repeat childlen_num / 2
//子を二点交叉で生成
j = 0
repeat len
child_gen(i, j) = gen(i, j)
child_gen(i + 1, j) = gen(i + 1, j)
j++
loop
left = rnd(len):right = rnd(len)
if(left > right):tmp = left:left = right:right = tmp
j = left
repeat
if(j > right):break
child_gen(i, j) = gen(k + 1, j)
child_gen(i + 1, j) = gen(k, j)
j++
loop
i += 2
k += 2
loop
//突然変異
i = parents_num
repeat childlen_num
j = 0
repeat len
p = rnd(len)
if(p == 0){
if(child_gen(i, j) == 0){
child_gen(i, j) = 1
}else{
child_gen(i, j) = 0
}
}
j++
loop
i++
loop
//世代交代
i = 0
repeat gen_num
j = 0
repeat len
gen(i, j) = child_gen(i, j)
j++
loop
i++
loop
await
loop
以上が、HSPでの実装の一例です。すごく長くて読みづらいコードになっていますが、少しづつ見ていけばなんてことはなく、ただただ遺伝的アルゴリズムの流れに沿ったコードであることがわかると思います。そのことを次の項で見ていきましょう。
3. コードの解説
さてこの項では、前の項で出したHSPのコードを分解してみていきます。上のコードを読んで理解できる方は飛ばしていただいて構いません。
今回は、わかりやすさを重視して、あまり専門用語や難しい言葉を使わないように解説していくつもりです。が、読みやすさの点から用語を使う場合は随時ちょっとした解説を挟みます。
では、見ていきましょう。
0. おさらい
ここで一瞬だけ、遺伝的アルゴリズムの流れをおさらいします。
- 初期遺伝子たちを作る
- それらを評価する
- 優れた個体を残し、劣った個体を捨てる
- 優れた個体同士を交配させる
- 出来た遺伝子の一部を突然変異させる
- 2へ戻る
これを踏まえてみていきましょう。
1. 初期遺伝子たちを作る
初期遺伝子を作っているのはこの部分です。
//初期遺伝子生成
i = 0
repeat gen_num
j = 0
repeat len
gen(i, j) = 0
j++
loop
i++
loop
ここで使っている変数を見ていきます。
・gen_num : 作る数列の数
・len : 各数列の長さ
・gen : 数列の各要素
以上でわかる通り、ここでの遺伝子とは「数列」のことです。どんな遺伝的アルゴリズムであろうと、遺伝子は「数列」で表されます。このことは次章でも使うので頭の隅に置いておいてください。
ここでは、すべての要素が0な長さlenの数列をgen_num個作っているということです。
2. 遺伝子たちを評価する
いよいよ、ここから、遺伝的アルゴリズムらしくなっていきます。
遺伝子をどう評価するかは、遺伝的アルゴリズムで最も重要なことの一つです。評価方法が変わるだけで、進化の過程や結果が大きく変わっていきます。
ちなみに、評価するために使う式や関数などを「評価関数」と呼んだりします。
今回OneMax問題においての評価関数は、「数列の要素の合計値」になります。これは、目標である「合計値を最大にする」を考えれば当然でしょう。
コードではこんな風に書かれています。
//評価関数
# defcfunc ScoreCalc array ans, array gen, int iter, int len
ret = 0
i = 0
repeat len
ret += gen(iter, i);
i++
loop
return ret
//スコア計算//個体評価
i = 0
repeat gen_num
score(i) = ScoreCalc(ans, gen, i, len)
i++
loop
ここで使っている変数と関数の説明です。
・score : 各数列の評価値
・ScoreCalc : 評価関数。ここでは、各数列の合計値を出力する。
3. 優れた個体を残し、劣った個体を捨てる
各遺伝子の優劣は先ほど評価関数で評価しました。優れた遺伝子のみを選びたいので、scoreを基準にソートします。それが、
//ソート
SortScore score, gen, gen_num, len
の部分です。
余談ですが、SortScoreは、選択ソートという方法でソートしていますが、あまり速度の出ないソートになっています。本当ならクイックソートを使いたいのですが、コードが長くなりすぎて分かりづらいので今回は選択ソートで妥協しました。より早く解にたどり着くには速いアルゴリズムを使うのがとても重要です。
話を戻します。
優れた個体を残すとは、具体的には次の世代に遺伝子をそのまま転写します。そうすることで、今ある優れた個体を超える個体が出てくるまで優れた個体は残り続けるため、次世代において前の世代よりも評価値が下がることがなくなり、より早く解にたどり着くことができます。
このコードでそれを行っています。
//優れた親を半分保存
i = 0
repeat parents_num
j = 0
repeat len
child_gen(i, j) = gen(i, j)
j++
loop
i++
loop
ここで変数の説明をします。
・parents_num : 次の世代に残す優秀な親の数
・child_gen : 次の世代の遺伝子
4. 優れた個体同士を交配させる
さて、遺伝的アルゴリズムで最重要な項目です。これが遺伝的アルゴリズムの特徴です。
優れた個体同士を交配させてより優れた個体を生み出すのが遺伝的アルゴリズムの基本的な考え方でした。
さて、今回は「二点交叉」という方法で交配を行いました。
下の図を見てください。
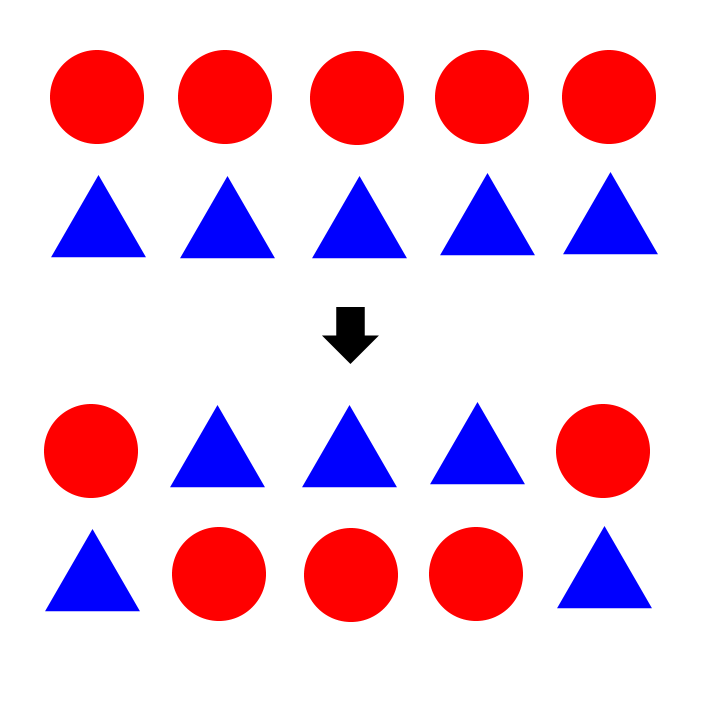
ざっくり、こんな感じで交配します。ある二点を選んで、その二点で挟まれた要素を入れ替えるのが二点交叉です。
これを、コードに落とすとこんな感じになります。
//子を生成
k = 0
repeat childlen_num / 2
//子を二点交叉で生成
j = 0
repeat len
child_gen(i, j) = gen(i, j)
child_gen(i + 1, j) = gen(i + 1, j)
j++
loop
left = rnd(len):right = rnd(len)
if(left > right):tmp = left:left = right:right = tmp
j = left
repeat
if(j > right):break
child_gen(i, j) = gen(k + 1, j)
child_gen(i + 1, j) = gen(k, j)
j++
loop
i += 2
k += 2
loop
ここで変数の説明をします
・childlen_num : 生成する子供の数
・left-right : 遺伝子を交換する範囲を示す
親二つに対して、子は二つ生成されます。
5. 出来た遺伝子の一部を突然変異させる
さて、突然変異させるとありますが、突然変異とは何ぞやという話からしましょう。
ここでの突然変異とは、遺伝子の一部を違う要素へ変えることを言います。
わかりづらいので例を出すと
もともと、
[0, 1, 1, 0, 1]
という遺伝子を持った個体に突然変異起こすと
[0, 1, 1, 1, 1]
という個体になったりします。
突然変異は、位置も数値もランダムで行います。(OneMax問題においては位置のみランダムです。)
では、なぜ突然変異が必要なのかという話は、この章の最後にコラムとして書きたいと思います。
とりあえずは、突然変異っていうのを起こしたほうがいいんだなぁ程度でいいです。
では、コードを見てみましょう。
//突然変異
i = parents_num
repeat childlen_num
j = 0
repeat len
p = rnd(len)
if(p == 0){
if(child_gen(i, j) == 0){
child_gen(i, j) = 1
}else{
child_gen(i, j) = 0
}
}
j++
loop
i++
loop
見て分かる通り、突然変異は、一個体に一回起こるぐらいの確率にしています。
ここの確率が高いと、なかなか解にたどり着けなくなり、逆に低くても解にたどり着けなくなります。
ほどほどの確率にするのが突然変異においては肝となります。
さて、ここまで来たら世代交代して、2に戻るだけです。
お疲れさまでした。これで大体の流れはつかんでもらえたかなと思います。
コラム. 遺伝的アルゴリズムは近親交配である
さて、コラムと称して遺伝的アルゴリズムについて少しだけ私の考えや、印象について語りたいと思います。ここで、さっきの「突然変異がなぜ必要か」という話も軽くします。
コラムのタイトル通り、私の遺伝的アルゴリズムの最初の印象は、「これってつまり、近親交配ってことじゃん」でした。遺伝的アルゴリズムでは親と子供、子供と子供同士が交配することが往々にして存在します。これすなわち、近親交配です。
自然界において、近親交配はあまり良い影響を与えません。劣性な遺伝が色濃く子供に受け継がれるため遺伝的疾患が起こりやすくなり、病気に弱くなったり、先天的な障害を引き起こす可能性があります。劣性の遺伝子の話は中学の教科書に載っている「メンデルの遺伝の法則」を参照するとよいと思います。
さて、遺伝的アルゴリズムは「生物進化の模倣」でした。そのため、この近親交配を行い続けるこの方法では劣性遺伝が強くなる一方です。そこで登場するのが「突然変異」です。劣性な遺伝子のある部分が「たまたま」優性な遺伝子になると優性遺伝はその後残り続けます。すると、より良い解にたどり着くことができるのです。これが、「突然変異」が必要な理由になります。
まあ、少し話を盛りながら話しましたが、少しでもなるほどと思っていただけたら幸いです。
2. ムダにクリエイティブな"Hello,World!" -前編-
さて、長々と遺伝的アルゴリズムについて書いてきましたが、いよいよ本題です。
遺伝的アルゴリズムを使って、"Hello,World!"をしていきます。が、このコードは上記のOneMax問題を少しだけいじるだけなのでコードすべてについての解説はしません。
OneMax問題のコードから変わった点についてだけをかいつまんで解説していきます。
1. さっそくコード
本当にさっそくですがコードを見ていきましょう。
# module _GA_
#defcfunc ScoreCalc str ans, str gen, int len
string1 = ans;
string2 = gen;
ret = 0
i = 0
repeat len
if(peek(string1, i) != peek(string2, i)):ret++;
i++
loop
return ret
#deffunc SortScore array score, array gen, int gen_num, int len
//選択ソート
i = 0
repeat gen_num - 1
maxi = i
j = i + 1
repeat
if(j >= gen_num):break;
if(score(j) < score(maxi)):maxi = j;
j++
loop
tmp = score(i):score(i) = score(maxi):score(maxi) = tmp
tmp = gen(i):gen(i) = gen(maxi):gen(maxi) = tmp
i++
loop
return
# global
randomize
screen 0, 800, 1000, 0, 0, 0
//初期化
//答え生成
gen_num = 100//個体数
parents_num = gen_num / 2//残す親の個数
childlen_num = gen_num - parents_num//作る子供の個数
ans = "Hello,World!"
len = strlen(ans)
sdim gen, len, gen_num
sdim child_gen, len, gen_num
dim score, gen_num
//初期遺伝子生成
i = 0
repeat gen_num
j = 0
repeat len
poke gen(i), j, rnd(256)
j++
loop
i++
loop
//メイン
font "", 16
repeat
//スコア計算//個体評価
i = 0
repeat gen_num
score(i) = ScoreCalc(ans, gen(i), len)
i++
loop
//ソート
SortScore score, gen, gen_num, len
//ソート後の遺伝子
redraw 0
color 255, 255, 255:boxf:color 0,0,0:pos ,0
i = 0
repeat gen_num
pos 0,
mes gen(i) + " " + score(i)
i++
loop
redraw 1
//交叉 + 突然変異
//親を半分保存
i = 0
repeat parents_num
child_gen(i) = gen(i)
i++
loop
//子を生成
k = 0
repeat childlen_num
//子を二点交叉で生成
child_gen(i) = gen(k)
child_gen(i + 1) = gen(k + 1)
left = rnd(len):right = rnd(len)
if(left > right):tmp = left:left = right:right = tmp
j = left
repeat
if(j > right):break
poke child_gen(i), j, peek(gen(k + 1), j)
poke child_gen(i + 1), j, peek(gen(k), j)
j++
loop
i += 2
k += 2
loop
//突然変異
i = parents_num
repeat childlen_num
j = 0
repeat len
p = rnd(len)
if(p == 0){
poke child_gen(i), j, rnd(256)
}
j++
loop
i++
loop
//世代交代
i = 0
repeat gen_num
gen(i) = child_gen(i)
i++
loop
await
loop
見て分かる通り、とてもよく似ていることがわかります。大きく変わった点は一つだけ。それは評価関数です。それについては次項で説明します。
1. 評価関数について
さて、評価関数は大きく変わりました。一体どのように変わったのか見ていきます。
評価関数を考えるにあたり最終目標を見てみます。
OneMax問題の最終目標は何だったかというと「数列の合計が最大になること」でした。なので評価関数もそのままです。
では、"Hello,World!"においての最終目標は何かというと「各文字の位置と種類が同じであること」です。このような「距離」を「ハミング距離」といい、この距離が0であれば同じ文字列であるといえます。
つまり、今回は「ハミング距離を0にする」ことが最終目標となります。
さて、では評価関数はどうなるかというと、ハミング距離を測るだけになります。非常に簡単ですね。
2. こまごまとした変更点
1. ソート
OneMax問題では降順でソートしていましたが、今回は評価値が小さいほうが優れた個体となるので昇順でソートします。
2. 文字型
OneMax問題では、遺伝子の要素は0と1しかありませんでしたが、今回は文字型なので0~255までの計256種類あります。
そのため、遺伝子の初期化や、突然変異でrnd(256)を多用することになります。
3. peek-poke命令の使用
今回は、文字列を遺伝子として扱うため、一文字の取り出しや置き換えはpeek-poke命令を使用します。HSPの厄介なところです。
ここで注意すべきなのはpoke命令です。確保したメモリより先を書き換えることができるこの命令は、メモリの破壊やメモリリークのもとになります。今回は、超えないように設定しているので安心ですが、3章で紹介するコードにおいてとても苦労したので注意してください。
3. ムダにクリエイティブな"Hello,World!" -後編-
第2章では、OneMax問題のコードを少し変えるだけでできる簡単バージョンの"Hello,World!"でした。
しかし、「ムダにクリエイティブ」にはまだ遠いことがあります。
それは、空文字列から作ってない点です。上のコードでは、長さを変更することができず、初期遺伝子も12文字と固定されていました。これでは「クリエイティブ」とは呼べないと私は強く思いました。
そこで強化版として、空文字列から"Hello,World!"するコードを書きました。
それがこれです。
# module _min_max_
#defcfunc min int a, int b
aa = a:bb = b;
if(aa < bb):return aa:else:return bb
#defcfunc max int a, int b
aa = a:bb = b;
if(aa > bb):return aa:else:return bb
# global
# module _LevenshteinDistance_
#defcfunc LevenshteinDistance str a, str b
aa = a
bb = b
alen = strlen(aa)
blen = strlen(bb)
dim dp, alen + 10, blen + 10
i = 0
repeat alen + 1
dp.i.0 = i
i++
loop
i = 0
repeat blen + 1
dp.0.i = i
i++
loop
i = 1
repeat alen
j = 1
repeat blen
if(peek(aa, i - 1) == peek(bb, j - 1)){
cost = 0
}else{
cost = 1
}
dp(i, j) = min(min(dp(i - 1, j) + 1, dp(i, j - 1) + 1), dp(i - 1, j - 1) + cost)
//pos i * 16, j * 16 : mes dp(i, j)
j++
loop
i++
loop
return dp(alen, blen)
# global
# module _GA_
#defcfunc ScoreCalc str ans, str gen
anslen = strlen(ans) : genlen = strlen(gen)
ret = 0
ret += (max(anslen, genlen) - min(anslen, genlen)) * 10
aa = ans : gg = gen
ret += max(anslen, genlen) * 2
i = 0
repeat min(anslen, genlen)
if(peek(aa, i) == peek(gg, i)):ret--
i++
loop
i = min(anslen, genlen) - 1
repeat min(anslen, genlen)
if(peek(aa, i) == peek(gg, i)):ret--
i--
loop
ret += LevenshteinDistance(ans, gen)
return ret
#deffunc SortScore array score, array gen, int gen_num, int len
//選択ソート
i = 0
repeat gen_num - 1
maxi = i
j = i + 1
repeat
if(j >= gen_num):break;
if(score(j) < score(maxi)):maxi = j;
j++
loop
tmp = score(i):score(i) = score(maxi):score(maxi) = tmp
tmp = gen(i):gen(i) = gen(maxi):gen(maxi) = tmp
i++
loop
return
# global
randomize
screen 0, 1000, 1000, 0, 0, 0
wait 500
//変数初期化
gen_num = 1024
ans = "Hello,World!"
len = strlen(ans)
sdim gen, 1000, gen_num
sdim child_gen, 1000, gen_num
dim score, gen_num
dim genlen, gen_num
//初期遺伝子生成
i = 0
repeat gen_num
gen(i) = ""
i++
loop
//スコア初期化
i = 0
repeat gen_num
score(i) = 0
i++
loop
//メイン
font "", 12
repeat
//スコア計算//個体評価
i = 0
repeat gen_num
score(i) = ScoreCalc(ans, gen(i))
genlen(i) = strlen(gen(i))
i++
loop
//ソート
SortScore score, gen, gen_num, len
//描画
redraw 0
color 255, 255, 255:boxf:color 0,0,0:pos ,0
i = 0
repeat gen_num
pos 0,
mes gen(i) + " " + score(i)
i++
loop
redraw 1
//完了してたら、break
if(gen(0) == ans):wait:break
//交叉
parents_num = min(gen_num / 2, 1000)
childlen_num = gen_num - parents_num
//親を半分保存
i = 0
repeat parents_num
child_gen(i) = gen(i)
i++
loop
//子を生成
k = 0
repeat childlen_num / 2
//子を二点交叉で生成
child_gen(i) = gen(k)
child_gen(i + 1) = gen(k + 1)
if(genlen(k) == genlen(k + 1)){//遺伝子長が同じとき
if(genlen(k) != 0){
left = rnd(genlen(k)):right = rnd(genlen(k))
if(left > right):tmp = left:left = right:right = tmp
j = left
repeat
if(j > right):break
poke child_gen(i), j, peek(gen(k + 1), j)
poke child_gen(i + 1), j, peek(gen(k), j)
j++
loop
}
}else{//遺伝子長が違うとき
if(genlen(k) != 0 && genlen(k + 1) != 0){//ゼロじゃないときは大丈夫
if(genlen(k) > genlen(k + 1)){//genlen(i + 1)のほうが長くなるように
tmp = child_gen(i) : child_gen(i) = child_gen(i + 1) : child_gen(i + 1) = tmp
tmp = genlen(k) : genlen(k) = genlen(k + 1) : genlen(k + 1) = tmp
tmp = gen(k) : gen(k) = gen(k + 1) : gen(k + 1) = tmp
}
left = rnd(genlen(k)):right = rnd(genlen(k)):iter = rnd(genlen(k + 1) - genlen(k))
if(left > right):tmp = left:left = right:right = tmp
j = left
repeat
if(j > right):break
poke child_gen(i), j, peek(gen(k + 1), j + iter)
poke child_gen(i + 1), j + iter, peek(gen(k), j)
j++
loop
}
}
i += 2
k += 2
loop
//突然変異
i = parents_num
repeat childlen_num
//変更
j = 0
repeat genlen(i)
p = rnd(genlen(i))
if(p == 0){
poke child_gen(i), j, rnd(256)
}
j++
loop
//削除
buf = child_gen(i)
child_gen(i) = ""
j = 0
repeat genlen(i)
p = rnd(genlen(i))
if(p != 0){
child_gen(i) += strf("%c", peek(buf, j))
}
j++
loop
//追加
buf = child_gen(i)
child_gen(i) = ""
j = 0
p = rnd(genlen(i) + 1)
if(p == 0){
child_gen(i) += strf("%c", rnd(256))
}
repeat genlen(i)
child_gen(i) += strf("%c", peek(buf, j))
p = rnd(genlen(i) + 1)
if(p == 0){
child_gen(i) += strf("%c", rnd(256))
}
j++
loop
i++
loop
//世代交代
i = 0
repeat gen_num
gen(i) = child_gen(i)
i++
loop
await
loop
cls
font "", 32
mes "完了!"
mes gen(0)
実は、このコード。2章で使ったコードとほとんど形は一緒なのです。
パッと見ただけではそうは感じ取れませんが、主な変更点は三つです。
一つ目は評価関数。二つ目は遺伝子交配。三つ目は突然変異です。
この三つについて簡単にどこが変わったのか見ていくことにします。
1. 評価関数
まずは、評価関数についてみていきましょう。
文字列の距離を測るという方針は、第2章と同じです。距離といっても様々なものがあげられます。
まず、長さの違いを距離として扱うもの。
次に、2章で取り上げたハミング距離。
最後に、編集距離として有名なレーベンシュタイン距離。
この三つを評価関数に入れています。
この中で今回、最も重きを置いているのは、長さの差です。長さがそろわないとまず文字列が同じになることはあり得ません。
そのため、距離の重さを他の倍にしています。
2. 遺伝子交配
遺伝子交配についても大きく変更されています。
何が変更されたかですが、違う長さの遺伝子同士を交配させることができるようにしました。
ただそれだけですが、非常に重要で厄介な変更でした。
3. 突然変異
突然変異についても、遺伝子交配と同じように違う長さの遺伝子に対応させるべく変更しました。
どう変更したかですが、もともと文字を変更することだけだったものに「文字の削除」「文字の追加」を加えました。
ここで注意すべきなのは、poke命令をなるべく使わず、文字列の足し算を使うことです。こうすることでメモリの破壊やメモリリークをなくします。
以上の点を変更することで、空文字列からHello,World!を生成し出力することに成功しました。
一応、動画を見せたいのでTwitterのリンクを貼っておきます。
https://twitter.com/Amefuri_Tell/status/1287052096412332032?s=20
完成させたときのTwitterの投稿です。いいねください。
4.おわりに
ここまで読んでくださった方ありがとうございました!
これにて、HSP3で遺伝的アルゴリズムを使って「ムダにクリエイティブに」 "Hello,World!" を出力する挑戦はこれにて完結です!
本当は、OpenCLを使ってマルチコアの計算をさせる予定だったのですが、頭がパンクして出来なかったので、これは今度個別にしてみたいと思います。
完走した感想として、"Hello,World!"は原点にして頂点なんじゃないかと思い始めています。皆さんも「ムダにクリエイティブ」な"Hello,World!"を書いてみてはいかがでしょうか?
よろしければ、コメントなどいただけたら幸いです。
改めまして、最後まで読んでいただき、ありがとうございました!