広島大学で社会基盤(土木)を専攻している3年生です。
現在は1年間休学して建設系のIT企業でインターンをしています。
今回はSemantic SegmentationのUNetとPSPnetを脳腫瘍データセットで学習させ評価検証までしたので評価検証部分について書いていきます。
UNet: (https://qiita.com/1998922akito/items/c5d178f8108b629c6a12)
※ただしUNetの方はなるべく未検出が出ないように推論結果のマスクから脳腫瘍部分にする閾値を低く設定しているので出力が異なります。
PSPNet: (https://qiita.com/1998922akito/items/0bd1174bf0c5fd149b7f)
colab環境で学習させたもの記事があるので学習部分が気になる方はそっちを見てみてください。
今回使ったデータセットについて
kaggeで公開されている脳のMRIデータセット(https://www.kaggle.com/mateuszbuda/lgg-mri-segmentation) を使用しました。
MRI画像のうち2827枚をtrain data、552枚をvalidation data、550枚をtest dataとしています。
評価検証内容
・推論の脳腫瘍部分と教師データの脳腫瘍部分が一致している画像の平均IoU
・未検出数(教師データの腫瘍部分が全く検出されていない画像の枚数)
・誤検出数(推論の脳腫瘍部分が教師データと全くかぶっていない画像の枚数)
・Recall (上記の未検出数 / 脳腫瘍がある画像)
・Precision (上記の誤検出数 / 脳腫瘍がある画像)
を求めました。
この評価検証方法にした理由は脳腫瘍がある人を特定することを最優先したからです。
なので評価で欲しい数値の優先順位が上位から
・未検出数
・誤検出数
・IoU
となりました。
脳腫瘍部分を検出できたかどうか、どれくらい間違えて検出してしまったかを優先し、どれくらい領域が正確に検出できたかどうかは評価としてはあったほうがいいので脳腫瘍が検出できた画像のみで評価しました。
IoU, Recall, Precisionなどの言葉を初めて知った人はこちらの記事を参考にしてください。
(https://qiita.com/K5K/items/5da52e99861483cae876)
例としてあげられている内容が医療について上記の内容についても分かりやすいと思います。
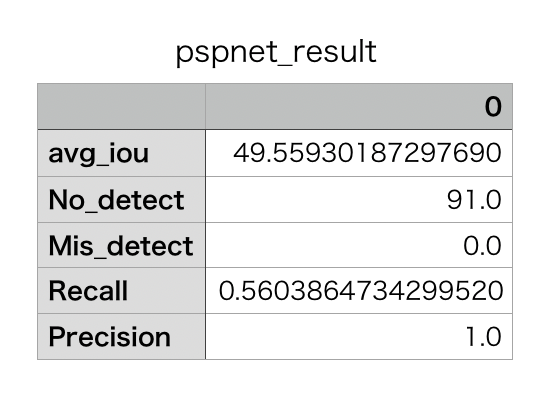
検証結果
550枚の脳腫瘍の評価検証結果
UNet
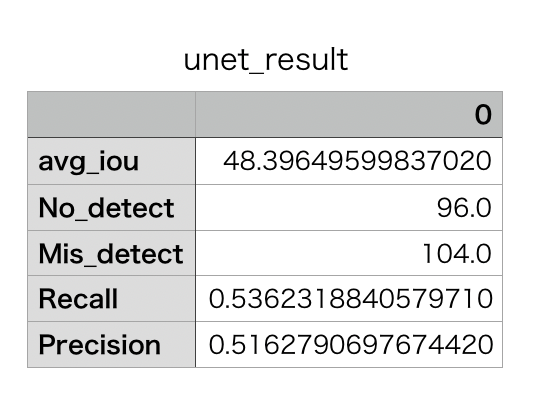
https://gyazo.com/1188ee803f9f26386e72a5d1bf5389cc
下記が評価内容との対応です。
avgiou: 推論の脳腫瘍部分と教師データの脳腫瘍部分が一致している画像の平均IoU
No_detect:(教師データの腫瘍部分が全く検出されていない画像の枚数)
Mis_detect: 誤検出数(推論の脳腫瘍部分が教師データと全くかぶっていない画像の枚数)
Recall: Recall (上記の未検出数 / 脳腫瘍がある画像)
Precision: Precision (上記の誤検出数 / 脳腫瘍がある画像)
PSPNetの特徴
・40%以上の数の脳腫瘍画像が検出できていない。
・誤検出はなくPrecision重視の学習になっている。
・検出できている領域もほとんどが小さめのため検出できた画像に絞っているにもかかわらずIoUは低め、、
UNetの特徴
・未検出を減らすために脳腫瘍とする領域を多くとる閾値にしているため可視化を行った時より未検出数は減っているためPSPNetとRecallの精度はあまり大差ない。
※PSPNetとRecallの値が同じくらいになるように自分が設定し他のでこの結果になってます。
・未検出を減らす処理をしたため誤検出数が多くなっている。可視化のときに設定した閾値ではPSPNet同様に誤検出はありませんでした。
・PSPNetよりも色の濃淡が少ない画像の検出に弱いため、色の濃淡で腫瘍を検出しやすい学習になっている。
・検出できた画像に関しては可視化を行った時の閾値だとおそらくPSPNetよりも精度が良いものが多いイメージ(ここはほとんど自分のイメージです、、)
評価検証コード
github URL(https://github.com/Akito-Fujihara/MRI-Brain-Eval-PSPNet-UNet)
に評価検証コードをあげました。
データセット、weightファイルなどはダウンロードして以下の構成にしてください。
MRI-Brain-Eval-PSPNet-UNet/
┣ kaggle_3m(kaggleからダウンロードしたデータセット※https://www.kaggle.com/mateuszbuda/lgg-mri-segmentation)
┣ PSPNet_eval.ipynb(PSPNet評価)
┣ UNet_eval.ipynb(UNet評価)
┣ pspnet_result.csv(PSPNetの全体の評価検証結果)
┣ pspnet_result_per_image.csv(PSPNetの画像ごとによる評価検証結果)
┣ unet_result.csv(UNetの全体の評価検証結果)
┣ unet_result_per_image.csv(UNetの画像ごとによる評価検証結
┣ utils
┃ ┣ init.py(このファイルが存在するディレクトリをパッケージとなる。)
┃ ┣ data_augumentation.py(前処理)
┃ ┣ dataloader.py(pytorchのdataloaderを作成)
┃ ┣ pspnet.py(モデル)
┃ ┣ pspnet256_show.py(可視化)
┃ ┣ unet.py(モデル)
┃ ┗ unet256_show.py(可視化)
┗weights(学習済みweightファイルを保存する URL: https://drive.google.com/drive/folders/1aM-4BSSu3I6F9wwQ0Vn3H_wsW6LvRhWY?usp=sharing)
まとめ
結果から今回の評価方法だとPSPNetの方が適していると思いました。
PSPNet: (https://qiita.com/1998922akito/items/0bd1174bf0c5fd149b7f)
で精度を上げるために試しておきたい内容を記しました。
追加として小さめに出力されるケースが多いのは教師データに脳腫瘍がない画像が多いためだと考えらるため、脳腫瘍がある教師データのみで学習させるといいと思いました。
また、時間があれば他のモデル(多分インスタンスセグメンテーションだけどMaskRCNNかDeepLab V3)なども試して評価してみたいと思います。