広島大学で社会基盤(土木)を専攻している3年生です。
現在は1年間休学して建設系のIT企業でインターンをしています。
今回はSemantic SegmentationのUNetを脳のMRI画像のデータセットを学習させ可視化まで行いました。
今回学習に使ったデータセットについて
kaggeで公開されている脳のMRIデータセット(https://www.kaggle.com/mateuszbuda/lgg-mri-segmentation )を使用しました。
UNetで脳の腫瘍を検出します。
以前はPSPNetで学習して可視化まで行いました。
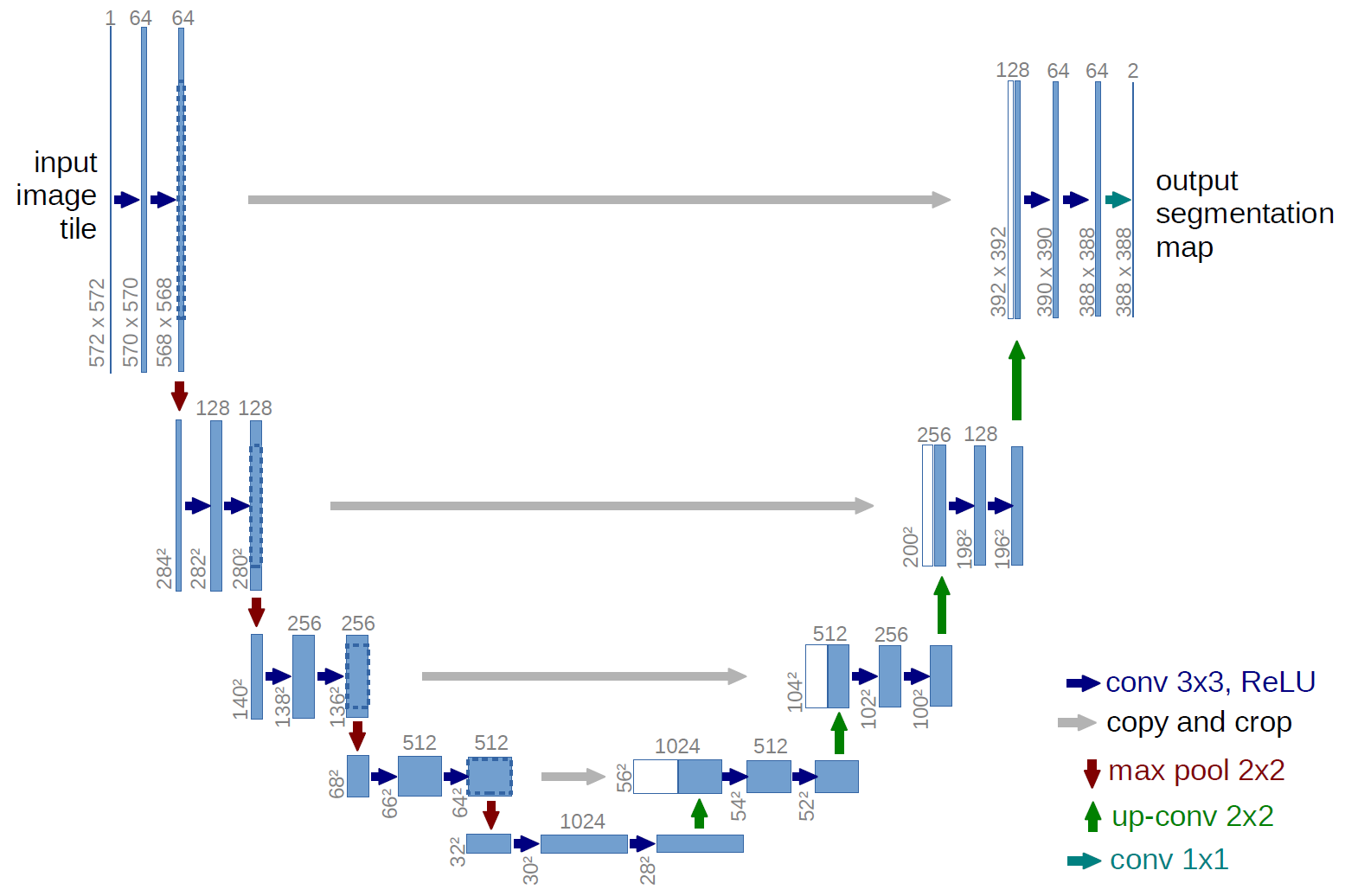
UNet とは
U-Netは2015年に生物医学的画像セグメンテーションのために作られたモデルです。
2014年に発表されたFCNがモデルのがベースとなっており、論文ではトレーニングのデータセットが少量でも正確なセグメンテーションができるとされている。
簡単にモデルの概要を説明すると
こちらが今回UNetについて調べた参考資料です。
https://arxiv.org/pdf/1505.04597.pdf
https://lib-arts.hatenablog.com/entry/paper15_U-Net
実装について
https://www.kaggle.com/arunmohan003/pytorch-brain-mri-segmentation-unet
実装についてはkaggleの公開されているnotebookを参考にしました!
UNetで実装している例が数個あるのでより理解を深めたい方は見てみると面白いかもしれません。
ファインチューニングしていないので少し学習時間がかかるかもしれません、、、
自分が実装したコードのgithubリンクがこちらです(https://github.com/Akito-Fujihara/MRI-Brain-Unet-model)
jupyter notebookとpyファイルを載せています。
データセット、weightファイルなどはダウンロードして以下の構成にしてください。
MRI-Brain-UNet256-model/
┣ kaggle_3m(kaggleからダウンロードしたデータセット※https://www.kaggle.com/mateuszbuda/lgg-mri-segmentation)
┣ log_output.csv(学習のlossを記録)
┣ UNet_test.ipynb(推論・可視化)
┣ UNet_train.ipynb(学習)
┣ result_image(推論結果を可視化した画像を保存するファイル)
┣ utils
┃ ┣ init.py(このファイルが存在するディレクトリをパッケージとなる。)
┃ ┣ data_augumentation.py(前処理)
┃ ┣ dataloader.py(pytorchのdataloaderを作成)
┃ ┣ unet.py(モデル)
┃ ┗unet256_show.py(可視化)
┗weights(weightファイルを保存する)
学習・推論結果と考察
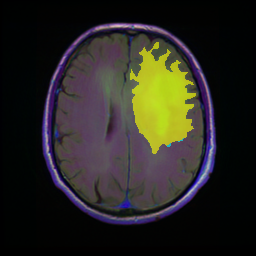
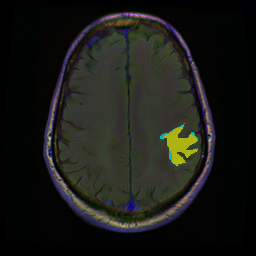
可視化した画像のは水色が教師データ、黄色が推論結果である。
上手く推論できている例
上手く推論できなかった例
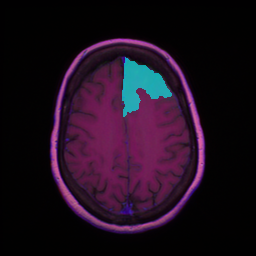
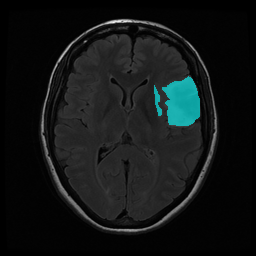
腫瘍部分と正常な部分の色の濃淡がはっきりしているところはかなり綺麗に検出することができました。濃淡が少ない腫瘍部分は以前試したPSPNet(https://qiita.com/1998922akito/items/0bd1174bf0c5fd149b7f )よりも検出できていない画像が多い印象でした。
今回のデータセットだと色の濃淡をはっきりさせる前処理などを勉強して取り入れると大幅に精度が改善しそうな感じがします。
また、UNetの論文ではdata augmentationを実装することで精度が大きく変わると書いてあるので様々な処理を加えてみたいと思います。