背景
以前の記事で、GTDBで提供されている系統樹をiTOLで開く方法を紹介しました。この系統樹は、10万近くの細菌種からなり、非常に重いです。種の系統樹を門(phylum)にまとめると、見やすくなります。この記事では、GTDBの系統樹からphylumを抽出し、iTOLで表示する方法を紹介します。
実行環境
- Windows11
- Ubuntu 22.04.3 LTS
- ete4 (以前の記事で環境構築を紹介)
入力ファイル
入力するGTDBの系統樹は、以前の記事によるbac120_to_itol_noboot.treeを想定しています。
サンプルプログラム
ete4を使用して、系統樹から門のみを抽出するプログラム例はこちらです。
実行の際はこのようなコマンド($ python prune_phylum.py > phylum.tree)を入力し、ファイル出力しました。
prune_phylum.py
from ete4 import PhyloTree
def prune_phylum(file_path,parser=1):
phylum_list = []
t = PhyloTree(open(file_path),parser=parser)
for node in t.traverse():
full_name = node.name
if full_name is None:
continue
if 'p__' in full_name:
# 以下のノード名に対応し、phylum(p__)を抽出
# p__Desulfobacterota_B|c__Binatia
print(full_name)
phylum = full_name.split("|")[0]
# ノード名はphylumに置き換える
node.name = phylum
phylum_list.append(phylum)
t.prune(phylum_list)
return t
if __name__ == '__main__':
t = prune_phylum('bac120_to_itol_noboot.tree',1)
# t.show()
print(t.write(parser=1))
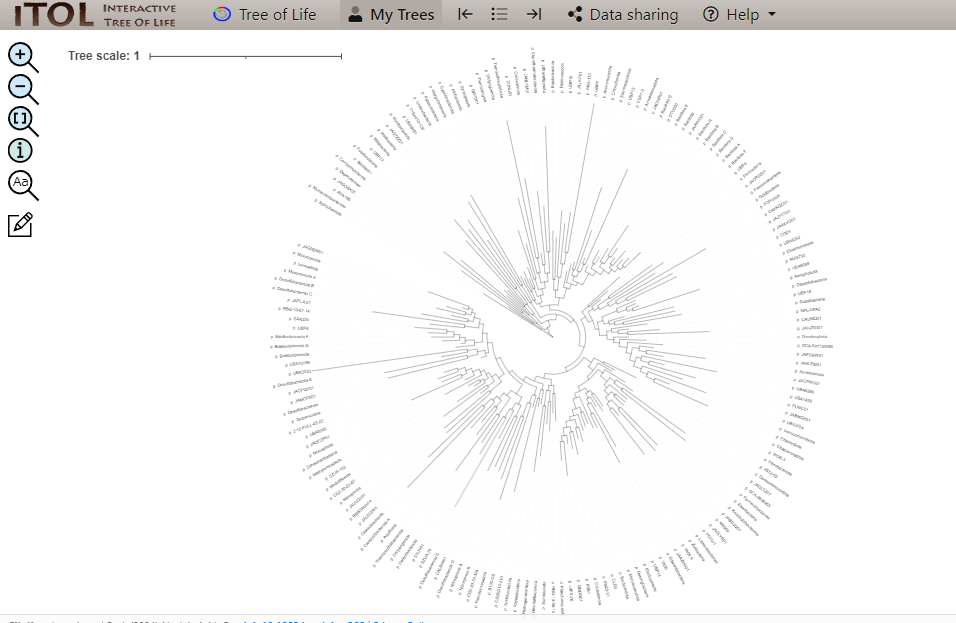
結果
まとめ
門のみを抽出した系統樹はAnnoTreeでも表示できますが、iTOLの方が自由度高く系統樹にアノテーションできると思っています(個人の意見です)。また、コードではコメントアウト(t.show())していますが、ete4でも系統樹を描画できます。この書き方についても紹介していきたいです。