(9/2/2014 追記:何故か後編の記事が削除されていましたので、分割後修正して再アップしました。)
はじめに
この記事は実際に手を動かし、コンピュータを使ってデータ可視化を行う人に向けて一般的なノウハウをお伝えする三回シリーズの第二回です。
わかりにくい可視化
昨夜寝る前に気づいたのですが、前回のプログラマ向けのニッチな記事をはてなブックマークのヘッドラインで見かけて驚きました。そしてその中に鋭いコメントを発見しました:
この手のグラフ系の可視化で本当に知見が得られたの?って思ってしまうな。わかりにくい。
これはまさにその通りで、これこそ私がこの記事をまとめようと思った理由の一つです。身も蓋もない事実を申し上げますと、分かりにくいどころか、ほぼ何の意味もないグラフ可視化がこの世には無数に存在します。結局各種ツールが発展して可視化が手軽にできるようになったと言うことは、それだけ簡単に大量のジャンクダイアグラムを作成できてしまうということにほかならないのです。Excelが普及して簡単なチャートがすぐに作れるようになった一方、ミスリーディングなチャートや酷い3Dグラフなどが量産されたのと理由は全く同じです。それなのに我々のような可視化ツールを研究のためのインフラとして作っている組織に、アメリカの政府機関や大手製薬会社が長きに渡りまとまった資金を投下してくれる理由は何なのでしょう?この理由について考えるのは「データ可視化で実際に手を動かす人々が効果的な可視化手順を追求する」と言うこの記事のテーマにぴったりで、とても意義深い議論だと思うので、予定を変更して中編を追加しそこを考察してみます。
なぜ分かりにくいのか?
Cytoscapeによって描かれる図は、node-link diagramと呼ばれます。ノードを線(エッジ)で繋ぎあわせて、様々なレイアウトアルゴリズムを使って配置すると言う、可視化の手法としてはとてもポピュラーなものです:
小規模なデータの場合、もしくは元のグラフが特殊な構造を持っている場合(例えば木)各ノードの関係性を直感的に理解できるので、何の工夫もない可視化でもそれなりに有効です。しかしこの手法には大きな問題点があります。
手法の限界と適切な手法の選択
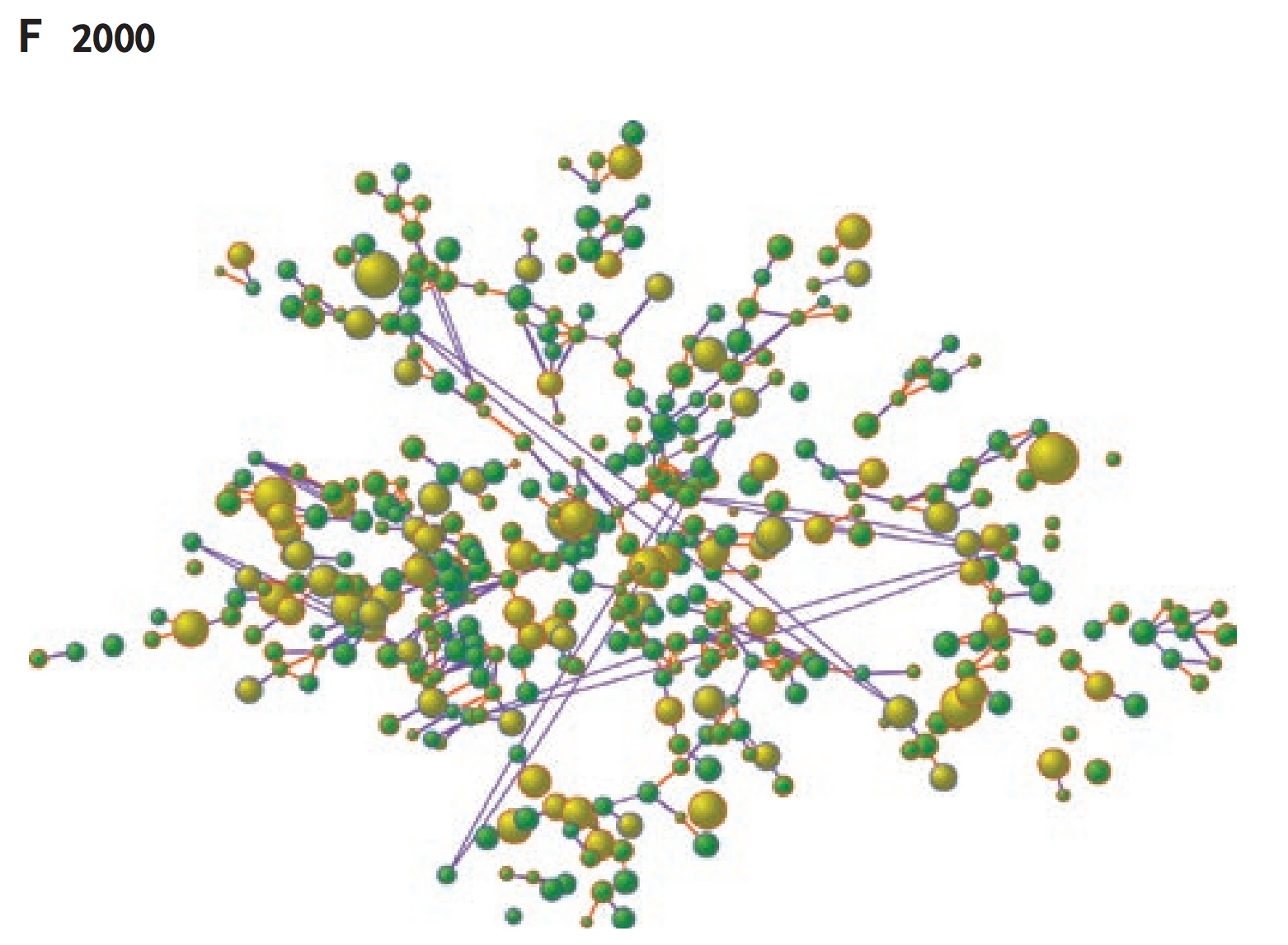
実はこの手法、巨大なグラフデータに対してスケールしません。このままGPUの進化が進めば、実装方法によっては数百万のノードとエッジを軽々と描画し、巨大な銀河の如きグラフデータをリアルタイムにレンダリングすることも可能になるでしょう。にもかかわらずスケールしないのです。これはどういうことでしょう?
上の2つは、以前私が「分野外の人に研究を説明するので、なんか派手なイメージを作ってくれ」と言うリクエストを受けて、Cytoscapeである生物のインタラクトームを描画したものです。これでもノード数は数千のオーダーです。数百万、数億のオーダーのグラフというものがどうなるかは想像がつくと思います。このような可視化に(イベントポスターや雑誌のカバーに使う以上の)意味はあるのでしょうか…?
テーマ無き可視化からの脱却
巨大ネットワーク描画はいつも無意味なのでしょうか?「ソーシャルネットワークのグラフデータがあるからとりあえず描画してみよう」と言うのは誰しも思いつくグラフ可視化ソフトウェアの使い方です。しかし最終的にそこから新たな知見を得られるレベルの可視化に仕上げるのはそれほど簡単ではありません。多くの場合に問題なのは、ただ漫然と巨大データを可視化したからというより、その前に明確なテーマを設定しなかったことだと思います。
メンロパークにあるFacebookの本社を訪れたことがある方は見たことがあると思いますが、ドリンクなどが置いてあるラウンジに、大量のタッチスクリーンを組み合わせた身長を超える大きさのディスプレイ・アレイが置いてあり、そこでFacebookに関する様々なデータを可視化しています(撮影禁止だったので画像はありませんが、検索すればいっぱい出てくると思います)。いわゆるデジタルサイネージに属するものですが、描画の元になっているのは実データであり、一種のデータ可視化です。恐らくあのディスプレイ・アレイで走っているアプリケーションと同じチームの仕事だと思いますが、FBはそのグラフデータ可視化を公開したことがあります:
これも巨大ネットワークのnode-link diagramですが、個人的にはこれが全く無意味だとは思いません。その理由はいくつかあります:
- ノードの位置が地球上の実際の場所に基づいていて、(x, y)ポジションそのものに意味がある
- サブグラフの濃度が、世界中の大都市を図らずも出現させている
- 視覚的に局所構造の濃度が見える
- 非常に密な米国とヨーロッパ
- 日本の隣に存在する巨大な空白地帯、もしくはGreat Firewallの存在
この可視化を作成した方の記事に、こんな一文があります:
I was interested in seeing how geography and political borders affected where people lived relative to their friends. I wanted a visualization that would show which cities had a lot of friendships between them.
(私は地理的・政治的な境界線がどのように人々の交友関係に影響をあたえるのかを見てみたいと思いました。どの都市の住民がたくさんの(密な)交友関係を持つのかという可視化が欲しかったのです。)
つまり、__疑問からスタートし、その疑問に対する回答を得る手段として可視化を用いている__のです。彼のゴールが完全に達成されたかどうかは皆さんの判断に委ねますが、少なくとも一定程度は成功していると思います。大切なのは「データがあるから可視化しよう」ではなく、「データのこの側面が見たいからこの手法で可視化しよう」という、手法の限界を知った上での選択です。
手法のスケーラビリティ
可視化のスケーラビリティには2つの意味があります。
- 計算量が大きすぎて、一般的なワークステーションでは処理しきれず描画できない
- 描画はできるが描画結果から有益な情報がほとんど読み取れない
私がここで議論しているのは2番目の問題です。上のような大きなグラフの一括描画から読み取れるものとは何でしょう?恐らく下記のような情報は何とか読み取れると思います:
- グラフの大雑把な大きさ。「少なくとも数千」程度の予想値
- グラフのおおよその密度、つまり単位ノードあたりのエッジの数
しかしこれらは、なにも重い描画をしてGPUのファンを回さなくても他に手法があります。
一つの手法の限界を超える
シンプルな手法で分かりやすい可視化を作成できないのならば、他の手法を検討したり併用するのが賢いやり方です。ネットワーク解析に限らず、最初にプロトタイプを作ってみた結果良くわからない可視化ができてしまった場合、そこから前に進むためには以下の方法があります:
- 各種統計量を利用する - 統計解析
- 特定のデータセットに使える他の手法を検討する - 描画手法の切り替え/併用
- 全く新しい可視化手法を考える - 可視化手法そのものの開発
- インタラクティブなアプリケーションに仕上げる - アプリケーションとしてのリリース
分かりにくい可視化の壁を超えるため、これらを一つ一つ検討してみましょう。
各種統計量を利用する
Node-link diagramがうまく問題に使えないならば、他の手法も検討します。前回の記事に「手法の選択」と言うステップを入れたのはこれも理由の一つです。Node-link diagramは言ってみれば__データポイントから画像へのone-to-oneマッピング__です。とてもシンプルですが、万能ではありません。それがうまく行かないのならば、可視化のテーマを補強するデータのエッセンスを抽出することを考えればいいのです。
例: グラフ密度
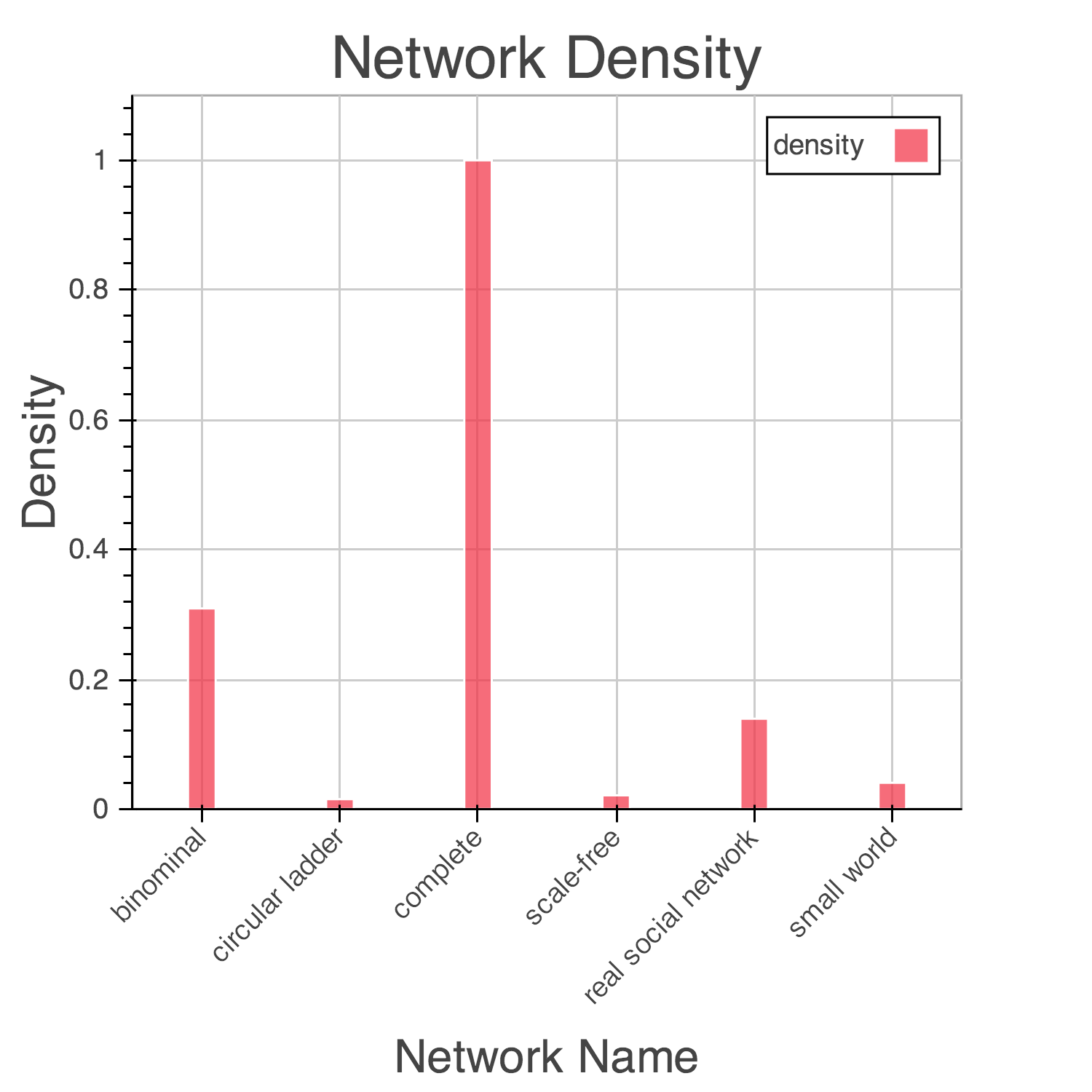
グラフの特性を知るには、ネットワーク図以外にもたくさんの方法があります。最もシンプルな指標の一つである密度(density)はただの数値ですから、複数の巨大グラフの特性を比較したいような場合、ただのバーチャートで間に合ってしまいます:
(ちなみにこのチャート、理想的なものではありません。「各グラフの密度の差を示す」と言うゴールなので、僅かな値の差はそれほど重要ではないため、縦横のグリッドは無くしたほうがスッキリしていいですね。)
上のチャートは、NetworkXでアルゴリズミックに生成したグラフ(と一つの実データ)の密度比較です。当然ですが、完全グラフの密度は1で、スケールフリーネットワークはとても小さな密度を持つことが読み取れます。非常に原始的ですが、可視化のテーマがネットワークの接続状態を知る、というものであれば、これだけでも充分テーマをサポートするデータ可視化です。
あまり深く立ち入るとデータ可視化ではなくグラフ解析の話題になってしまうのでここでは述べませんが、グラフ/ノード/エッジの性質を調べる統計量は他にもたくさんあります。
- 次数分布
- 情報中心性(重み付きエッジの場合)
- PageRank (有向グラフの場合)
これらの値を一般的なチャートにプロットするだけでもネットワークの性質に関する様々な情報が得られます。当然ですが、これらの指標の計算もリニアな計算量のものばかりではありませんので、グラフが巨大になればかなりの時間がかかってしまう場合もあります。それでも出力される結果はチャート化しやすいものが多いので、ネットワークの性質を理解すると言うゴールには有効です。ただしここで注意すべきは、闇雲に統計値を計算したとしても、それが自らのテーマを補強/検証するのに無意味な値ならば、結局はむやみに可視化を行うことと何も変わり無いということです。あるデータセットに対し、どのような統計処理をすればそのデータセットのどのような性質が分かるのかを理解しなければこの手段を上手く使うことは出来ません。
統計によってデータの複雑性を整理しそれを可視化に活かすという方法は、データ解析の専門家が使うPrincipal Component Analysis (PCA)などの考え方がアナロジーとして適切かもしれません。つまり安直な、生のデータプロットが意味のある可視化にならない場合、元のデータに統計処理を施し、データの元々の性質を失わずに意味のある知識を抽出すると言う方針です。統計の世界は巨大すぎてとてもここでは扱えませんし、私も専門家ではありませんので他の専門書(家)に譲りますが、可視化分野に関わる限り、ある程度は理解しないと限界に突き当たると思います。
描画手法の切り替えと併用
あるデータタイプに対して利用できる可視化手法は一つではありません。Node-link diagramのような一般的な描画手法以外にも、特定の構造を持つネットワークに対して使える便利な手法もあります。データがその基準に適合する場合、別の描画手法を検討するのもいいアイデアです。
階層構造を持つデータに対するTreemap
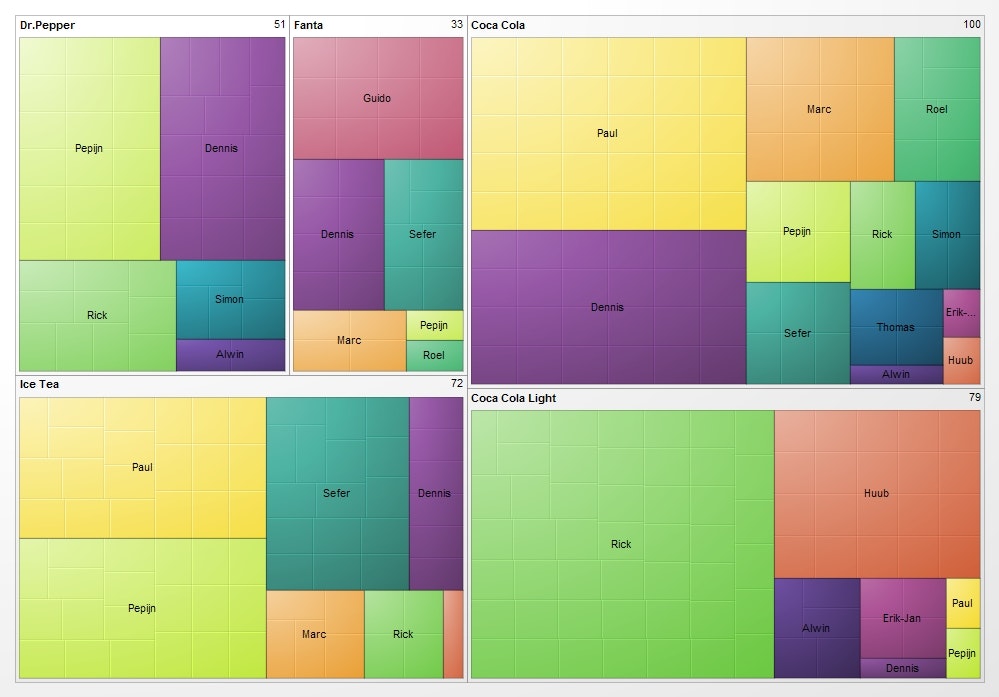
Gradient grouped treemap by User:GBoshouwers
現実世界に現れるデータには、全くのランダムなネットワーク以外にも、階層構造を持つものが散見されます。こういった場合、Treemap (Javaのあれではなく、可視化手法の名前です)が便利です。これは、長方形でデータ全体を表し、その中で各下部構造がどのような大きさを占めているのか、それはどのようなさらなる下部構造を持っているのか、と言うのをタイル状の入れ子構造で表したものです。D3.jsでも標準でサポートされている手法で、以下は同一データのTreemap表現と、node-link diagramです:

Radial Reingold–Tilford Tree by Mike Bostock
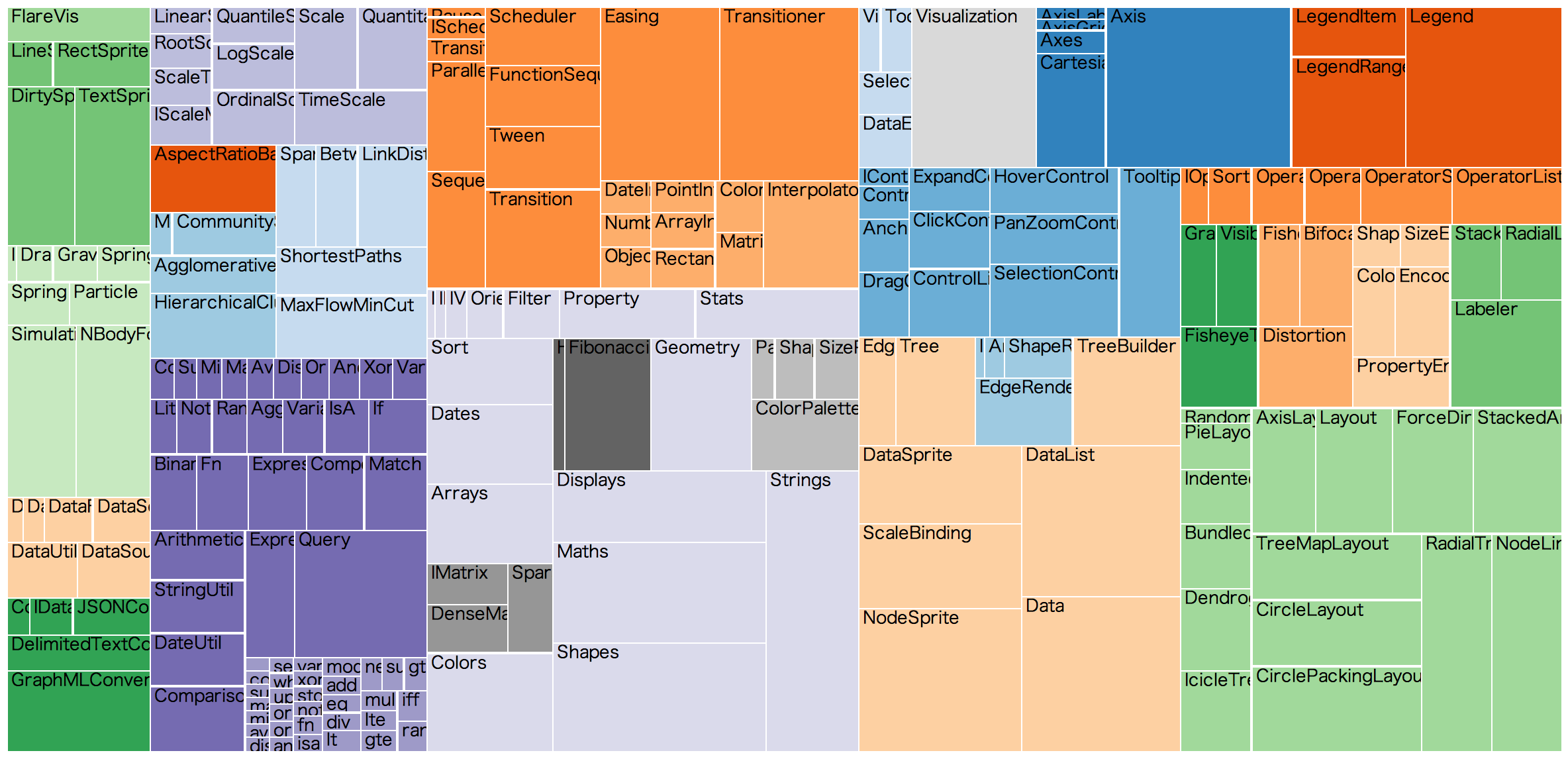
Treemap by Mike Bostock for Flare visualization toolkit classes
上記の例は同一の小さなJSONデータを描画していますが、ネットワークデータに対するこの手法の上手い使い方は以下の様なものです:
- データ全体の傾向をTreemapで示す
- どのサブグループ/サブネットワークがどのくらいの規模を持っているか
- グループの親子関係
-
各カテゴリのサブグラフを必要に応じてnode-link diagramとして展開する
- Treemapで見えないコネクションの詳細を見せる
この組み合わせは__親となるデータが巨大な場合に特に有効です__。Treemap化により、サブグラフ内のコネクションに関する情報は消えますが、かなり大きなデータでも全体と部分の関係性を直感的に理解できるうえに、内部コネクション(エッジ)の詳細が必要な場合は、人間が理解できる大きさの小規模なサブグラフとして展開できるため、全体と詳細の両方をカバー出来ます。これはインタラクティブなWebアプリケーションとして実装した時に特に効果を発揮する方法です(詳細は後述)。
グループ間の関連性が重要なデータに対するChord Diagram
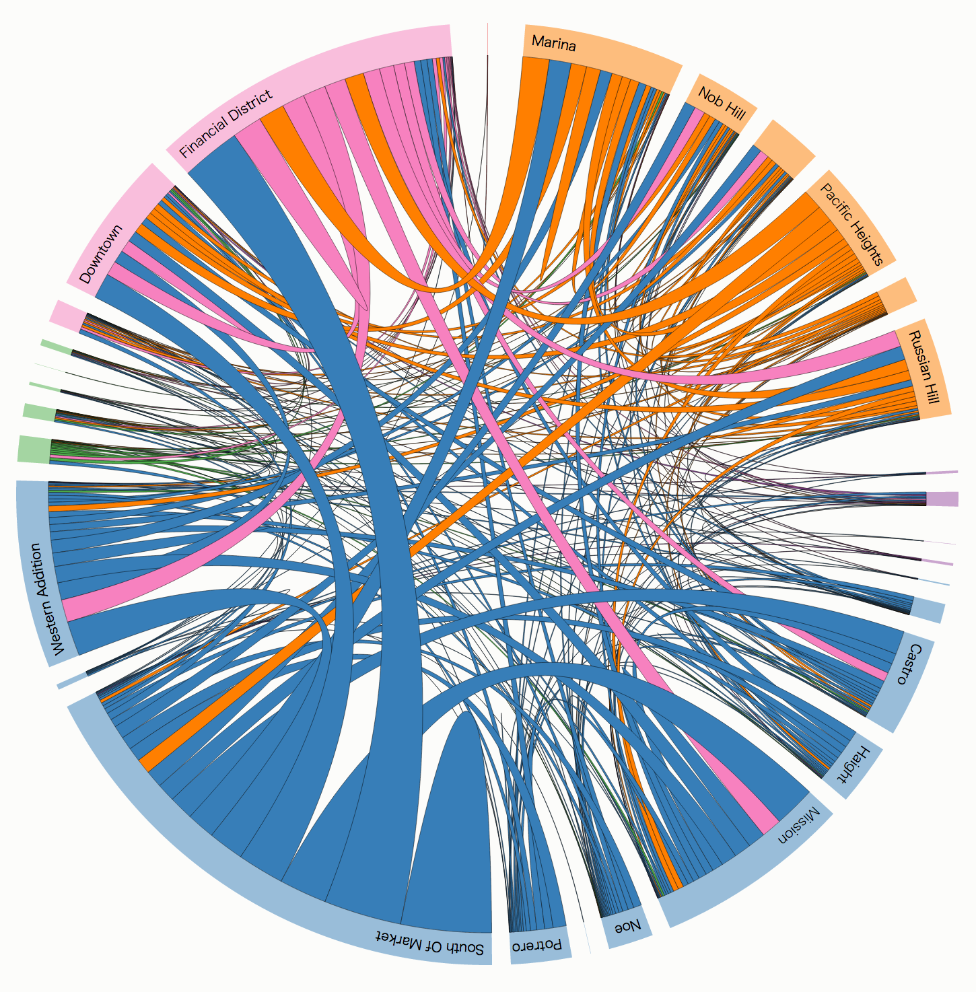
Uber Rides by Neighborhood by Mike Bostock
これはある意味node-link diagramの一種とも言えますが、親となるネットワーク内のグループ間のおおよその繋がりが重要な場合には利用を検討してみる価値があります。
これの発展形でcircosと言うとても美しい画像を生成する手法があるのですが、個人的には見る人に求めるリテラシーが高過ぎるかな…と言う立場です。もちろん利用に明確な意図のある場合は別です。例えばバクテリアは我々と異なり環状DNA構造を持っていますから、Circosのような手法でベースとなるリングを構築し、その上に様々な情報をマッピングするのは理にかなっています。
最初に書いたように、これらの手法は元になるデータの状態により限定されます。__ここでもデータの傾向を考慮に入れず利用すれば、「美しいジャンク」の出来上がり__ですからご注意を。
全く新しい描画方法を開発する
巨大グラフの可視化は、可視化そのものが研究対象の専門家も試行錯誤している段階で、様々な手法が考案されています。しかしまだ決定打と呼べるものはありません。比較的新しいグラフ可視化の手法としては、以下の様なものがあります:
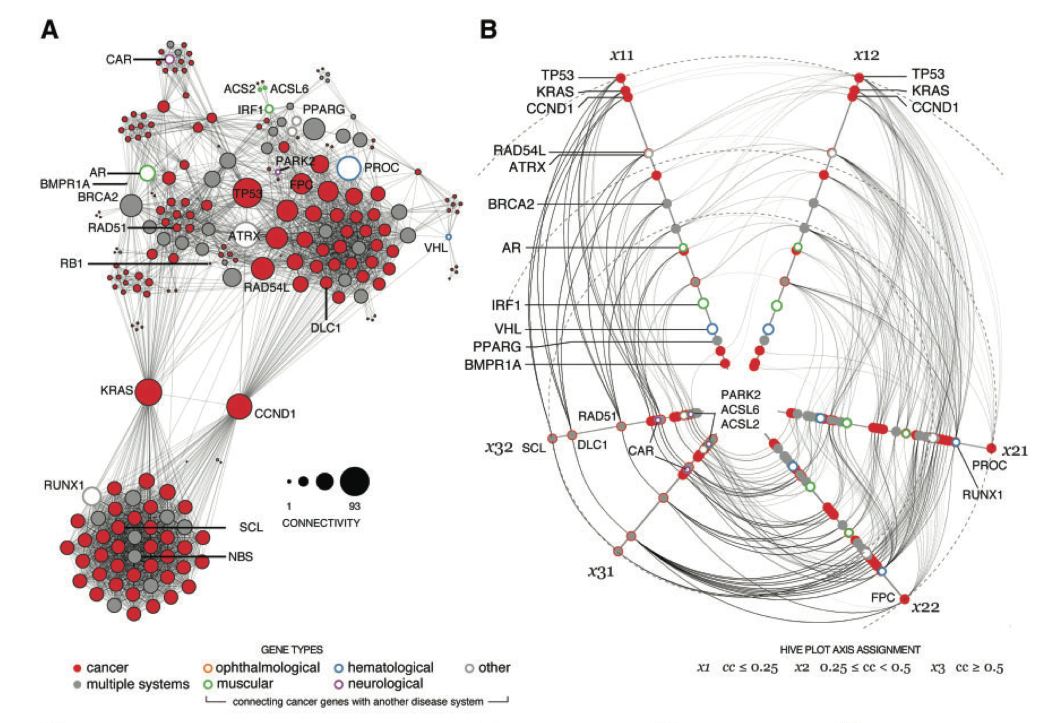
Hive Plot
Krzywinski M, Birol I, Jones S, Marra M (2011). Hive Plots — Rational Approach to Visualizing Networks. Briefings in Bioinformatics (early access 9 December 2011, doi: 10.1093/bib/bbr069).
BioFabric
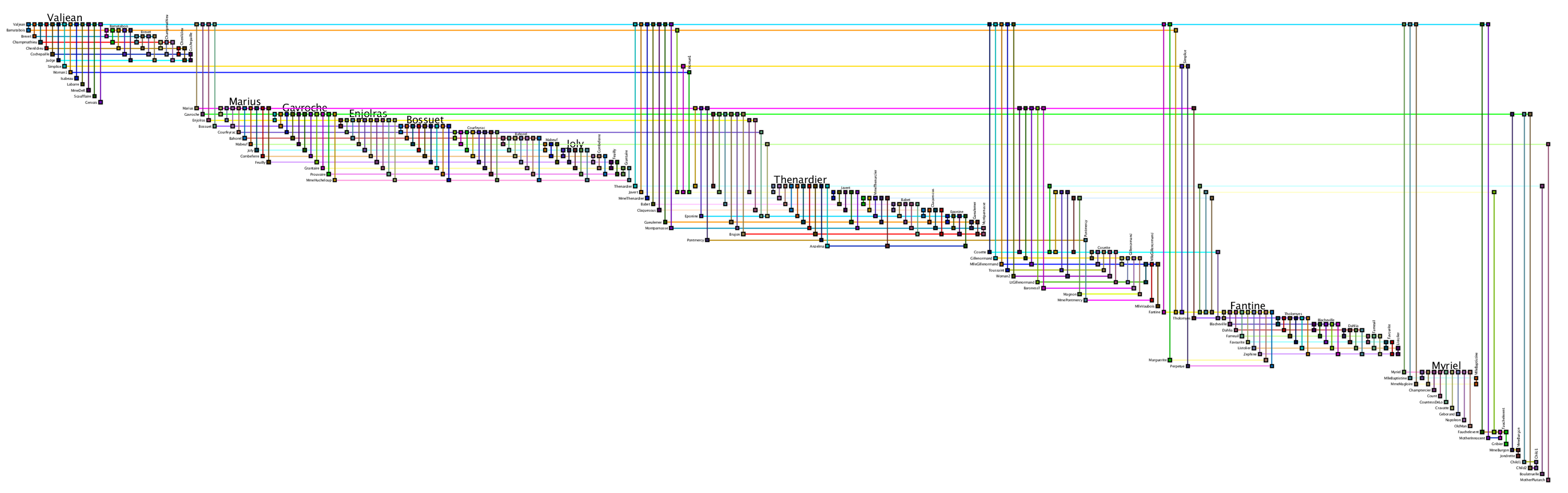
Les Miserables Network visualized with BioFabric (Longabaugh, W.J.R. Combing the hairball with BioFabric: a new approach for visualization of large networks. BMC Bioinformatics, 13:275, 2012.)
これらも万能とはいえない上に、単なるnode-link diagramに比べると、見る人が読み方を学ぶのに少し時間がかかると言う弱点があります。私は研究者ではなく、研究者が開発した結果をプログラマや一般ユーザーが使えるレベルに下ろしてくるのが仕事なため、基本的にここへは深入りしないようにしています。私の観測範囲がどうしても生物学周辺での応用に偏っているので、まだまだ他にもたくさんの実験的手法があると思いますが、基本的な方向性は似ています。つまりよく「巨大な毛玉」と揶揄される大規模ネットワークのnode-link diagramを、なんとか直感的に理解できるものに変えようという試みです。これはすなわち今までなかった可視化手法そのものの開発であり、なかなか難しいトピックです。この辺りを追求したい方はもう既にやっていると思いますが、IEEEのカンファレンスペーパーなどを追いかけるのが良いと思います。
インタラクティブ性
ネットワークのサイズが大きくなるにつれて、シンプルなnode-link diagramの優位性・自明性は急速に失われます。しかしそれをそのまま使いながら弱点を克服する全く別のアプローチもあります。それが__インタラクティブな可視化__です。ここまでは、最終的にPDFなどへ出力するユースケースを想定してきました。しかし計算機ベースの可視化は手描きのイラストとは異なり、インタラクティブ性を付与できるのが大きな違いです。同じ手法を使ったとしても、インタラクティブ性があることにより多くの弱点を克服できる場合があります。
データ可視化としてのGoogle Earth
Google Earthを使ったことがある方は多いと思います。あのアプリケーションが素晴らしいのは、データのクオリティもさることながら、地球の俯瞰図という「ズームレベル0」からストリートビューという「ズームレベルMax」までを切れ目なくナビゲートすることが出来る点です。つまり、もしあれが単なる高精細な地球の静止画像だった場合、その優位性は地球儀に対してほとんどありません。Google Earthを有用な可視化たらしめているのは、そのコアをなす機能としてインタラクティブ性があるからです。
インタラクティブな可視化に必要な要素
インタラクティブな可視化アプリケーションに必要とされる基本的な機能はとてもシンプルです:
- ベクターグラフィックスとして実装された基本描画 - ズームによる描画クオリティの毀損を防ぐ
- ズーム/パンの基本的ナビゲーション - 俯瞰図からは見えない詳細情報を提供する
- 検索 - 興味のある細部への素早いアクセスの提供
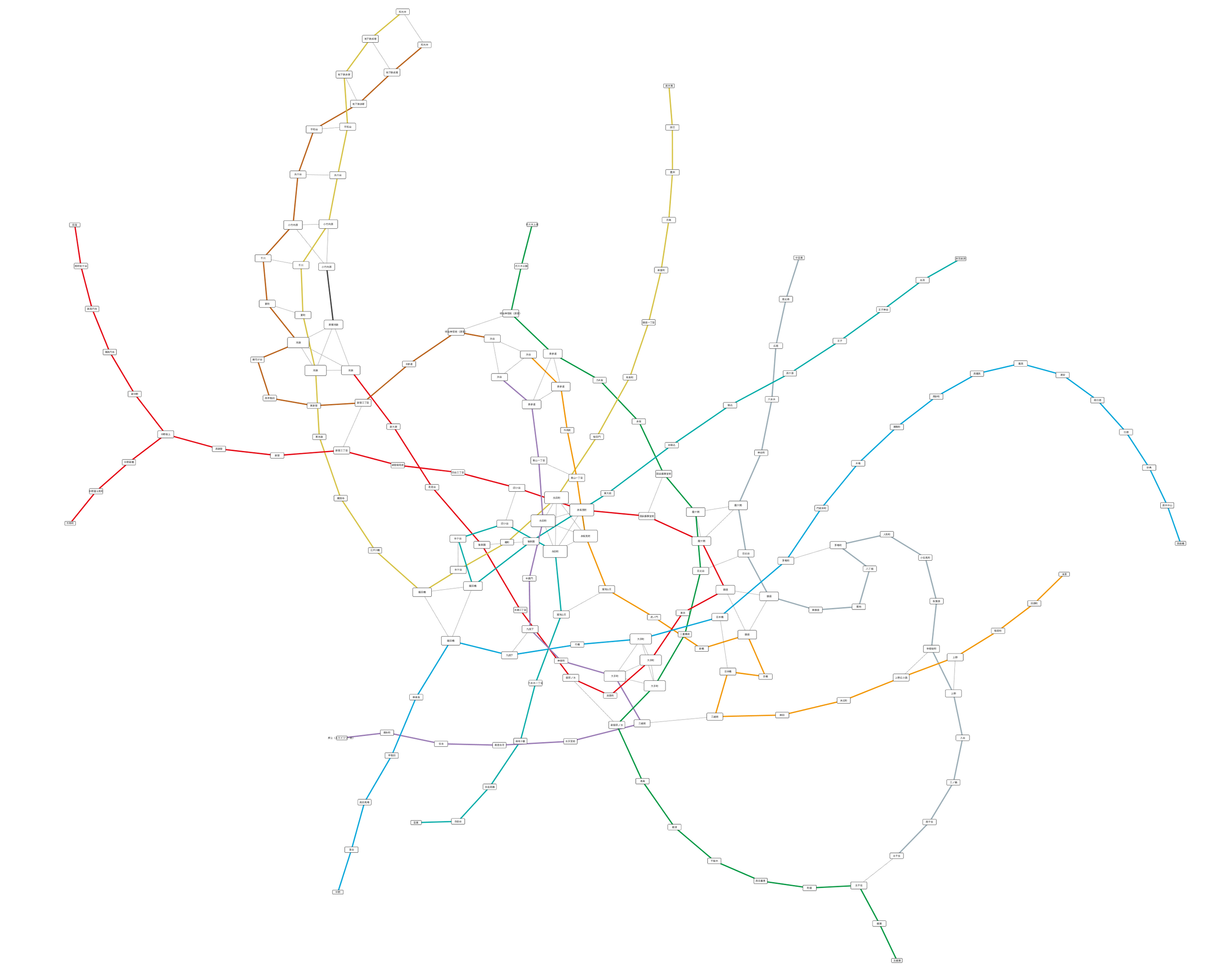
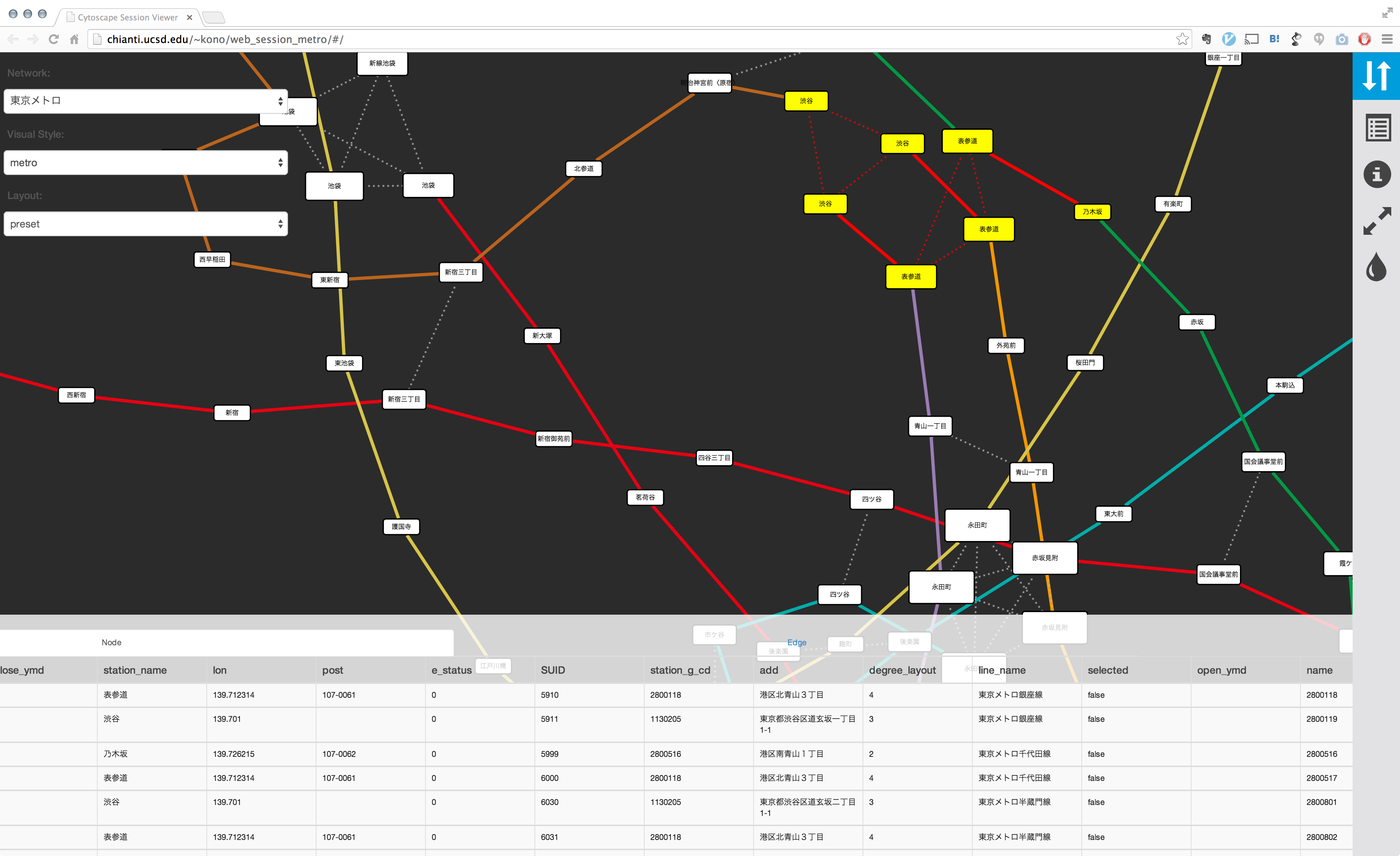
この基本機能を提供するだけでも、細かすぎてよくわからなかった図から様々な情報を読み取ることが可能になります。下の例は私が以前の記事で作ったサンプルですが、両者を比較してみてください:
- 静止画としての東京メトロ路線図グラフ
- 上のデータをJavaScriptでインタラクティブな画像として埋め込んだ例 (注: _style_を_metro_に切り替えてみてください)
これはただ単にグラフとして再構成した東京メトロの各路線をCytoscape.jsで描画しただけですが、ここから様々なインタラクションを追加することにより、静止画では理解するのが難しかった大きさのネットワークもユーザーが能動的にブラウズすることが出来るようになります。考えらる追加機能は色々ありますが、単純な例として次のようなものを思い浮かびます:
- Transform - ユーザー自身が見やすい形への動的な変換
- Filtering - 興味のない情報の排除
手法の切り替えと併用のセクションで述べたTreemapとnode-link diagramの融合は、このテクニックの非常に良い応用例です。Treemapと小さなネットワーク図を並べて静止画を作るのもいいのですが、それよりもTreemap自体をクリッカブルにして、必要に応じて「グループ内をnode-link diagramとして展開」と言うメニューから新しいビューを開けるようにすれば、とても自然なUXを構築できると思います。これはD3.jsのTreemapレイアウトとForce、もしくはCytoscape.jsなどのグラフ専用ライブラリを利用することにより、比較的少ないコード量で実現できます。
このようなアプリケーションを一から開発するのは、かつてはとても大変でした。しかし現在は才能ある多くのJavaScriptハッカーたちがウェブベースの可視化ライブラリを開発しており、これらを組み合わせることで比較的短期間でウェブアプリケーションとしての可視化を構築できます。我々もデスクトップアプリケーションと一緒にCytoscape.jsと言うJavaScriptライブラリも開発しているのは、可視化を発表するプラットフォームとしてのウェブは、もう避けられない未来だからです。
これもまた大きな話題ですので、いずれ何か記事を書いてみたいと思います。
手法の限界を知った上で適切な使い方をする
ここまでは、「上手くいかない場合はどうしたらよいか」を考えてきました。スケーラビリティに問題がある一方、node-link diagramは正しく使えば研究者にとって非常に強力なツールになることも事実です。ここからは逆に「では正しい利用法とはどのようなものか」という点を、いくつか実際の研究で使われている例を通して見てみましょう。
ネットワーク相同性
手前味噌で申し訳ありませんが、私が勤務する研究室でかつて行われていた研究に、生体内機能モジュールの異種間での保存の調査というものがありました。これは比較的よく調べられている生物種のタンパク質間相互作用ネットワークと、そうではない種を比較し、似たような構造を持つサブネットワークを抽出するというものです。以下の図は、かつて在籍した大学院生が在学中に行った研究で、マラリア原虫と酵母のネットワークを比較し、異種間で保存されているモジュールにはどのような物があるのかを調べたものです[Suthram 2005]。現代分子生物学の研究の要となっている相同性検索の考えをネットワークのレベルに拡張したものです。
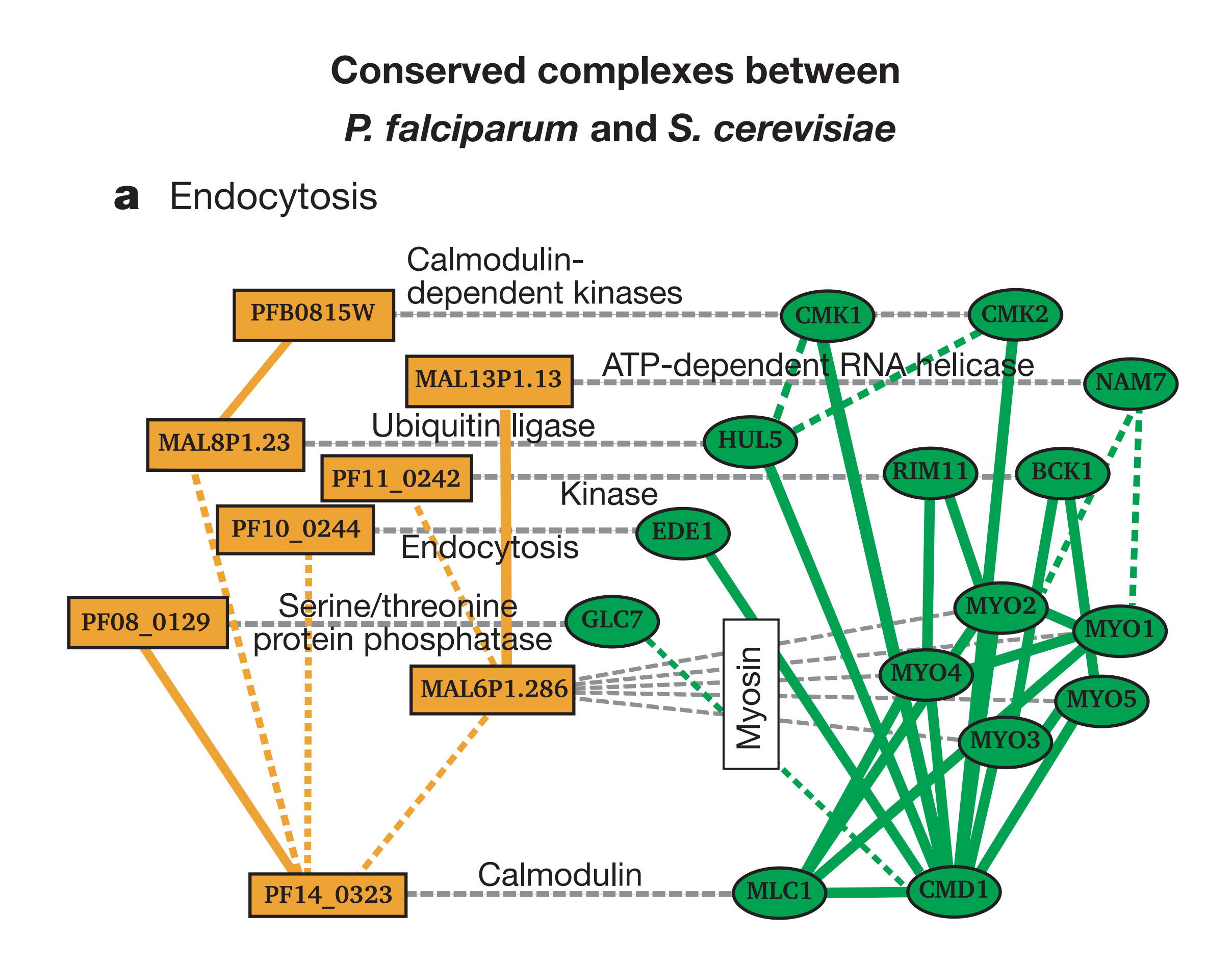
Suthram, S., Sittler, T., and Ideker, T. The Plasmodium protein network diverges from those of other eukaryotes. Nature 438(7064):108-12 (2005).
ここで実際に使われた手法は、
- 似たようなサブモジュールを統計的手法で抽出
- Side-by-sideで両モジュールを比較できるように自動レイアウト
- Cytoscapeで読めるデータとして出力
- そのアウトプットを、論文にするときに少しだけ手でレイアウト修正
4番目のステップに「インチキだ!」と思われる方も居るかもしれませんが、この辺りはバランスで、人間がやったほうがうまくいく、もしくはそれほど手間がかからない場合は、最後の手直しをマニュアルで行うことも有効です。このワークフローにより、異なるネットワークに存在する似通った部分構造というものが分かりやすく提示できていると思います。生物系のデータではなくても、部分構造の類似性比較、あるいはネットワークモチーフの抽出といった場合に、同じような手法で可視化が行えると思います。ここでのポイントは、__可視化は計算によって導かれたサブネットワークにのみ適用されていて、大元の巨大なネットワークをそのまま描画しているわけではない__ことです。グラフクラスタリングなどもそうですが、全体の可視化よりも意味のあるモジュールの抽出と可視化をペアで利用するのが有効です。
肥満のソーシャルネットワーク上での伝播
ここではちょっと毛色の変わったデータを見てみましょう。上記の例は分子生物学の専門的な論文で用いられたもので、なかなかその背景が伝わりにくいと思うのですが、こちらは誰にでも理解しやすいユニークな研究です。我々のプロジェクトのコラボレーターにジェームス・ファウラー教授が居ます。日本語でも著書の翻訳が出ているようです:
彼はソーシャルネットワークをキーワードに、様々な研究を行っています。その中でも有名なものに、「肥満は果たして身近な人を通じて伝播するのか?」と言う疑問を検証した論文があります。
Nicholas A. Christakis, James H. Fowler. The Spread of Obesity in a Large Social Network Over 32 Years. New England Journal of Medicine 357 (4): 370–379 (26 July 2007)
元となった論文で彼の使っている可視化手法は、一種のSmall Multipleと考えることが出来ます。年ごとに同じネットワークのデータを使い、そこに別のデータセット(この場合は調査参加者のBMI)をマッピングし、タイムポイントごとの変化を提示しています。アニメーションを使わないで変化を示すのにこれはとても有効で、各実験参加者のBMIをそのままチャートにするよりも、「ソーシャルネットにおける肥満の伝播を時系列で可視化する」と言う研究のテーマを上手く表していると思うのですがいかがでしょう?
なお、これらのデータをCytoscapeのセッションファイルとして配布すれば、ユーザーは自由にその中身にアクセスしたり、見やすく改変することも出来ます。操作できるデータ(Cytoscapeのセッション、ウェブアプリケーションでの公開、Rなどを使ったのならばそのセッションのデータファイル、IPython Notebook等)としてユーザーが好きな様に加工できる、見られるようにして配布するのも、データのサイズによっては有効ですから検討してみてください。
まだまだ山のように実例はあるのですが、長くなりそうなのでとりあえずリンクだけ張っておきます。これらは生物学の論文で利用されているものですが、基本的な可視化の考え方はあらゆる分野に応用可能です。
まとめ
結論としては「定番となった可視化手法にはそうなった理由があり、正しく使うことによりデータの理解を助ける可視化を作成することは可能である」と言えます。しかし同時に、__手法・ツールの性質・限界を理解し、ゴールを達成するためにそれらを柔軟に組み合わせる__ことが大切です。我々も、自前で開発したものだけで何とかしようというのはリソースの無駄遣いだと認識していますので、できるだけ外に開いたツール体系を構築するのを目標としています。二次加工が便利なフォーマットでデータを保存することを心がければ、ある手法から別の手法に移るのも容易ですから、そのあたりにも気をつけてあなたにとって便利なワークフローを構築してみてください。
急ぎ足で書いたので説明不足の点も多々あるかと思いますが、いずれもうちょっとブラッシュアップします。