はじめに
sklearn.datasetsに収録されている乳がんの検査のデータを使って、ガンが発生するかどうかを予測してみる。
データの読み込み
ライブラリと乳がんデータを読み込む。
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
% matplotlib inline
import seaborn as sns
from sklearn.datasets import load_breast_cancer
bc = load_breast_cancer()
df_data = pd.DataFrame(bc.data, columns=bc.feature_names)
df_target = pd.DataFrame(bc.target, columns=['class'])
df = pd.concat([df_data, df_target], axis=1)
データ分析と特徴量エンジニアリング
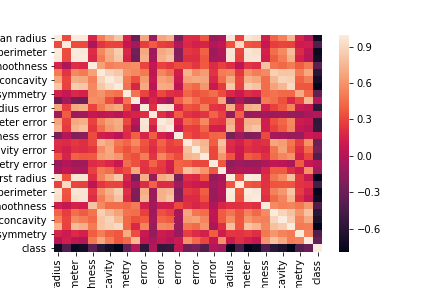
とりあえず相関みる。
df_corr = df.corr()
sns.heatmap(df_corr)
色々分析はしたいが医学の知識が全くないため、相関の絶対値の高いものだけ特徴量として使うことにする。
X = df.loc[:, df_corr["class"].abs().sort_values(ascending=False).index[1:10]].values
y = df.loc[:, ['class']].values
データを標準化する。
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
scaler.fit(X)
X_std = scaler.transform(X)
学習用と評価用にデータを分ける。
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X_std, y, test_size=0.3, random_state=0)
学習と評価
グリッドサーチする。サポートベクターマシンを使うのはただの好みです。
from sklearn.svm import SVC
from sklearn.model_selection import GridSearchCV
param_grid = {'C': [0.001, 0.01, 0.1, 1, 10, 100], 'gamma' : [0.001, 0.01, 0.1, 1, 10, 100]}
grid_search_svm = GridSearchCV(SVC(), param_grid)
grid_search_svm.fit(X_train, y_train)
学習用のデータと評価用のデータでモデルの正答率を確認。
print('train acc: %.3f' % grid_search_svm.score(X_train, y_train))
print('test acc: %.3f' % grid_search_svm.score(X_test, y_test))
print(grid_search_svm.best_params_)
その結果、
train acc: 0.970
test acc: 0.942
{'C': 100, 'gamma': 0.01}
とでた。悪くないスコアだと思うが少し過学習しているようなので、パラメータを軽く調整していく。
svm = SVC(C=300,gamma=0.01)
svm.fit(X_train,y_train)
print('train acc: %.3f' % svm.score(X_train, y_train))
print('test acc: %.3f' % svm.score(X_test, y_test))
上のパラメータで、
train acc: 0.972
test acc: 0.965
だった。結構高いので満足。
まとめ
データ分析の部分がてきとうだったが、精度はよかったので良しとしよう。