概要
先日Augmentationのライブラリであるimgaugを用いてBounding BoxごとのAugmentationを行なっていた際、rotate(回転)を使うとBounding Boxが大きくなることを知ったため、しっかりアノテーションしたものと差があるのか比較してみました。
行なったこと
- LabelImgを用いてアノテーション。
- imgaugを用いて、Bounding Boxごと画像をAugmentation。
- Augmentationした画像に対して、再度アノテーションを実施してアノテーション情報を変更した学習用データと、変更していない学習用データの2つを用意。
- それぞれKerasのYOLO-v3を用いて学習、予測。
データについて
学習にはKaggleのDatasetsにあるBlood Cell ImagesのJPEGImagesに格納されている白血球の画像を使用しました。
白血球は基本的には以下の5種類に分類されます。今回はこの5種類の分類、検出を行なっています。
| 白血球名 | 画像 | 拡張前の枚数 |
|---|---|---|
| 好中球(NEUT) |  |
205枚 |
| リンパ球(LYM) |  |
32枚 |
| 単球(MON) |  |
16枚 |
| 好酸球(EOS) |  |
50枚 |
| 好塩基球(BAS) |  |
7枚 |
(本当にこの分類で正しいか怪しい画像は削ったり、枚数の少なかった好塩基球は拾ってきた画像を何枚か追加したりしています)
Augmentation
今回は画像が各500枚ずつになるように、以下の種類のAugmentationを行いました。
seq = iaa.SomeOf(4, [iaa.Affine(rotate=(-45, 45)),
iaa.Fliplr(0.5),
iaa.Flipud(0.5),
iaa.GaussianBlur(sigma=(0.0, 1.0)),
iaa.ContrastNormalization((1, 1.5), per_channel=0.7),
iaa.Dropout(p=(0, 0.03))
])
こんな感じになりました。
| 拡張前 | 再アノテーションした画像 | 再アノテーションしていない画像 |
|---|---|---|
 |
 |
 |
一番右がBounding BoxごとAugmentationした画像ですが、回転させた場合、Bounding Boxが大きくなっていることが分かります。
結果
テスト画像には、上記と同じく、KaggleのDatasetsにあるBlood Cell ImagesのTEST_SIMPLEに格納されている画像を使用しました。
- 再アノテーションした学習用データの場合
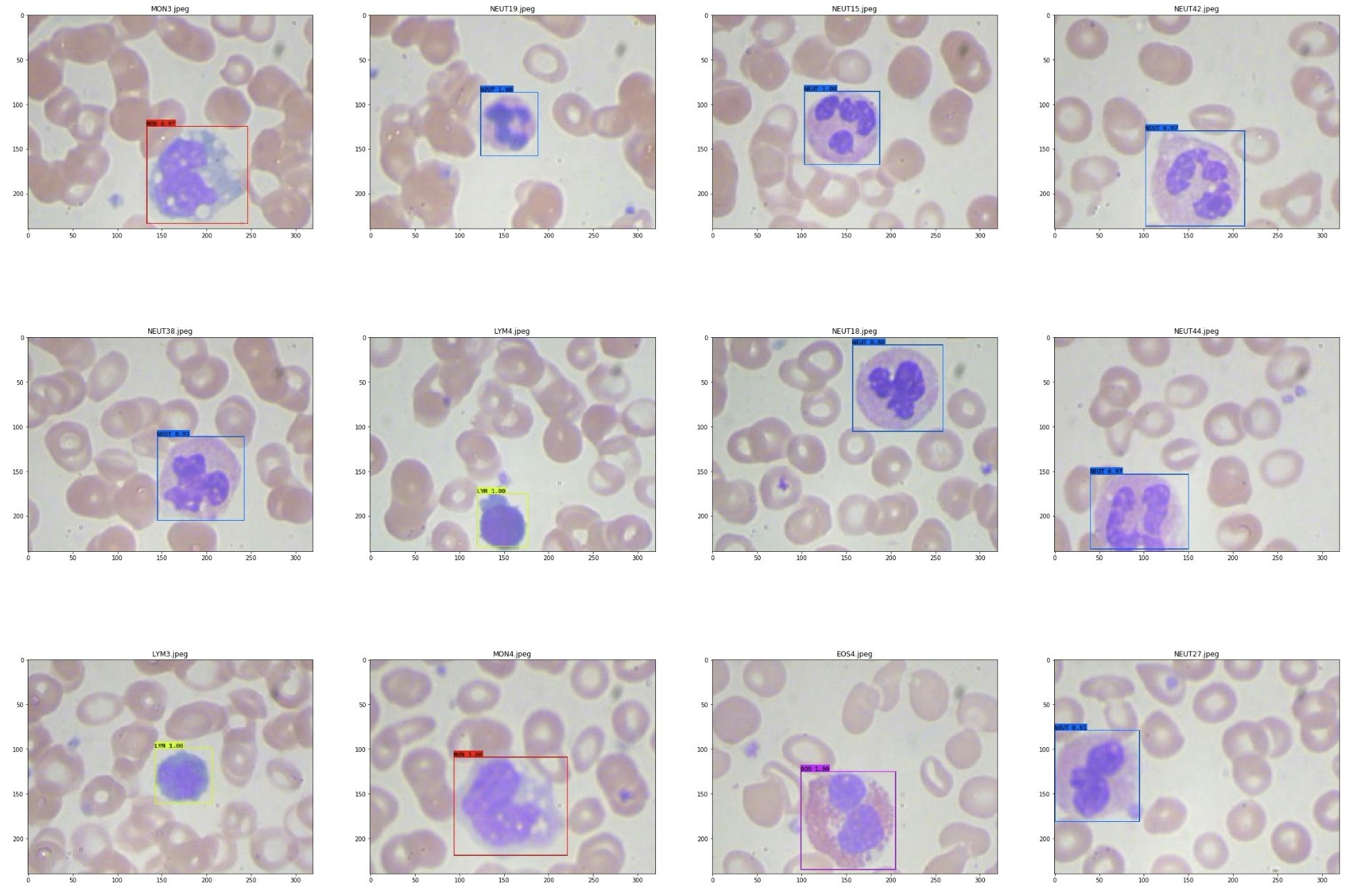
- 再アノテーションしていない学習用データの場合
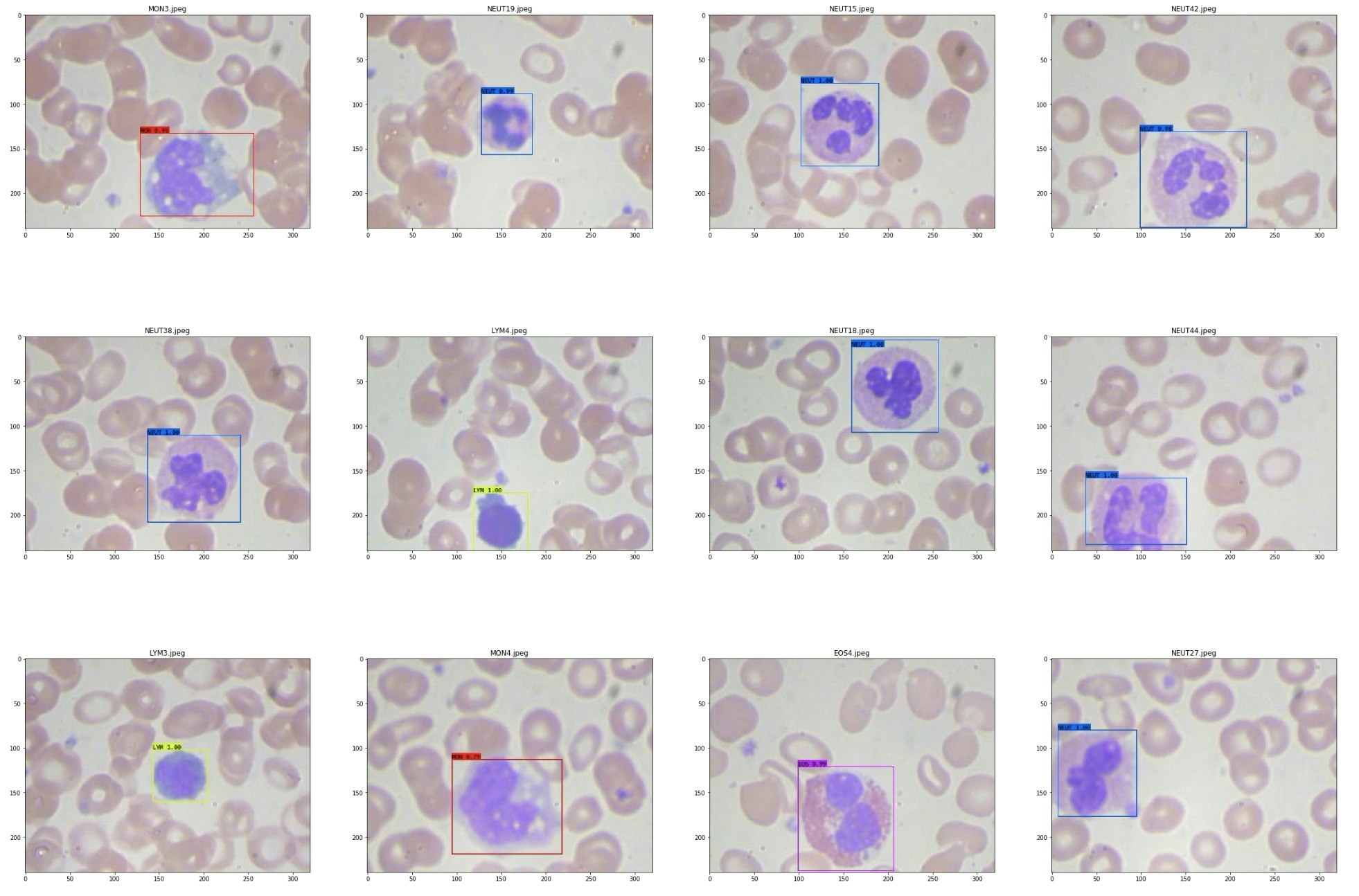
分類の結果は同じでしたが、いくつかの画像ではBounding Boxに若干ですが違いが見られました。
左が再アノテーションした学習用データを用いた場合で、右が再アノテーションしていない学習用データを用いた場合です。
上の画像は、左のBounding Boxに比べると右のBounding Boxは少し小さくなり(Bounding Boxから白血球がはみ出している)、下の画像は逆にBounding Boxが大きくなっていました。
まとめ
今回の結果ではBounding Boxに若干の違いはありましたが、分類やBounding Boxの位置には問題はなかったため、個人的にはあまり気にしなくても良いのかなと感じました。



