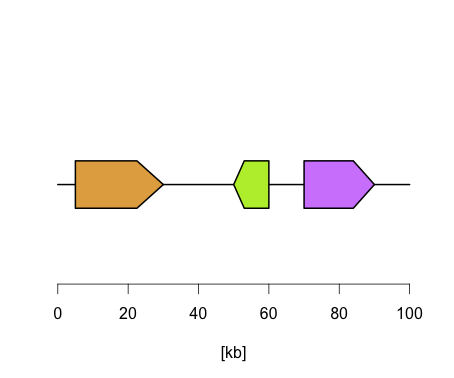
よく論文にある「複数の遺伝子が五角形として並べてある図」をRで描いてみます.
遺伝子のstartとendとstrandから,五角形を描くのに必要な座標を計算する関数
convertToOperon <- function(x1, x2, strand, heightOfOperon=1, ratioOfTriangle=0.3, y1=1){
y2 <- y1 + heightOfOperon
y3 <- y1 + heightOfOperon/2
if(strand == 1){
xAngle <- (1-ratioOfTriangle)*(x2-x1)+x1
x <- c(x1, x1, xAngle, x2, xAngle)
y <- c(y1, y2, y2, y3, y1)
return(list(x, y))
}else{
xAngle <- ratioOfTriangle*(x2-x1)+x1
x <- c(x1, xAngle, x2, x2, xAngle)
y <- c(y3, y2, y2, y1, y1)
return(list(x, y))
}
}
たくさんの遺伝子をまとめて描く関数
# 入力は,複数の遺伝子のstart, end, strand, 色を格納したデータフレーム(dat)と,どこから(start)どこまで(end)
plotOperons <- function(dat, start, end, heightOfOperon=0.7, ratioOfTriangle=0.3, y1=1, lwd=2, xlab="[kb]"){
genome <- c(start, end)
height <- c(0,3)
# Define plot space
plot(genome, height, type = "n", xlab = xlab, ylab="", axes=F)
axis(1)
# Draw horizontal line
lines(c(start, end), c(y1+heightOfOperon/2, y1+heightOfOperon/2), lwd=lwd)
if(is.factor(dat[,4])){
dat[,4] <- as.character(dat[,4])
}
for(i in 1:nrow(dat)){
coo <- convertToOperon(dat[i,1], dat[i,2], dat[i,3],
heightOfOperon=heightOfOperon,
ratioOfTriangle=ratioOfTriangle,
y1=y1)
print(dat[i, 4])
polygon(coo[[1]], coo[[2]], col=dat[i,4], lwd=lwd)
}
}
入力データ
dat <- data.frame(start=c(5, 50, 70),
end=c(30, 60, 90),
strand=c(1, -1, 1),
color=c("orange", "green", "magenta"))
プロット
plotOperons(dat, 0, 100)
追記
こんなのもあるそうです genoPlotR