はじめに
Pythonで2次元配列データのピーク検出のライブラリが見当たらなかったので、自分で関数を作ってみました.
2次元配列データのピーク検出
結論から言うと、以下の関数を使用すればピークを検出できます.
関数が何をやっているかについては2次元配列データのピーク検出の仕組みで説明しています.
# ピーク検出関数
def detect_peaks(image, filter_size=3, order=0.5):
local_max = maximum_filter(image, footprint=np.ones((filter_size, filter_size)), mode='constant')
detected_peaks = np.ma.array(image, mask=~(image == local_max))
# 小さいピーク値を排除(最大ピーク値のorder倍以下のピークは排除)
temp = np.ma.array(detected_peaks, mask=~(detected_peaks >= detected_peaks.max() * order))
peaks_index = np.where((temp.mask != True))
return peaks_index
| 引数 | 説明 |
|---|---|
| image | 入力データ(2D numpy array) |
| filter_size | ピーク検出の解像度 値が大きいほど荒くピークを探索 |
| order | ピーク検出の閾値 最大ピーク値のorder倍以下のピークを排除 |
| 返り値 | 説明 |
|---|---|
| peaks_index | ピークが格納されている要素のインデックス |
- シミュレーション
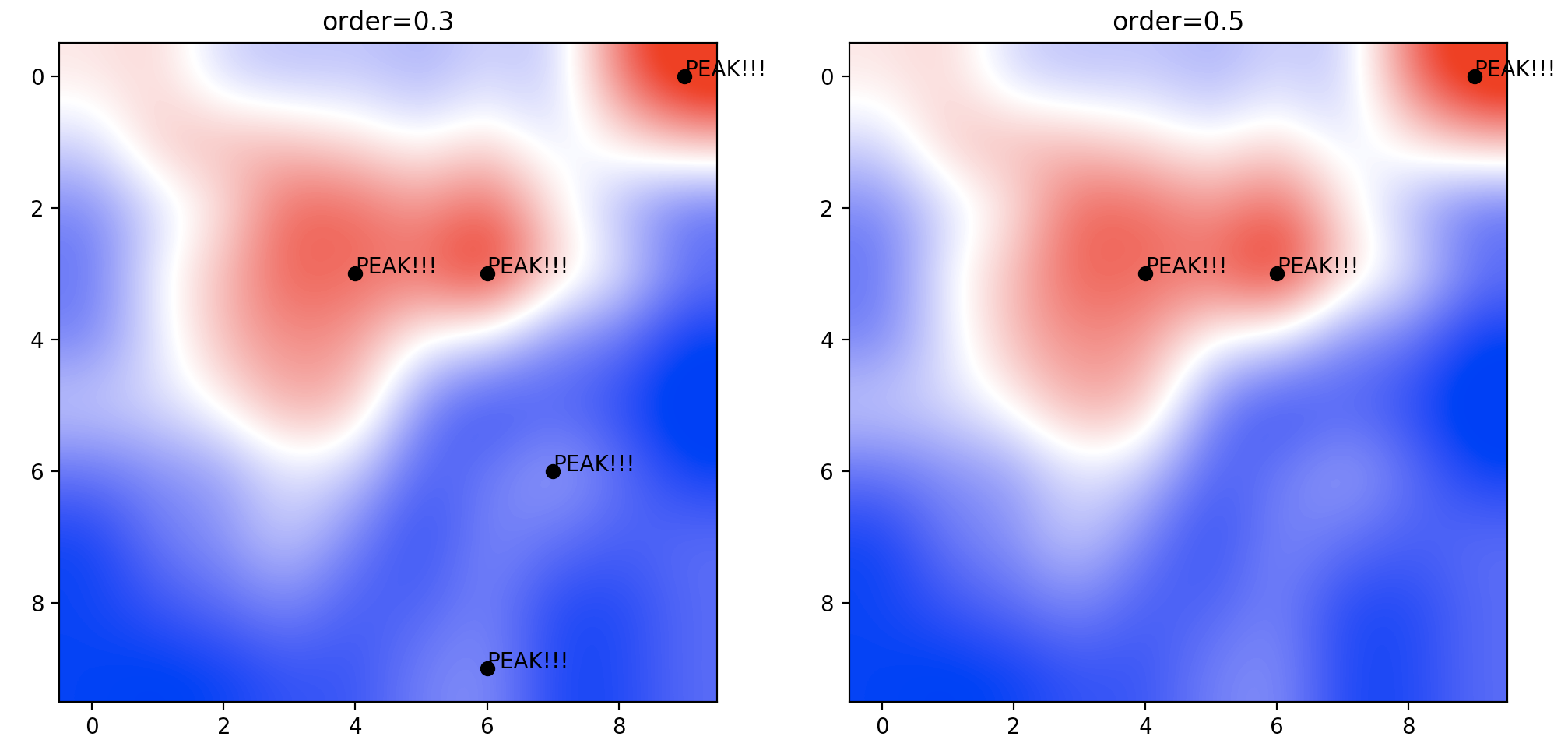
以下の2次元配列データからピークを検出してみます.
右図は左図を上から見たものです.
- サンプルコード
import numpy as np
import matplotlib.pyplot as plt
from scipy.ndimage.filters import maximum_filter
# ピーク検出関数
def detect_peaks(image, filter_size=3, order=0.5):
local_max = maximum_filter(image, footprint=np.ones((filter_size, filter_size)), mode='constant')
detected_peaks = np.ma.array(image, mask=~(image == local_max))
# 小さいピーク値を排除(最大ピーク値のorder倍のピークは排除)
temp = np.ma.array(detected_peaks, mask=~(detected_peaks >= detected_peaks.max() * order))
peaks_index = np.where((temp.mask != True))
return peaks_index
# データ読み込み(好きな2d arrayを読み込んでください)
Map_val = np.loadtxt('Map2_val2.txt')
Map_val = np.rot90(Map_val, k=-2)
# ---------- ピーク検出 ---------- #
maxid1 = detect_peaks(Map_val, order=0.3)
maxid2 = detect_peaks(Map_val, order=0.5)
# ---------- 描画 ---------- #
Fig = plt.figure(figsize=(12, 6))
Map1 = Fig.add_subplot(121)
Map2 = Fig.add_subplot(122)
Map1.set_title('order=0.3')
Map1.imshow(Map_val.T, interpolation='spline36', cmap="rainbow")
for i in range(len(maxid1[0])):
Map1.scatter(maxid1[0][i], maxid1[1][i], color='black')
Map1.text(maxid1[0][i], maxid1[1][i], 'PEAK!!!')
Map2.set_title('order=0.5')
Map2.imshow(Map_val.T, interpolation='spline36', cmap="rainbow")
for i in range(len(maxid2[0])):
Map2.scatter(maxid2[0][i], maxid2[1][i], color='black')
Map2.text(maxid2[0][i], maxid2[1][i], 'PEAK!!!')
plt.show()
2次元配列データのピーク検出の仕組み
簡単に言うと周りより値が高いところをピークとして検出しています.
その後、小さい値のピークを排除しているだけです.
「周り」をどれぐらいまで考慮するかのパラメータをfilter_sizeで、「小さい」っていう尺度をorderで決定しています.
具体的には、以下の処理によりピークを検出しています.
- 最大値フィルタによる処理
- マスク処理
- 低値ピークの排除
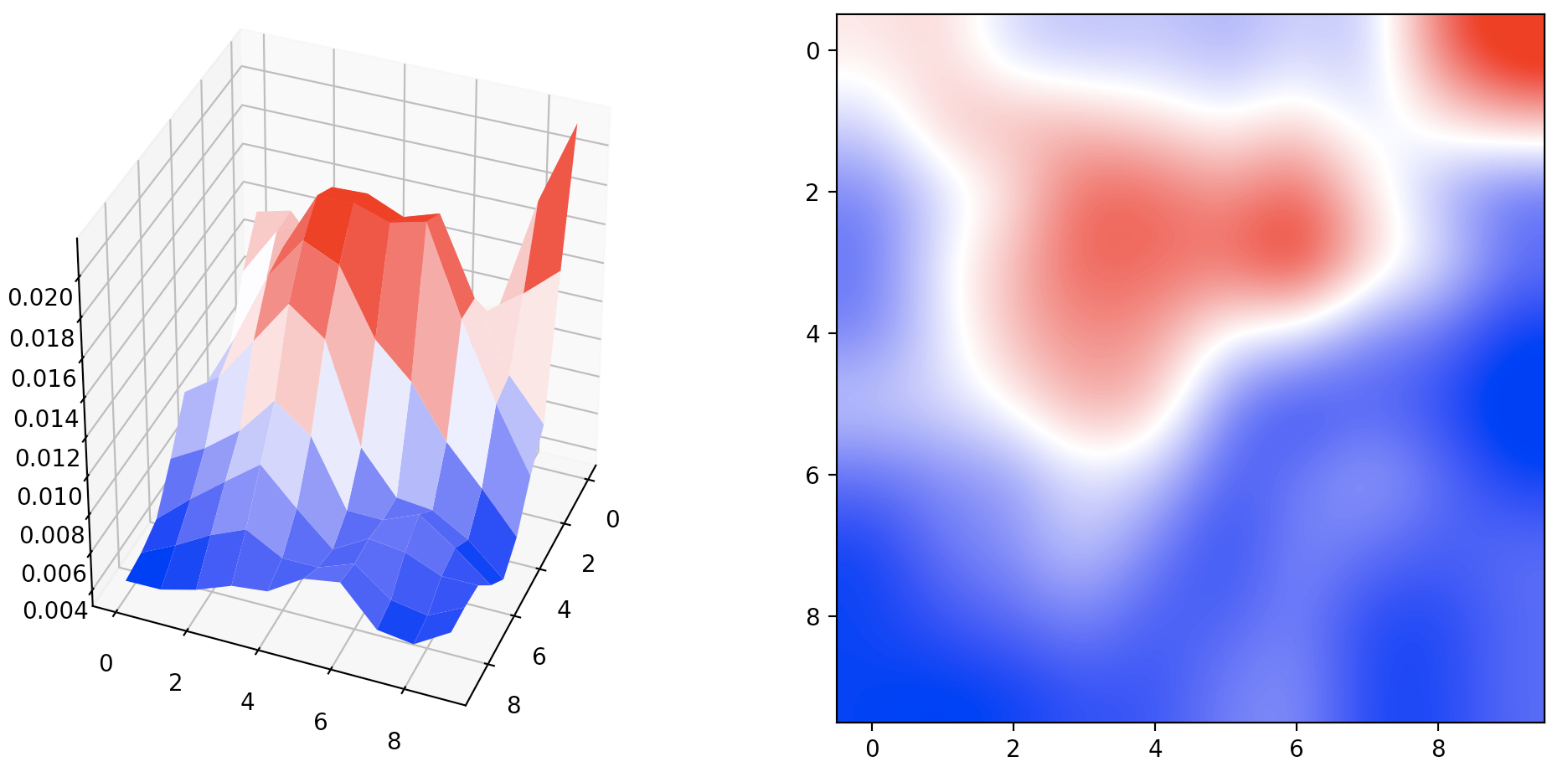
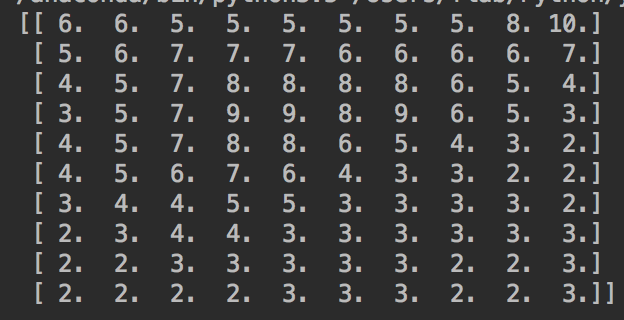
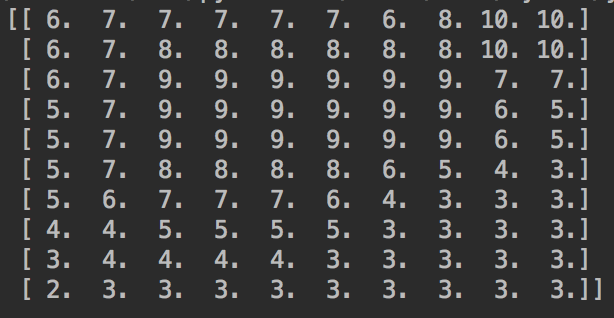
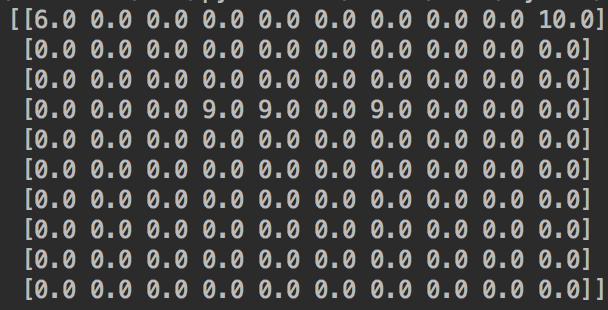
今回は、以下のようなデータを例に説明します.
元配列
上の配列を可視化したもの
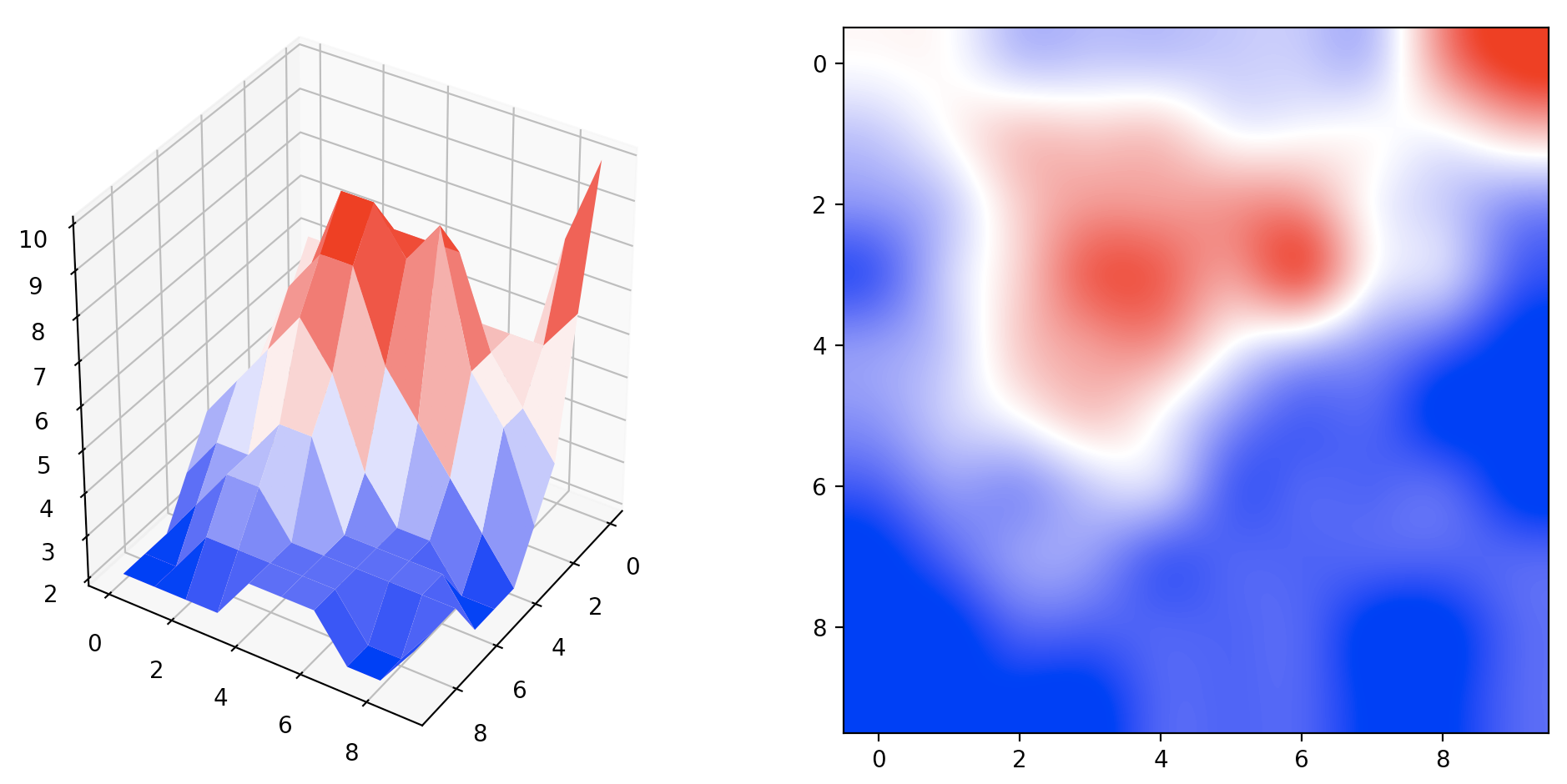
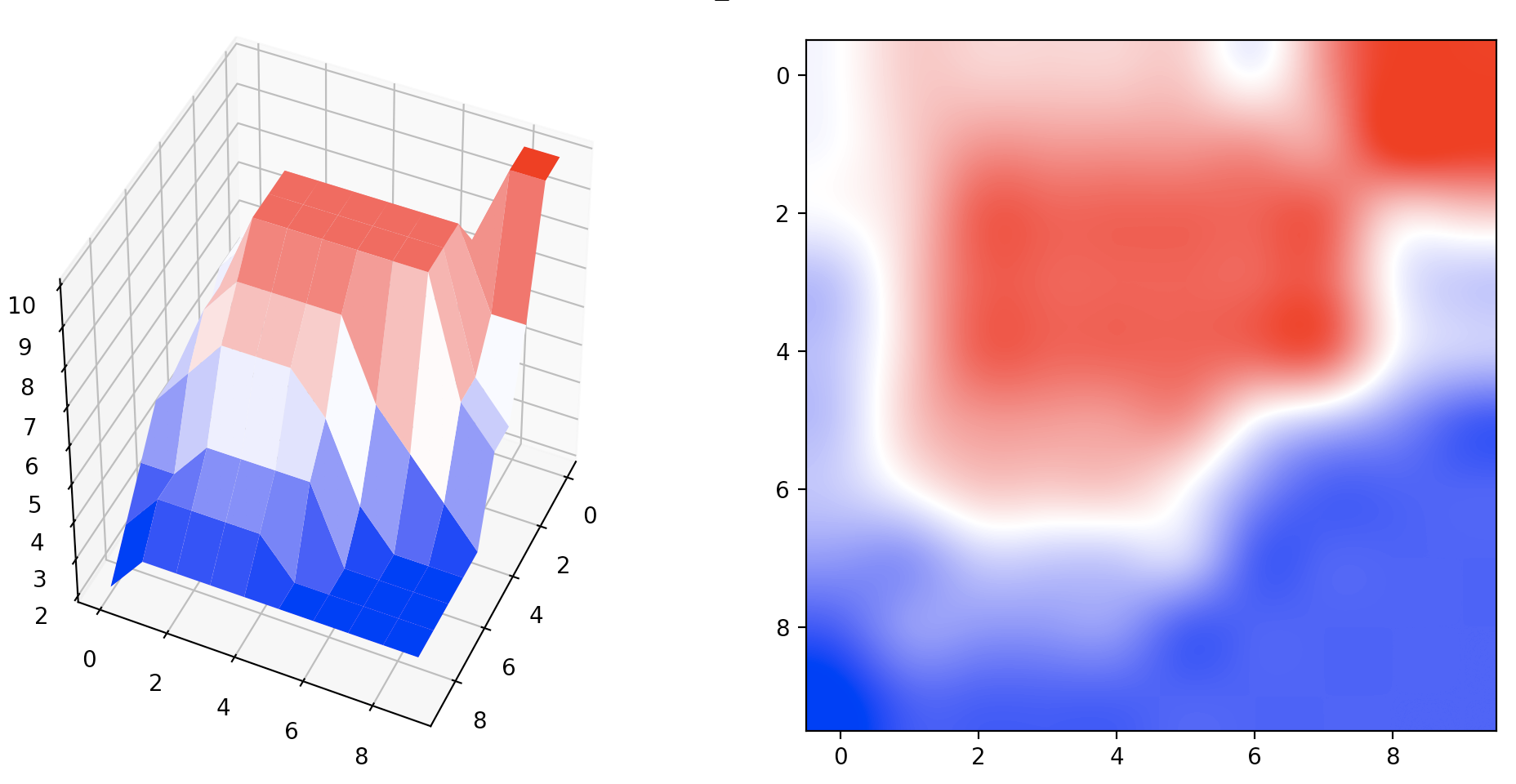
- 最大値フィルタによる処理
まず、filter_sizeで設定した大きさの最大値フィルタでデータをのっぺりとさせます.
3×3サイズの最大値フィルタで処理後
- マスク処理
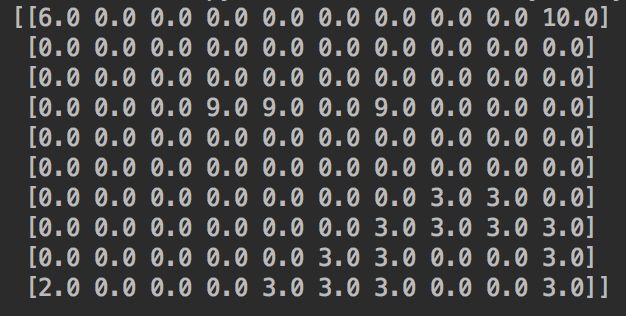
元の配列と同じ値を持つ要素だけ残し、それ以外の要素を0にします.
そうすることで、周りより値の高い要素だけが残ります.
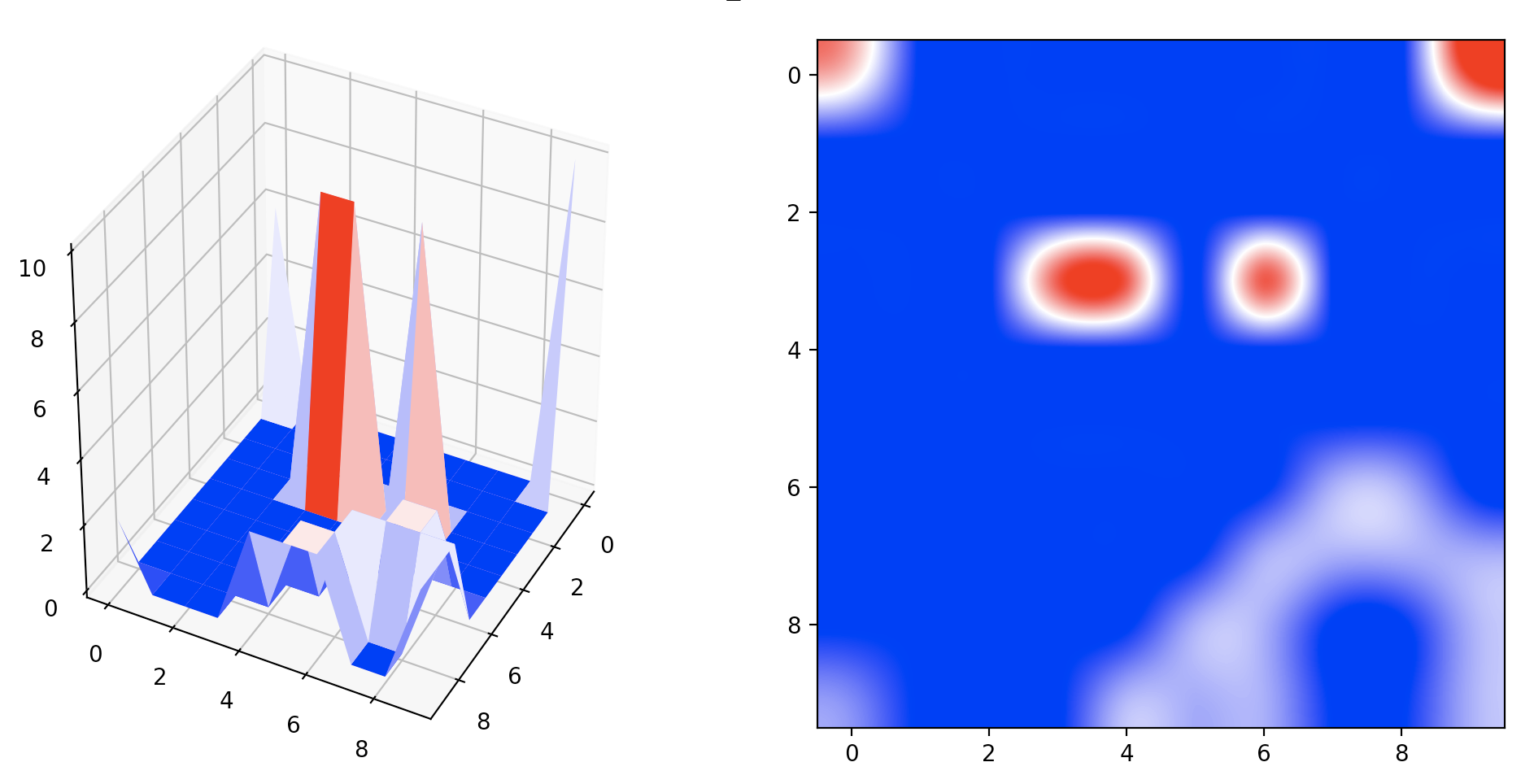
- 低値ピークの排除
orderで設定した値に応じて、小さい値のピークを排除します.

これでピークを検出することができました.
最後に
今回は「値が周りより大きい所」and「値が全体の平均より大幅に大きい所」をピークとして検出する関数を作りました.
しかし、「値が周りより大きい所」and「値が周りの平均値より大幅に大きい所(周りとの変化が大きい所)」をピークと考えることもでき、その場合は「3.低値ピークの排除」のところの処理が変わりますので、興味ある方はぜひ実装して見てください(平均値フィルタを使えば実装できると思います).
またピーク検出について調べていると、そもそもピークをどう定義するかやノイズが大きい場合のノイズとピークの違いなど、なかなか奥が深いことがわかりました.
おまけ
1次元配列データのピーク検出
scipy.signal.argrelmaxを使用すればできます.
引数のorderの値によって検出するピークの閾値を決定します.
値が低いほど、敏感にピークを検出します.
- シミュレーション

- サンプルコード
import numpy as np
import math
import matplotlib.pyplot as plt
from scipy import signal
# 人工データの作成
N = 100
x = np.linspace(-1, 1, N)
theta = 2 * math.pi * ((x + 1.0) / 2.0)
y = np.sin(theta) + np.random.randn(N)*0.05
# ピーク値のインデックスを取得
maxid = signal.argrelmax(y, order=5) # 最大値
# 描画
Fig = plt.figure(figsize=(6, 6))
Map1 = Fig.add_subplot(111)
Map1.plot(x, y)
Map1.scatter(x[maxid[0]], y[maxid[0]])
for i in range(len(maxid[0])):
Map1.text(x[maxid[0][i]], y[maxid[0][i]], 'PEAK!!!')
plt.show()