Python は遅いとよく言われます。そのときによく引用されるものに一つに、Julia Micro-Benchmarks のページがあります。Python は C と比較すると、recursion_fibonacci だと 100倍ぐらい遅く、テスト 8 項目のうち 5 項目で 10 倍以上遅いという結果になっています。これを見ると Python は激遅だという印象を持つことは間違いありません。
しかしながら、Python は Numpy を始めとして、高速化のためのツールが充実しているので、Python で作成したアプリケーションが遅いわけではありません。Julia Micro-Benchmarks では、Cython, Numba 等の Python を高速化するツールの存在が無視されています。Julia Micro-Benchmarks では、ベンチマークのコードが公開されている(Micro-Benchmarks)ので、それを使用して Numba, Cython 等についてベンチマークしてみました。
結果
結果は以下のとおりで、Julia Micro-Benchmarks のページと同様に C の所要時間を1として他の処理言語の処理時間が何倍になるかを表にしています。
||C | Julia | Numba | Cython | Pythran | Java | PyPy | Python |
|:-----------|-----:|-----:|-----:|-----:|-----:|-----:|-----:|-----:|-----:|
| iteration_pi_sum | 1 | 1.00 | 1.00 | 1.00 | 1.00 | 2.05 | 5.79 | 51.43 |
| recursion_fibonacci | 1 | 1.48 | 2.88 | 0.85 | 1.91 | 3.33 | 10.71 | 70.77 |
| recursion_quicksort | 1 | 0.96 | 1.10 | 1.07 | 1.10 | 2.26 | 7.61 | 19.19 |
| parse_integers | 1 | 1.42 | 0.49 | 1.08 | 3.97 | 3.33 | 6.42 | 12.27 |
| print_to_file | 1 | 0.63 | 0.32 | 1.00 | 1.76 | 15.21 | 2.49 | 4.62 |
| matrix_statistics | 1 | 1.51 | 1.62 | - | 1.27 | 4.90 | 34.15 | 9.83 |
| matrix_multiply | 1 | 0.86 | 0.95 | 0.96 | 1.01 | 12.37 | 1.16 | 0.97 |
| userfunc_mandelbrot | 1 | 0.80 | 0.75 | 0.79 | 0.72 | 1.07 | 4.73 | 47.70 |
テスト環境: OS Ubuntu 18.04, CPU Intel Core i7-7700
使用したソフトウェアのバージョン:
Julia 1.0, Python 3.7, Numba 0.46.0, numpy 1.17.2, Cython 0.29.13,
Pythran 0.9.3.post1, Java openjdk 1.8.0_222, PyPy 7.1.1-beta0
Pythonについては、Numba では Anacondaを、それ以外では Ubuntu のパッケージを使用
使用したコード、測定結果の詳細については、GitHubで公開しています。
Numba の場合、8つのテスト項目の中で一番遅いのが「recursion_fibonacci」で、C よりも 2.89 倍の時間がかかっています。Numba は、再帰の処理が遅いためです。次に遅いのが、matrix_statistics で 1.67 倍の時間がかかっていますが、Julia との比較だと 1.11 倍遅いだけです。recursion_fibonacci のように再帰が処理の殆どを占めるるケースは現実には殆どないので、Julia と Numba は、ほぼ同じ速さだといっていいと思われます。
Cython の場合は、7つのテスト項目ともほぼ C と同じ速さです。matrix_statistics については、C のコードを見ればわかると思いますが、openblas のライブラリーを使わないと C と同じぐらいの速さにはできません。自分の能力を超えるのでやめました。
Cython, Numba をつかって Julia Micro-Benchmarks をしている記事には既に以下のものがあります。
- どうすればPythonをJuliaと同じくらい速く動かせるのか? : 様々なやり方で計算の高速化を図る
- An Updated Analysis for the “Giving Up on Julia” Blog
「どうすればPythonをJuliaと同じくらい速く動かせるのか?」では、Fibonacci の計算では、Python の方が 3.8 倍速くなっている他、pisum で 1.7 倍、Mandelbrot で 1.6 倍、quicksort で 1.4 倍速いという結果になっています。今回テストした限りにおいては、それほど Python が速いということはなくて quicksort では、むしろ Julia の方が若干速かったです。
一方で、Python が 0.2 倍と遅かった randmatstat については、Numba と Pythran がこの計算で必要な Numpy の関数 np.linalg.matrix_power 等に対応したため、Julia と同程度の速さになりました。当時よりは Numba も成熟してきたといえるでしょう。
「An Updated Analysis for the “Giving Up on Julia” Blog」という記事は、「[どうすればPythonをJuliaと同じくらい速く動かせるのか?」への反論ですが、その記事にある Cython, Numba のコードについては以下のように最適化ができていないので、その記事の内容は信用できません。詳しくは、公開しているコードと見比べてください。
- parse_integers では、Cython のコードの中で hex, int という Python の関数を使用している。
- Cython を Jupiter Notebook 上で %%cython マジックを使ってテストしているが、その場合は、最適化レベルが -O3 になっていない。C を -O3 でコンパイルをしているのならば、Chython も最適化レベルを -O3 でコンパイルすべきである。
- userfunc_mandelbrot では、np.linspace を使っているが、numba を使う場合には C やjulia の場合と同様に for 文で処理した方が速い。
テスト項目 print_to_file については、Julia の文字列は内部的には UTF-8 です。それに対して、Python は、UTF-32, Java は UTF-16 です。ファイルへの書き出しは UTF-8 の場合変換が必要ありません。一方で UTF-16 や UTF-32 の場合には、UTF-8 に変換する必要があるので遅くなります。それで、Numba で UTF-8 でまとめて文字列を作成し、ファイルへの書き出しを1回にするコードを書きました。そうしたら、Numba の方が Julia よりも 2倍速くなりました。時間ができたら Cython のコードも修正しようと思います。また、Java も 15 倍も遅いということになっていますがそのように修正すると随分速くなると思います。
Numba について
Numba は、Python のコードで関数の頭に @jit デコレータを付けるだけで手軽に Julia と同じぐらいに高速化できます。両者は LLVM を使っていてよく似た方法で処理をしているので、Numba で Numpy の処理を高速化する場合には処理速度は Julia と同じぐらいになるはずです。最近は jitclass, list, dict, set, str 等が使えるようになりましたが、それらについては処理が遅い場合があるので使うときに注意が必要です。
Numba という名前は Numpy と Munba(世界で最速で移動する猛毒の蛇)を掛け合わせたものす。名前から考えても、Python + Numpy では for 文で個々の要素にアクセスする処理が極めて遅いという弱点を解消するためのツールだと考えておいた方が無難です。最近、Pandas で Numba を使う議論がなされていますが、Pandas のような大規模なライブラリーで使うのには、まだまだ問題が多いようです。
Numba を使う場合注意しないといけないのは、Numba が対応している型や関数は、Python と Numpy の一部だけだということです。もし、対応していない型や関数があると object mode になってしまうので、Python の場合よりも遅くなってしまうことがあります。Qiita の記事でも、Numba が対応していない型や関数を使って Numba は Julia よりも遅いというような記事がしばしば見受けられます。そういう間違いをなくすためか、今年の年末か来年のはじめに公開される予定の Numba 0.47.0 から、「nopython」モードがデフォルトになり、Numba が対応していない型や関数があるとエラーになる予定です。それまでは、@jit(nopython=True) として「nopython」モードに設定して使うようにした方がいいです。
Numba は手軽に使えますが、コードを C ライクに書く必要があります。C/C++ を学習するまでの必要はありませんが Numpy は使えるようにしておく必要があります。その上で、使うときには、公式ドキュメントに「Numba 5分ガイド (5 minute guide to Numba)」 というページがあるので、少なくともそれは読んでおくようにしましょう。
以下が、「Numba 5分ガイド」の最初にあるコードです。Numba の特徴がよくわかります。
from numba import jit
import numpy as np
x = np.arange(100).reshape(10, 10)
@jit(nopython=True) # 最高のパフォーマンスを得るために「nopython」モードに設定、 @njit と書くことも可
def go_fast(a): # 関数は最初に呼ばれた時にマシンコードにコンパイルされる
trace = 0
for i in range(a.shape[0]): # Numba は、ループを好む
trace += np.tanh(a[i, i]) # Numba は、NumPy 関数を好む
return a + trace # Numba は、NumPy ブロードキャストを好む
print(go_fast(x))
Cython について
Cython は、ベンチマークの結果をみればわかるように C と同じ速さすることが可能です。でも、Numba と違って Python や Numpy の型や関数を使うと速くならないので、C/C++ の型や関数を使う必要があります。Cython は、実質的には C だと思って使ったほうがいいです。Cythonを使って便利なのは、Numpy の ndarray を memory views として扱うことで高速に各要素にアクセスできることです。
上に書いた「An Updated Analysis for the “Giving Up on Julia” Blog」の Parse Int で、Julia が 176 μs に対して、Cython は 378 µs 秒と 2 倍以上遅いという結果になっている理由は、hex, int という Python の関数を使用しているためです。C の関数に修正すると 3 倍近く速くなるので、Julia よりも Cython の方が速くなります。Qiita の記事等でも Python の関数を使っているため最適化ができていないケースは多いです。Cython がどのように Python のコードをコンパイルしているかは、-a オプションをつけてコンパイルするとわかります。詳細は、参考 Cython のコンパイルの確認 の方に書いておきます。
Cython を使う場合には、C を学習することが必須で、Numba を使うよりは、ハードルが高いです。一方で、Cython は、Pandas, Scikit-learn の開発に使われており、大規模開発での使用に適しています。
そうはいっても、Pandas の開発者である Wes McKinney 氏は、Pandas を Cython で開発するのに限界を感じて、C++ で Apache Arrow を開発中です。最近は C++ が使いやすくなってきているので、Cython ではなくて、C++ で開発し、Pybind11 等で Python に組み込むという開発スタイルを採用するケースも増えてきています。
Pythran について
Pythran は殆ど知られていませんが Python と Numpy のサブセットの科学技術計算用のコンパイラーです。Cython が Python や Numpy の関数を使うと速くならないのに対して、Python や Numpy の関数を一部ですが、それを C++ のコードに変換してコンパイルをしてくれます。C を知らなくても使えるというのが特徴です。ちょっとしたコードを軽量の実行可能バイナリーやモジュールにしたい時に便利です。
最後に
Python 自体は遅く、The Computer Language Benchmarks Game のベンチマークによると、C/C++よりも概ね10〜100倍遅いという結果になっています。一方で、Python は、機械学習、データサイエンス、科学技術計算という計算速度が要求されている分野で広く使われています。矛盾しているようですが、The Computer Language Benchmarks Game では、Python のライブラリーを使えないため、高速化のためのツールが豊富にある Python の実態が反映されていません。機械学習、データサイエンス、科学技術計算の分野であれば、Numoy, Pandas, Keras, TensorFlow, PyTorch等の豊富なライブラリを使うことで、Java や C# よりも使いやすいだけでなく、処理も速い場合が多いです。特に GPU が使用できるのであれば、Python が「速くて使いやすい言語」の No1 であることは間違いないでしょう。
また、スクリプト言語の名誉のために書いておきますが、スクリプト言語が遅いのは繰り返しの多い数値計算での話です。Web のプログラミングでは、このような数値計算をすることは少なく入出力の処理のウェートが高くなります。入出力の処理に関しては、スクリプト言語では多くの部分が C 言語を使って実装されているので、処理が遅いということはありません。現実に Web プログラミングでは、スクリプト言語が広く使われています。
どのような言語やライブラリーを学習したらいいかについては、各種の調査結果を参考にするのもいいと思います。
2018 Kaggle ML & DS Surveyによると、Q16 の日頃使用している言語(複数選択可)では、Python が 15711人で最も多くなっています。C/C++ の使用者は4383人で4番目に多く、C/C++ もかなり使われています。Python の場合は既存のライブラリを使う場合は便利ですが、新しいエンジンを作ろうとすると C/C++ が必要になります。
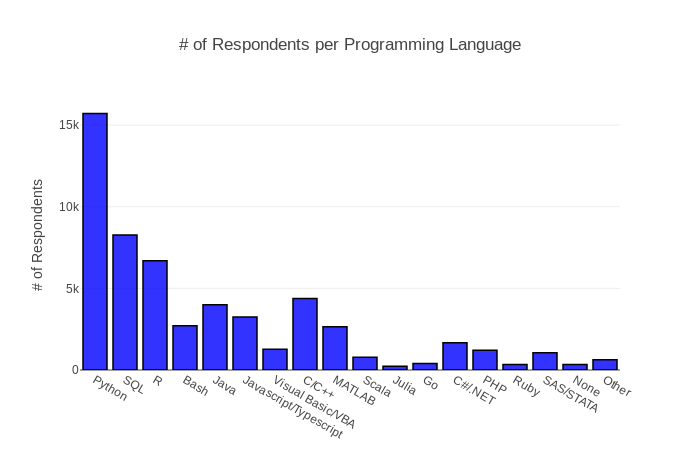
図は、2018 Kaggle Machine Learning & Data Science Surveyの Q16 から引用
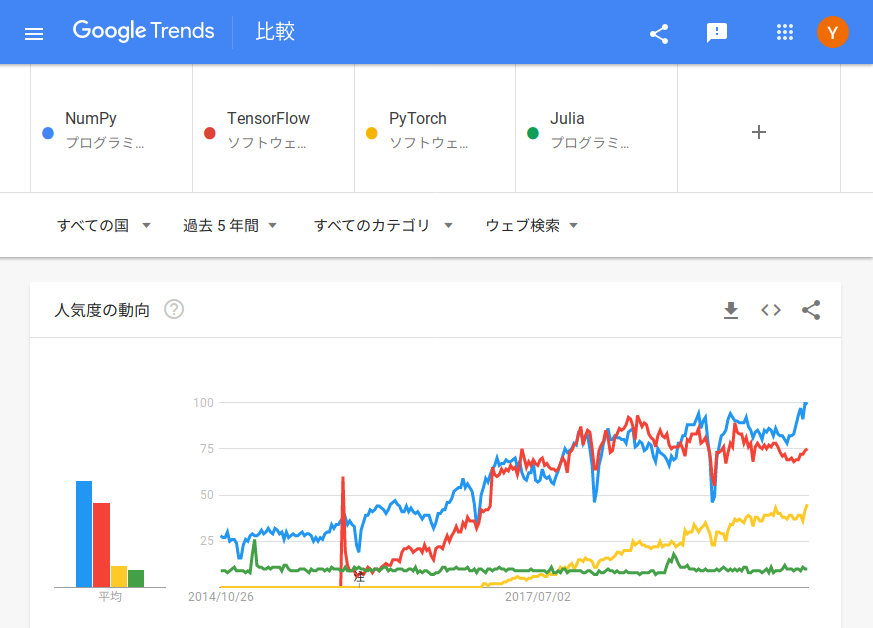
次に、Google トレンドを見てみましょう。Numpy は過去5年間で人気度が3〜4倍に増加しています。意外に感じますが、機械学習が人気で TensorFlow, PyTorch 等のソフトウェアの使用が急増したため、それらへのインターフェースとして利用が増えたと思われます。なお、PyTorch の行列演算は numpy like で操作が可能です。また、TensorFlow の行列演算は独自方式だったのですが、Python + Numpy のプログラムを GPU 用にコンパイルしてくれる JAX というソフトを Google が非公式ですが公開しています。Numpy を学習すれば、GPU を使う場合でもその知識は活用できます。
参考 Cython のコンパイルの確認
以下のように Cython を -a オプションを付けて実行すると。
%%cython -a
import numpy as np
import cython
@cython.boundscheck(False)
@cython.wraparound(False)
cpdef parse_int_vec_cython():
cdef:
long i,m
long[:] n
n = np.random.randint(0,2**31-1,1000)
for i in range(1,1000):
m = int(hex(n[i]),16)
実行が遅くなるコードが、黄色で表示されます。
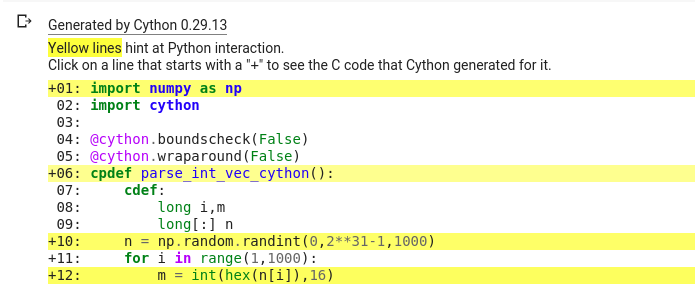
黄色で表示されたコードのうち、for ループ内にあって多数回実行されるコードから集中的に修正していくといいです。この場合だと m = int(hex(n[i]), 16) が一番の問題です。
該当のコードをクリックすると Cython がどうコンパイルされているかが表示されます。

赤の太字で表示されている部分が一番問題で、内容をみると下のように hex と int の処理でPyObject を呼んでいるのがわかります。
__pyx_t_1 = __Pyx_PyObject_CallOneArg(__pyx_builtin_hex, __pyx_t_2); if (unlikely(!__pyx_t_1)) __PYX_ERR(0, 12, __pyx_L1_error)
__pyx_t_1 = __Pyx_PyObject_Call(((PyObject *)(&PyInt_Type)), __pyx_t_2, NULL); if (unlikely(!__pyx_t_1)) __PYX_ERR(0, 12, __pyx_L1_error)
修正については、Python の関数をやめて C の関数を使うことです。hex の代わりには sprintf, int の代わりには strtol が使えるのですが、perf.c のコードをみて int については C の関数を使わずにコードで書きました。そのコードが以下です。
%%cython -a
from libc.stdio cimport sprintf
cimport cython
cpdef void parse_int_core(long[:] datain, long[:] dataout):
cdef:
long i
long num = datain.shape[0]
char s[11]
char c
long n
for i in range(num):
sprintf(s, "%lx", datain[i])
n = 0
for j in range(11):
c = s[j]
if c == b'\0':
break
if c > b'9':
n = 16 * n + c - 87
else:
n = 16 * n + c - 48
dataout[i] = n
このコードを実行すると以下のように表示されます。
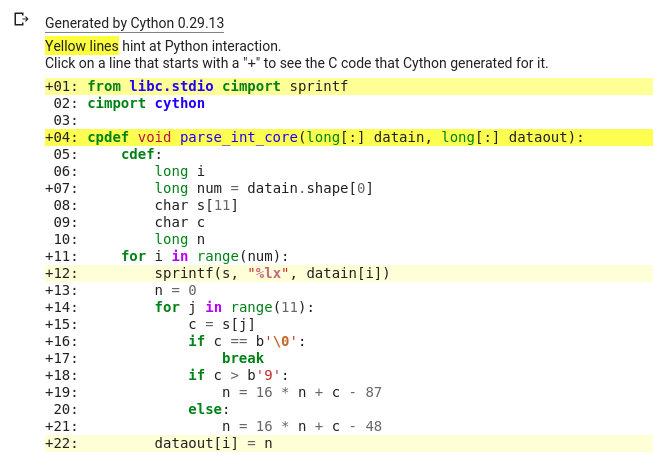
cpdef 以外のコードから、濃い黄色の部分が消えています。薄い黄色に付いては、datain, dataout にメモリービューを使っているためで、@cython.boundscheck(False), @cython.wraparound(False) のオプションを付けると薄い黄色も消えます。
numba の方は、hex の方もコードで書いたので、C よりも速くなりました。C 及び Cython も同じように sprintf を使わずにコードで書けば同じように速くなります。速くなる理由は、sprintf が汎用で使用するものに対して、パラーメータの内容が予めわかっているのでチェック等を省略できるためです。
このテスト項目をみていると Julia が C/C++ のように柔軟に使えるかは疑問。The Computer Language Benchmarks Game で binary-trees は Julia が C より 6 倍遅いということで、要するに動的にメモリー確保することが遅いのでその懸念は強いと思われます。