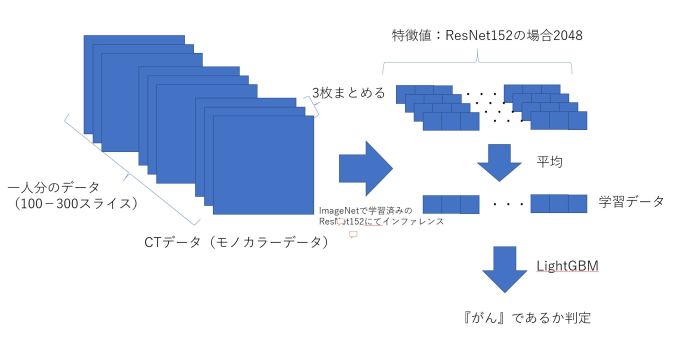
概略
Deep Learningの分野で早くから結果が出ている分野になります。がんであるかどうかは入力データを2つに分類すれば最低限よく機械学習としてもモデル化が単純にできます。一定のデータ量があれば、一からモデルを構築し使用するということが可能です。ただし、今回は転移学習として一般的なImageNetの画像認識モデルを使う手段をご紹介したいと思います。もともとは下記からなのですが、Cortana Ingelligenceのギャラリーに存在することにより、Azure MLでどうやって実行するのか等聞かれたことがあります。そのような事情からまずはこの内容に沿って解説したいと思います。
https://gallery.cortanaintelligence.com/Notebook/Medical-Image-Recognition-for-the-Kaggle-Data-Science-Bowl-2017-with-CNTK-and-LightGBM-1
Kaggleというデータサイエンティストのコミュニティーサイトがあり、そこでのコンテストでData Science Bowl 2017が実施されました。そこで提供されたテーマの一つとしてCT画像からのがん検知がありました。コンテストでは、実際のCTデータ1500人分程度が提供されそこから、分析ロジックを競うというものです。現状(2017年3月時点)のトップの認識率は99%を超えています。
https://www.kaggle.com/c/data-science-bowl-2017
方法
正確に分析しようとすると3次元の畳み込みが必要になりますがこの記事では、2次元の画像を少し無理やりですが、ImageNetのトレーニング済みのモデルを使います。この処理の概要を図に示します。
LightBGM学習は数秒で終わりますので、ほとんどがResNetモデルを使った特徴抽出の時間です。
事前準備
-CNTKの導入
以下を参照ください。CNTK2.0RC1を使っています。
https://analyticsai.wordpress.com/2017/04/04/cntk2-0b15%e3%82%a4%e3%83%b3%e3%82%b9%e3%83%88%e3%83%bc%e3%83%ab/
-データ
Kaggleからダウンロードします。サイズが大きいのでかなり時間がかかります。
https://www.kaggle.com/c/data-science-bowl-2017/data
-Python
CNTKは通常Anacondaの環境で動作しています。
CT画像を読み込むためのpydicomを導入します。pipで導入可能です。
pip install pydicom
-LightGBM
導入はこちらを参照ください
https://analyticsai.wordpress.com/2017/04/04/lightgbm/
-PreTrainedモデル
転移学習であるため、ImageNetのモデルが必要になります。CNTKはトレーニング済みのモデルが各種提供されており、自由にダウンロード可能になります。
https://www.microsoft.com/en-us/research/product/cognitive-toolkit/model-gallery/
この中からResNet152のモデルをダウンロードします。(こちらはCaffeのモデルからコンバートされたものになるようです)。
https://github.com/Microsoft/CNTK/tree/master/Examples/Image/Classification/ResNet#cntk-examples-imageclassificationresnet
オリジナルの記事はこちらからダウンロードするようです。
https://migonzastorage.blob.core.windows.net/deep-learning/models/cntk/imagenet/ResNet_152.model
フォルダ構造
\data\stage1\ --CTスキャンのデータ
\feartures\features0001 --スキャンデータから計算された特徴値事前にフォルダを作成しておく必要があります。
ResNet_152.modelはモデル以下のフォルダに格納します。
\local\models --ResNetのモデルファイルを格納
作業
stage1以下のフォルダにダウンロードしたCT画像データを展開します。
もともとのPythonコードはそのままでは動作しないので、下記に修正します。
正直色々修正しながら動かしました。
CNTKがRCになったことにより、途中のレイヤーの値はnumpy.arrayから、pythonのlistに変更されました。この部分は転移学習のコードには影響しますので、修正が必要になります。
この記事のままでは『誰』ががんなのか推定できますが、『どこ』がガンなのかわからない状態です。機械学習の場合は、予測することが目的となっていることから、『なぜこうなるのか?』という問いには答えられないことがほとんどです。場所を推定するのは別途方法を考える必要があります。この部分は次回に書きたいと思います。
一応コードは下記に添付します。上記サイトのコードを修正したものになります。
# Load libraries
import sys,os
import numpy as np
import dicom
import glob
from cntk.device import set_default_device, gpu
from matplotlib import pyplot as plt
import cv2
import pandas as pd
import time
from sklearn import cross_validation
from cntk import load_model
from cntk.ops import combine
from cntk.io import MinibatchSource, ImageDeserializer, StreamDef, StreamDefs
from lightgbm.sklearn import LGBMRegressor
print("System version:", sys.version, "\n")
#print("CNTK version:",pkg_resources.get_distribution("cntk").version)
#Put here the number of your experiment
EXPERIMENT_NUMBER = '0001'
# Put here the path to the downloaded ResNet model
MODEL_PATH='/local/models/ResNet_152.model'
# Put here the path where you downloaded all kaggle data
DATA_PATH='/'
# Path and variables
STAGE1_LABELS=DATA_PATH + 'stage1_labels.csv'
STAGE1_SAMPLE_SUBMISSION=DATA_PATH + 'stage1_sample_submission.csv'
STAGE1_FOLDER=DATA_PATH + 'stage1/'
FEATURE_FOLDER=DATA_PATH + 'features/features' + EXPERIMENT_NUMBER + '/'
SUBMIT_OUTPUT='submit' + EXPERIMENT_NUMBER + '.csv'
# Timer class
class Timer(object):
def __enter__(self):
self.start()
return self
def __exit__(self, *args):
self.stop()
def start(self):
self.start = time.clock()
def stop(self):
self.end = time.clock()
self.interval = self.end - self.start
def get_3d_data(path):
slices = [dicom.read_file(path + '/' + s) for s in os.listdir(path)]
slices.sort(key=lambda x: int(x.InstanceNumber))
return np.stack([s.pixel_array for s in slices])
def get_data_id(path, plot_data=False):
sample_image = get_3d_data(path)
sample_image[sample_image == -2000] = 0
if plot_data:
f, plots = plt.subplots(4, 5, sharex='col', sharey='row', figsize=(10, 8))
batch = []
cnt = 0
dx = 40
ds = 512
for i in range(0, sample_image.shape[0] - 3, 3):
tmp = []
for j in range(3):
img = sample_image[i + j]
img = 255.0 / np.amax(img) * img
img = cv2.equalizeHist(img.astype(np.uint8))
img = img[dx: ds - dx, dx: ds - dx]
img = cv2.resize(img, (224, 224))
tmp.append(img)
tmp = np.array(tmp)
batch.append(np.array(tmp))
if plot_data:
if cnt < 20:
plots[cnt // 5, cnt % 5].axis('off')
plots[cnt // 5, cnt % 5].imshow(tmp[0,:,:], cmap='gray')
cnt += 1
if plot_data: plt.show()
batch = np.array(batch, dtype='int')
return batch
def get_extractor():
node_name = "z.x"
loaded_model = load_model(MODEL_PATH)
node_in_graph = loaded_model.find_by_name(node_name)
output_nodes = combine([node_in_graph.owner])
return output_nodes
def calc_features(verbose=False):
net = get_extractor()
for folder in glob.glob(STAGE1_FOLDER+'*'):
foldername = os.path.basename(folder)
if os.path.isfile(FEATURE_FOLDER+foldername+'.npy'):
if verbose: print("Features in %s already computed" % (FEATURE_FOLDER+foldername))
continue
batch = get_data_id(folder)
if verbose:
print("Batch size:")
print(batch.shape)
feats = np.array(net.eval(batch))
if verbose:
print(feats.shape)
print("Saving features in %s" % (FEATURE_FOLDER+foldername))
np.save(FEATURE_FOLDER+foldername, feats)
def train_lightgbm():
df = pd.read_csv(STAGE1_LABELS)
x = np.array([np.mean(np.load(FEATURE_FOLDER+'%s.npy' % str(id)), axis=1).flatten() for id in df['id'].tolist()] )
y = df['cancer'].as_matrix()
print(x.shape)
trn_x, val_x, trn_y, val_y = cross_validation.train_test_split(x, y, random_state=42, stratify=y,
test_size=0.20)
clf = LGBMRegressor(max_depth=50,
num_leaves=21,
n_estimators=5000,
min_child_weight=1,
learning_rate=0.001,
nthread=24,
subsample=0.80,
colsample_bytree=0.80,
seed=42)
clf.fit(trn_x, trn_y, eval_set=[(val_x, val_y)], verbose=True, eval_metric='l2', early_stopping_rounds=300)
return clf
def compute_training(verbose=True):
with Timer() as t:
clf = train_lightgbm()
if verbose: print("Training took %.03f sec.\n" % t.interval)
return clf
def compute_prediction(clf, verbose=True):
df = pd.read_csv(STAGE1_SAMPLE_SUBMISSION)
x = np.array([np.mean(np.load((FEATURE_FOLDER+'%s.npy') % str(id)), axis=1).flatten() for id in df['id'].tolist()])
with Timer() as t:
pred = clf.predict(x)
if verbose: print("Prediction took %.03f sec.\n" % t.interval)
df['cancer'] = pred
return df
def save_results(df):
df.to_csv(SUBMIT_OUTPUT, index=False)
calc_features(verbose=True)
clf = compute_training(verbose=True)
df = compute_prediction(clf)
print("Results:")
df.head()
save_results(df)
結果がファイルに出力されます。
出力ファイルの一部です。
id,cancer
026470d51482c93efc18b9803159c960,0.40692197700808486
031b7ec4fe96a3b035a8196264a8c8c3,0.3037382335250943
03bd22ed5858039af223c04993e9eb22,0.2324142770435242
06a90409e4fcea3e634748b967993531,0.20775132462023793
07b1defcfae5873ee1f03c90255eb170,0.3054943422884507
0b20184e0cd497028bdd155d9fb42dc9,0.21613283326493204
12db1ea8336eafaf7f9e3eda2b4e4fef,0.2073817116259871
159bc8821a2dc39a1e770cb3559e098d,0.28326504404734154
174c5f7c33ca31443208ef873b9477e5,0.23168542729470515
1753250dab5fc81bab8280df13309733,0.21749273848859857
1cf8e778167d20bf769669b4be96592b,0.18329884419207843
1e62be2c3b6430b78ce31a8f023531ac,0.3276063617078295
1f6333bc3599f683403d6f0884aefe00,0.2092160345076576
1fdbc07019192de4a114e090389c8330,0.2276893908994118
2004b3f761c3f5dffb02204f1247b211,0.20776188822078875
例えば、idが、2703df8c469906a06a45c0d7ff501199の人は、0.5561665263809848という確率なので、かなり怪しいということがいえると思います。