はじめに
時系列データを扱っていると、
・データが一様な時間間隔でサンプリングされていない
・しかも多次元データ
な場合があります。
一様にサンプリングされていない多次元データを、任意のサンプリングレートで一気にリサンプリングする関数を作成しました。
開発環境
Python 3.5.2 Anaconda 4.2.0 (x86_64)
Mac OS X EI Capitan 10.11.6
まず時系列データを生成
# coding: UTF-8
# 各種必要なライブラリをインポート
import pandas as pd
import numpy as np
from datetime import datetime
from scipy import signal, interpolate
import matplotlib.pyplot as plt
collist=["cos","sin","tan"]
df = pd.DataFrame([np.zeros(3)],columns=collist,index=[0])
starttime = datetime.now()
for i in range(1000):
elapsed = (datetime.now() - starttime).total_seconds()
sin_df = pd.DataFrame({"sin" : np.sin(elapsed)},index = [elapsed])
df = df.append(sin_df)
elapsed = (datetime.now() - starttime).total_seconds()
cos_df = pd.DataFrame({"cos" : np.cos(elapsed)},index = [elapsed])
df = df.append(cos_df)
elapsed = (datetime.now() - starttime).total_seconds()
tan_df = pd.DataFrame({"tan" : np.tan(elapsed)},index = [elapsed])
df = df.append(tan_df)
今回はsin,cos,tanの3次元のデータを取り扱うこととしています。また、各データのタイムスタンプを、pandas.DataFrame()のindexとして保持しています。
多次元時系列データをリサンプリングする関数
def multidim_resample(df, Hz=100):
resample_df = pd.DataFrame()
for col in df.columns:
tmp_df = df[col].dropna()
#補間とリサンプリング
f = interpolate.interp1d(tmp_df.index,tmp_df)
resample_x = np.arange(tmp_df.index.min(),tmp_df.index.max(),1/Hz)
resample_y = f(resample_x)
#リサンプリングした結果をresample_dfに保存
resample_y= pd.DataFrame(resample_y, index=[resample_x],columns=[col])
resample_df = pd.concat([resample_df, resample_y], axis=1)
resample_df = resample_df.dropna()
return resample_df
各列を順番にリサンプリングし、最後にconcatしています。
今回は線形補間として、
f = interpolate.interp1d(tmp_df.index,tmp_df)
としましたが、引数を色々と変えることで、スプライン補間やらなんやらも使えるようです。
[scipy.interpolate.interp1d]
(https://docs.scipy.org/doc/scipy/reference/generated/scipy.interpolate.interp1d.html "scipy.interpolate.interp1d¶")
使ってみる
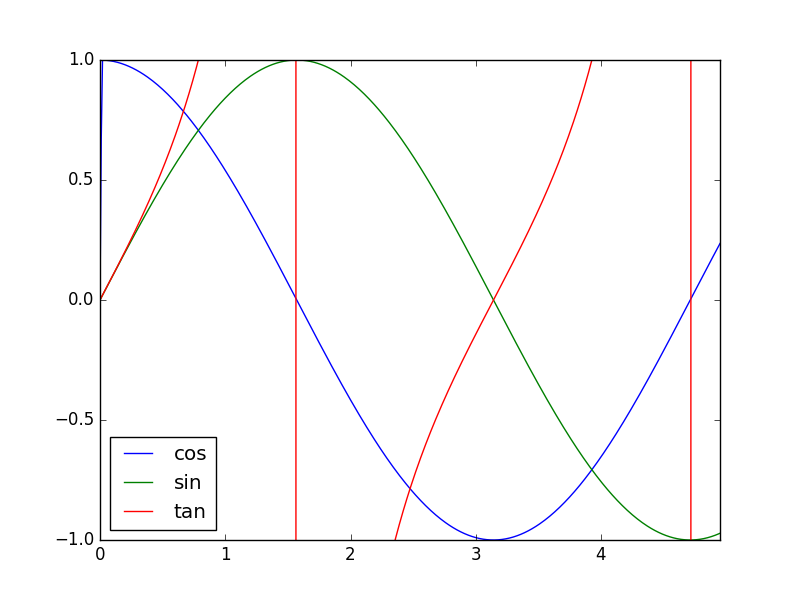
resample_df = multidim_resample(df,100)
resample_df.plot(ylim=[-1, 1])
plt.show()
いい感じで補間されたっぽいです。
まとめ
一様にサンプリングされていない多次元データを、任意のサンプリングレートで一気にリサンプリングする関数を作成しました。
サンプリングレートが1秒とかなら、pandas.DataFrame.resampleで一気にやっちゃうのが楽ですね。