はじめに
JMedRoBERTaは、日本語の医学論文で事前学習した言語モデルです。
詳細は、こちらを確認ください。
このJMedRoBERTaモデルを用いて、病名の分散表現を生成し、その特徴観察やちょっとした活用を行います。
その中で、病名の分散表現の活用へ理解を深めることが本記事の趣旨です。
・分散表現とは? - 分散表現の基本的な概念やその重要性、そして医療分野での応用について触れます。
・病名の分散表現の生成 - JMedRoBERTaを用いた病名の分散表現の生成方法について説明します。
・病名の分散表現の特徴観察 - JMedRoBERTaで生成された病名の分散表現が持つ特徴を観察します。
・病名の分散表現のちょっとした活用 - この分散表現を利用した関連病名検索をしてみます。
・病名の分散表現の実用的な活用について - ChatGPT(GPT-4)に病名の分散表現の活用方法を検討させます。
分散表現とは?
分散表現は、単語やフレーズを固定長のベクトルとして表現する方法です。このベクトルは、単語間の関連性から単語の意味を捉えることができます。そのため、ベクトル空間上で近い位置に配置される単語は、似た意味を持つと解釈できます。
単語の意味が似ていること、つまり、類似した分散表現を持つことに対しては、もう少し説明が必要です。医療分野を例に補足します。
例えば、"咳"と"頭痛"は、どちらも風邪の症状なので、類似した分散表現を持ちます。また、"肺炎"と"咳"という単語は、ある病気とそれに伴う症状という関連があるので、こちらも分散表現が似ます。他には、"はい炎"と"肺炎"も同じ病気を指しているので、こちらも似た分散表現を持つでしょう。このように「似る」について、単語のどことどこが近い意味を持つのか、色々なバリエーションがあります。
医療分野で、この分散表現を使うことで、「似る」の使い分けはややこしいものの、病名や症状、治療法などの関連性が把握できると期待できます。
病名の分散表現の生成
分散表現は、Word2Vecのモデルの重みやBERTの隠れ層から取得できます。後者のBERT由来のものは、BERTに与えたインプットの文脈を加味して、単語や文章の意味をとらえることができます。
今回は、BERTを利用し、病名のみをインプットにし、その分散表現を得ます。
BERTの説明は、Webや書籍などに、優れたものが多数存在していますので、ここでは割愛します。
病名には、ICD10対応標準病名マスターの病名表記を用います。
それでは、以下に分散表現の生成のためのコードを記載します。
※以下のコードの動作確認は、Google Colabolartory上で行っています。
ライブラリのインストール
!pip -q install transformers[ja] datasets
ICD10対応標準病名マスターをCSVで読み込み
以下のリンクから取得したマスタをcsv形式でGoogleDriveに配置しました。
ColabolartoryのストレージをGoogle Driveにマウントすることで読み込めるようにしています。
import pandas as pd
filename_prefix = '/content/drive/MyDrive/work/ICD10分散表現/'
header = pd.read_csv(filename_prefix + 'ttl_main.csv', encoding='cp932', header=None).values.tolist()[0]
df_main = pd.read_csv(filename_prefix + 'nmain512.csv', encoding='cp932', names=header)
JMedRoBERTaモデルのセットアップ
JMedRoBERTaモデルは、4種類HuggingFace上で提供されています。
トークナイザーについて、文字列の分割方法に選択肢が2つあり、その語彙数にも選択肢が2つあるので、計4種類あります。
※釈迦に説法かもしれませんが、トークナイザーは入力を要素(単語など)に分割し、要素に数字を割り当てるもので、トークナイザーで作成した要素のことトークンと呼びます。分割方法は、PreTraining用の教師データなどから、最適な分割方法をアルゴリズムにより決定します。
2種類のトークナイザーについての説明は、こちらに記載があります。引用します。
万病 WordPiece トークナイザは,MeCab2) を用いて入力文を単語列に分かち書きした後, WordPiece [17] によりサブワード化を行うものであ る.MeCab の辞書選択においては,通常用いられ る ipadic に加えて,大規模な病名辞書である万病 辞書 [18] を使う.これにより,病名が分かち書き の時点で複数の単語列に分割されることを防ぐ.
一方,SentencePiece トークナイザは,SentencePiece [19](Unigram モデル)により,文から直接サブワー ド化を行うものである.出現頻度の高い文字列はひ とかたまりのトークンとして処理されるため,医学 論文において頻度の高い医学用語は,分割されずに 1 トークンとして扱われることが期待される.
今回は、病名のみをモデルのインプットとするので、病名が分かち書きされにくい、万病WordPieceトークナイザを用いることにします。語彙数は50000語のものを用います。
HuggingFaceに記載のモデルの利用方法に倣って、モデルに加えて、万病辞書も読み込みます。
# download Manbyo-Dictionary
!mkdir -p /usr/local/lib/mecab/dic/userdic
!wget https://sociocom.jp/~data/2018-manbyo/data/MANBYO_201907_Dic-utf8.dic && mv MANBYO_201907_Dic-utf8.dic /usr/local/lib/mecab/dic/userdic
!ls /usr/local/lib/mecab/dic/userdic
from transformers import AutoModelForMaskedLM, AutoTokenizer, AutoConfig
model_name = "alabnii/jmedroberta-base-manbyo-wordpiece-vocab50000"
model_config = AutoConfig.from_pretrained(model_name, output_hidden_states=True)
model = AutoModelForMaskedLM.from_pretrained("alabnii/jmedroberta-base-manbyo-wordpiece-vocab50000", config=model_config)
print(model)
print(model_config)
model.eval()
tokenizer = AutoTokenizer.from_pretrained("alabnii/jmedroberta-base-manbyo-wordpiece-vocab50000")
分散表現を抽出するため、modelのconfigでoutput_hidden_statesをTrueに設定しています。
分散表現の生成
tokenizerで各病名をトークン化し、それをPytorchのDataLoaderに直接渡せるDatasetに変更しています。
from torch.utils.data import DataLoader
from datasets import Dataset
# https://huggingface.co/docs/datasets/use_with_pytorch
# を参考にした
byoumei_list = df_main['病名表記'].values.tolist()
inputs = tokenizer.batch_encode_plus(byoumei_list, return_tensors='pt', padding=True)
ds = Dataset.from_dict({"input_ids": inputs['input_ids'], "token_type_ids": inputs['token_type_ids'], "attention_mask": inputs['attention_mask']}).with_format("torch")
作成したDatasetをDataLoderで読み込み、バッチでBERTによるエンコード処理を行います。
BERTの最終隠れ層の出力からCLSトークンの分散表現を抽出し、これを入力とした病名の分散表現とみなします。
この処理は、Colabolatoryのフリープラン、GPUなしで1hぐらいかかりました。一方で、ColabolatoryのフリープランでGPUにT4を利用すると、batch_size = 64としたとき、5分程度で終了しました。
batch_size = 256 #任意
dataloader = DataLoader(ds, batch_size=batch_size)
# GPUが利用可能ならGPUにデータを移動
device = torch.device('cuda' if torch.cuda.is_available() else 'cpu')
model = model.to(device)
word_vec_from_CLS_list = [] #CLSトークンのエンコードは、インプット全体の分散表現を表しているらしい。
for batch in dataloader:
# print(batch)
batch ={key:value.to(device) for key, value in batch.items()}#辞書
with torch.no_grad(): # 勾配計算なし
outputs = model(**batch)
last_hidden_state = outputs[1][-1] # 最終層の隠れ状態
for i in range(len(batch['input_ids'])):
word_vec_from_CLS = last_hidden_state[i][0]
word_vec_from_CLS_list.append(word_vec_from_CLS.cpu().numpy().tolist())
作成した分散表現を外部に保存します。
# CSVファイルに分散表現を保存する
import csv
f = open(filename_prefix + 'word_vec_from_CLS_list.csv', 'w', newline='')
data = word_vec_from_CLS_list
writer = csv.writer(f)
writer.writerows(data)
f.close()
参考までにCSVに保存した分散表現をメモリに読み込むためのコードも記載します。
# CSVファイルから分散表現を読み込む
import csv
with open(filename_prefix + 'word_vec_from_CLS_list.csv') as f:
reader = csv.reader(f)
l = [row for row in reader]
word_vec_from_CLS_list_loaded = [[float(v) for v in row] for row in l]
病名の分散表現の特徴の観察
このセクションでは、JMedRoBERTaモデルから得られた分散表現の特徴を可視化してみます。そして、この分散表現が医療ドメインの知識を反映していることを確かめるために、早稲田大学が開発したドメイン特化でない日本語RoBERTaと比較します。
分散表現は、768次元のベクトルであるため、これを可視化するためには次元削減を行います。次元削減の手法としては、PCA(主成分分析)を採用します。
from sklearn.decomposition import PCA
# 分散表現
vectors = np.array(word_vec_from_CLS_list_loaded)
# PCAで次元を768次元から2次元に削減します。
pca = PCA(n_components=2)
pca_result = pca.fit_transform(vectors)
可視化にあたり、各病名には、ICDコードの大分類に応じて、ラベルを設定することにします。
大分類は、ICD10 国際疾病分類第10版(2013年版)を参照ください。
各病名にラベルを割り当てるためのコードが以下になります。
# ラベルを取得します
raw_labels = df_main['ICD10‐2013'].values
numeric_labels = []
def is_in_range(label, start, end):
"""指定された範囲にラベルが含まれるかどうかを確認するヘルパー関数"""
return start <= label <= end
def assign_numeric_value(label):
"""ラベルを数値にマッピングするヘルパー関数"""
# レンジとそれに紐づく数字のマッピング
ranges = {
"A00-B99": 1,
"C00-D48": 2,
"D50-D89": 3,
"E00-E90": 4,
"F00-F99": 5,
"G00-G99": 6,
"H00-H59": 7,
"H60-H95": 8,
"I00-I99": 9,
"J00-J99": 10,
"K00-K93": 11,
"L00-L99": 12,
"M00-M99": 13,
"N00-N99": 14,
"O00-O99": 15,
"P00-P96": 16,
"Q00-Q99": 17,
"R00-R99": 18,
"S00-T98": 19,
"V01-Y98": 20,
"Z00-Z99": 21,
"U00-U99": 22
}
# 与えられたラベルがどのレンジに一致するかをチェック
for range_, number in ranges.items():
start, end = range_.split("-")
if is_in_range(label, start, end):
return number
# どのレンジにも一致しない場合
print('レンジ不一致発見', label, 23)
return 23
for i, raw_label in enumerate(raw_labels):
# raw_label が文字列で、長さが3以上の場合のみ処理を行います
if isinstance(raw_label, str) and len(raw_label) >= 3:
# ラベルの先頭3文字を取得
first_three = raw_label[:3]
numeric_labels.append(assign_numeric_value(first_three))
else:
print('UNK発見', i, raw_label, 23)
numeric_labels.append(23)
print(numeric_labels)
# プロットようにラベルの一意なリストを作成し、それをソートします
unique_labels = sorted(list(set(numeric_labels)))
print(unique_labels)
出力結果 ICDコードが割り当たっていない病名がいくつかあるようです。
UNK発見 841 nan 23
UNK発見 5580 nan 23
UNK発見 8126 nan 23
UNK発見 9489 nan 23
UNK発見 9495 nan 23
UNK発見 10852 nan 23
UNK発見 10921 nan 23
UNK発見 12776 nan 23
UNK発見 14793 nan 23
UNK発見 14919 nan 23
UNK発見 14922 nan 23
UNK発見 14925 nan 23
UNK発見 16526 nan 23
UNK発見 16872 nan 23
UNK発見 19889 nan 23
UNK発見 20281 nan 23
UNK発見 21076 nan 23
UNK発見 21135 nan 23
UNK発見 23655 nan 23
UNK発見 23819 nan 23
UNK発見 23923 nan 23
[4, 4, 17, 4, 17, 17, 17, 17, 7, 7, 10, 7, 11, 4, 4, 4, 4, 4, 4, 4, 4, 4, 4, 4, 4、・・・省略
[1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23]
全病名の分散表現をプロットしてみます。
# 必要なライブラリのインポート
import matplotlib.pyplot as plt
plt.figure(figsize=(16, 9))
scatter = plt.scatter(pca_result[:, 0], pca_result[:, 1], c=numeric_labels, cmap=plt.cm.get_cmap('jet', len(unique_labels)), s=1)
legend1 = plt.legend(*scatter.legend_elements(num=len(unique_labels)), title="Classes")
plt.gca().add_artist(legend1)
plt.title('PCA')
plt.show()
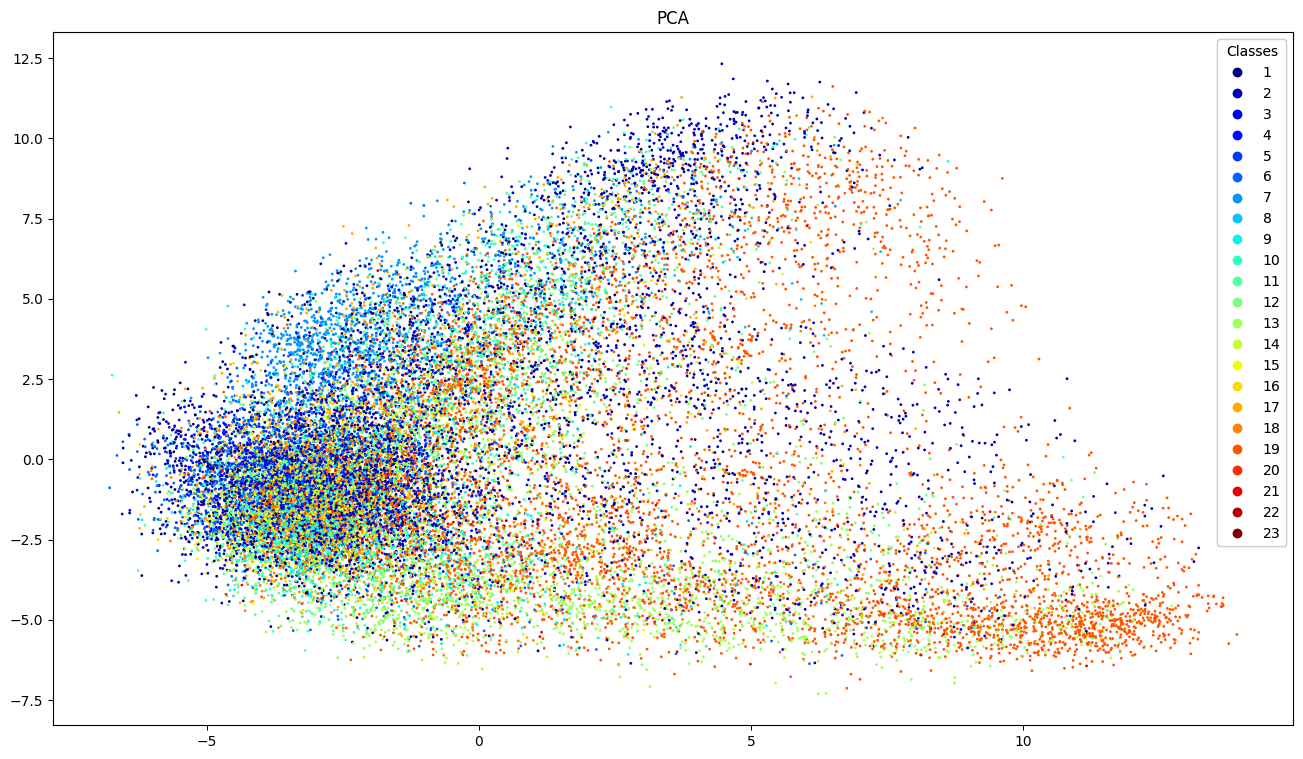
図1に示す散布図が得られました。ラベルの数字とICD10コードの大分類の対応は、上記のassign_numeric_value関数を確認ください。縦横の軸は、主成分分析の第一主成分、第二主成分であり、その意味合いは残念ながら不明瞭です。
図1 JMedRoBERTaにより取得した病名の分散表現
さて、データを見てみますと、左下に主として濃い青(ラベル1~4)のデータがに凝集しており、右側にオレンジ(ラベル17~19)のデータが広範囲に広がっているように見えます。「く」の字に緑(ラベル11~13)のデータが広がっているのも確認できます。24個のラベルのすみわけを分析するのは難しいですが、ある色のデータが多く散布している場所(各ラベルの分布の偏り)があるようです。
とはいえ、この画像だけだと考察が難しいので、早稲田大学が開発したドメイン特化でない日本語RoBERTa(早大RoBERTa)でも同じことをやってみます。結果だけを表示します。
図2に示す散布図が得られました。ラベルと軸の説明は、JMedRoBERTaの結果と同じになります。
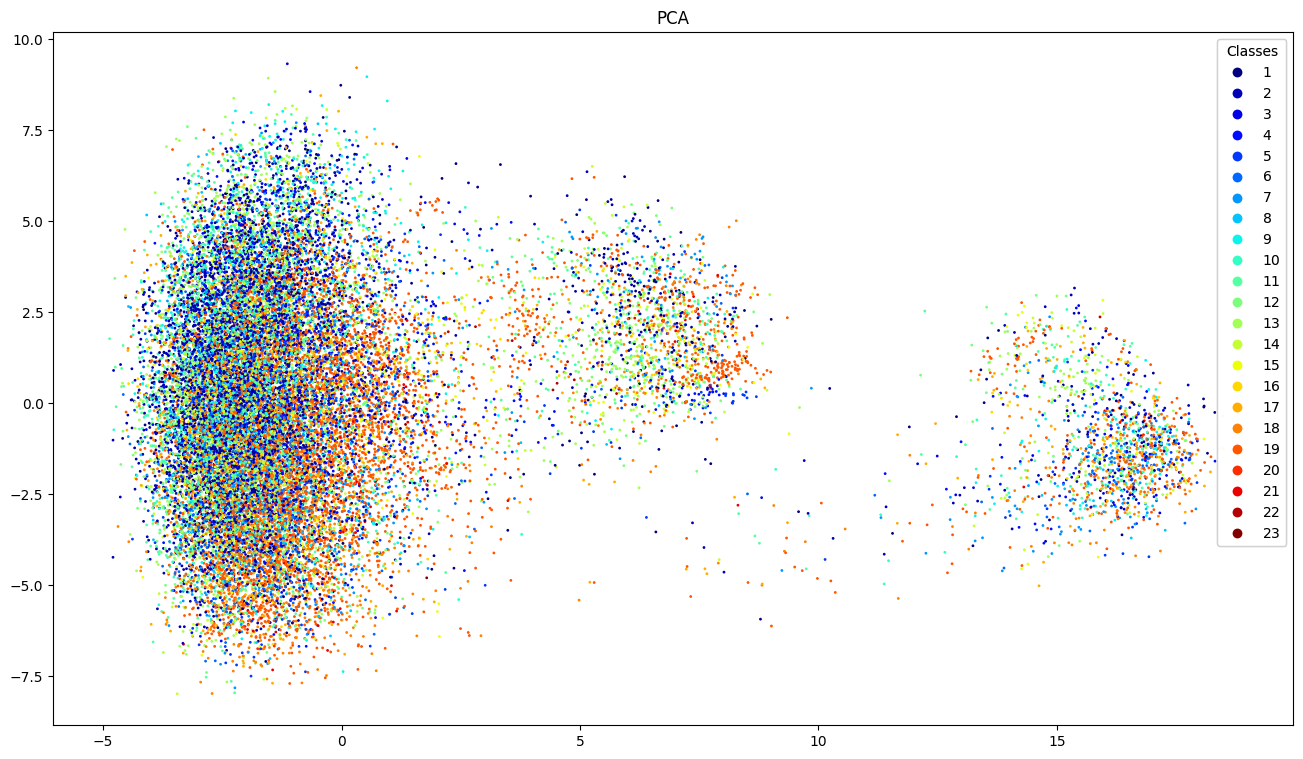
図2 早稲田大RoBERTaにより取得した病名の分散表現
まず、分布の形状がJMedRoBERTaの結果と大きく異なり、3つの島ができました。加えて、各ラベルの分布には偏りがあまり見られず、どの島にも青、オレンジ、緑のデータが散布している様子が確認できました。比較すると、JMedRoBERTaの方がまだ各ラベル、つまり、ICD10の大分類ごとのすみわけができているように見えます。
本記事の「分散表現とは?」のセクションで、単語が「似る」には、複数の意味合いがあることを述べました。JMedRoBERTaは、医学論文の知識を蓄えることで、ICD10を分類するのに役立つ「似る」という感覚を磨けているのだと考えます。
病名の分散表現のちょっとした活用
ICD10対応標準病名マスターを基にJMedRoBERTaで作成した病名の分散表現から、インプットに対して最も類似度するものを検索したいと思います。意味で検索しているので、検索対象のフォーマットが自由です。最近、親知らずを抜歯し、歯の痛みが気になっていたので、その思いをインプットにぶちまけることにしました。
from sklearn.metrics.pairwise import cosine_similarity
import numpy as np
# 検索対象の名前
input_name = '親知らず抜歯後の歯の先の痛み'
# 検索対象の分散表現を取得
inputs = tokenizer([input_name], return_tensors='pt', padding=True)
with torch.no_grad(): # 勾配計算なし
inputs ={key:value.to(device) for key, value in inputs.items()}
outputs = model(**inputs)
last_hidden_state = outputs[1][-1] # 最終層の隠れ状態
word_vec_from_CLS = last_hidden_state[0][0].cpu().numpy().tolist()
selected_vector = word_vec_from_CLS
vectors = word_vec_from_CLS_list_loaded # ICDマスタの病名の分散表現
# 検索対象の分散表現とICDマスタの病名の分散表現とのコサイン類似度を計算します。
similarities = cosine_similarity([selected_vector], vectors)
# コサイン類似度が高い順にソートし、そのインデックスを取得します。
indices = np.argsort(-similarities[0])
# 最も類似度が高い5つのベクトルのインデックスを表示します。
top5_indices = indices[0:5].tolist()
print(input_name, "に似た病名を探します。")
print("Top 5 similar vectors' indices:")
for index in top5_indices:
print('Index:', index, '病名', byoumei_list[index])
以下、その結果です。
親知らず抜歯後の歯の先の痛み に似た病名を探します。
Top 5 similar vectors' indices:
Index: 24590 病名 放散性歯痛
Index: 12379 病名 神経痛性歯痛
Index: 22893 病名 非定型歯痛
Index: 4849 病名 顎痛
Index: 8488 病名 咬合性外傷
安易な判断はもちろんできませんが(本当に問題あれば病院に行きましょう)、こんな病気が関連してるかもな~と知ることができます。この路線から実用化を目指すのであれば、症状から病名を当てるタスクでFTする必要がありそうです。JMedRoBERTaの論文で、そのようなタスクに対してモデルの評価が行われていたので、気になる方はご参照ください。
ちなみに。。。
放散性歯痛 目,耳など隣接する臓器の疾患により歯に痛みを感じるもの
非定型歯痛 歯や歯肉、抜歯した部位を中心とした不規則で慢性持続性の疼痛症
とのことです。
病名の分散表現の実用的な活用について (Generated by ChatGPT(GPT4))
病名の分散表現は、医療の多様なタスクにおいて実用的な価値を持っています。分散表現は、病名や症状、治療法などの医学的な情報をベクトルとして捉えることで、これらの情報間の関連性や特性を瞬時に分析することができます。
診断支援:病名の分散表現を使用することで、患者の症状や過去の医療履歴に基づき、関連する病名や疾患を迅速に特定することができます。これにより、医師はより正確な診断を迅速に行うことができるようになります。
医学論文の検索と分析:病名の関連性や類似性を基にして、関連する医学論文や研究を高速に検索することができます。また、新しい治療法や研究結果を迅速にキャッチアップするのに役立ちます。
患者情報の解析:病名の分散表現を利用して、患者の診療記録や医療履歴を分析することで、疾患の進行や治療の効果を評価することができます。
医薬品の推薦:病名のベクトルと医薬品のベクトルを比較することで、最も関連性が高い医薬品や治療法を推薦することができます。
このように、病名の分散表現は、医療現場の効率性や精度を向上させるための強力なツールとして活用されています。今後も、この技術の発展により、さらに多岐にわたる応用が期待されています。
おわりに
医療ドメインは、なかなか利用できるデータセットがなく(見つけられていないだけかもですが)、個人でアクセスできるもので何にトライできるのか、そこを考えるのが大変でした。面白いデータセットがあればぜひ共有いただければ幸いです!!!