本チュートリアルはLinux PCで行われることを想定しています。また、コマンドプロンプトやcondaなどの基本的な使い方については全く触れません。
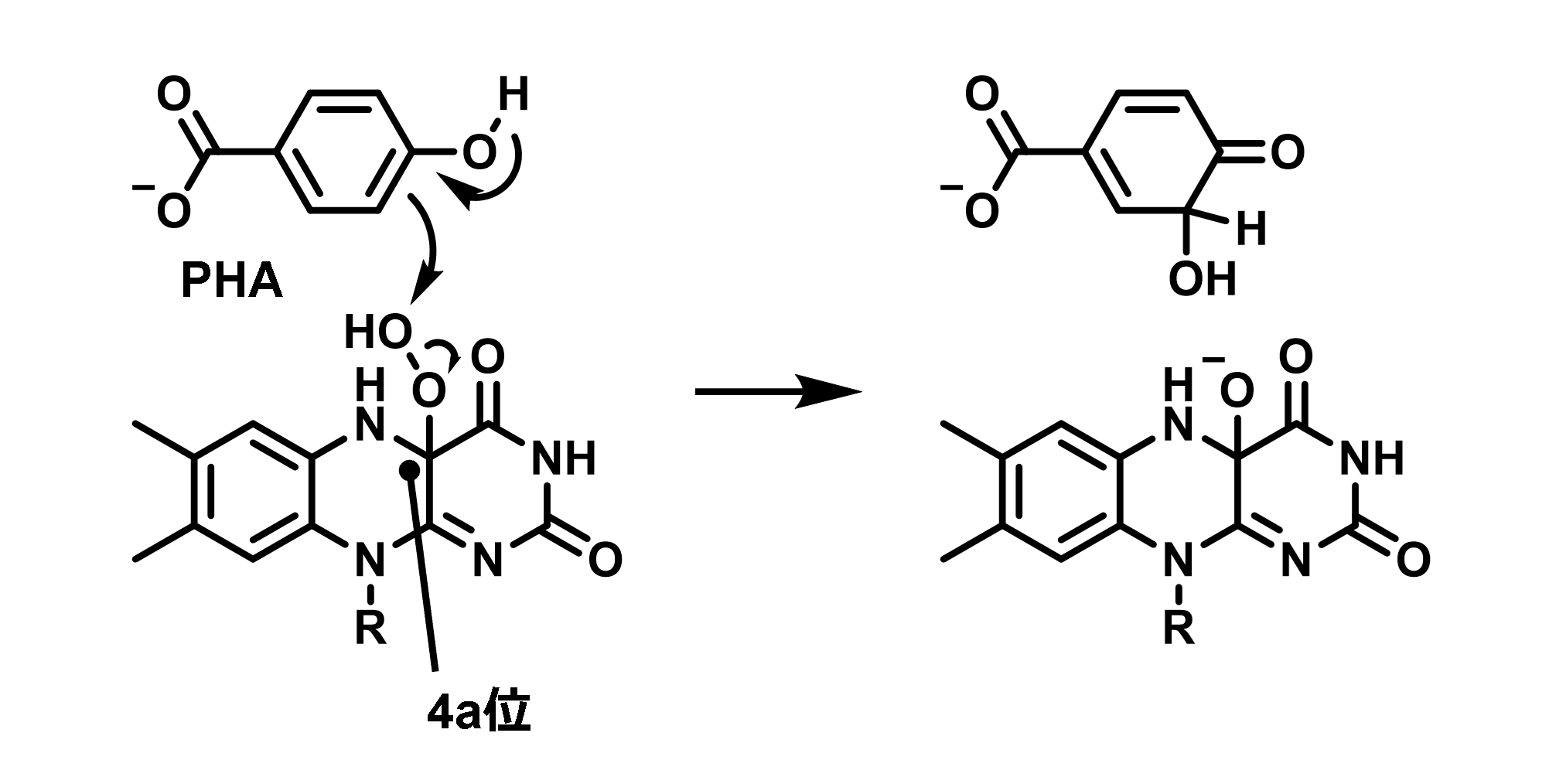
本チュートリアルではPobA(p-hydroxybenzoate hydroxylase)のalpha-fold model(AF model)とFAD、p-hydroxybenzoate(PHA)との複合体モデルを作成します。PobAはPHAの3位炭素を水酸化するFAD依存性の酸化酵素です(図1)。PobAを対象として選定した理由は以下の2つです。
- PobA-FAD-PHA複合体のX線結晶構造が解かれている
- FADとPHAとでligandの異なる扱い方を学ぶことができる
1. AF modelをダウンロード
AlphaFold Protein Structure DatabaseからPobAのAF modelをダウンロードします。ダウンロードしたPDBファイルのファイル名をPobA.pdbに変更しておきます。
2. FADについて
基質-酵素複合体モデルを作成するにあたり、基質の結合部位(基質ポケット)を予想する必要があります。一般に基質ポケットの予想はホモログ酵素や変異実験の結果を考慮して行います。
PobAの反応にはFADが関与するため、FADの近傍に基質PHAが結合すると予想できます。そこで、FAD → PHA の順でポケットを予想してみます。
2-1. FADポケットを予想
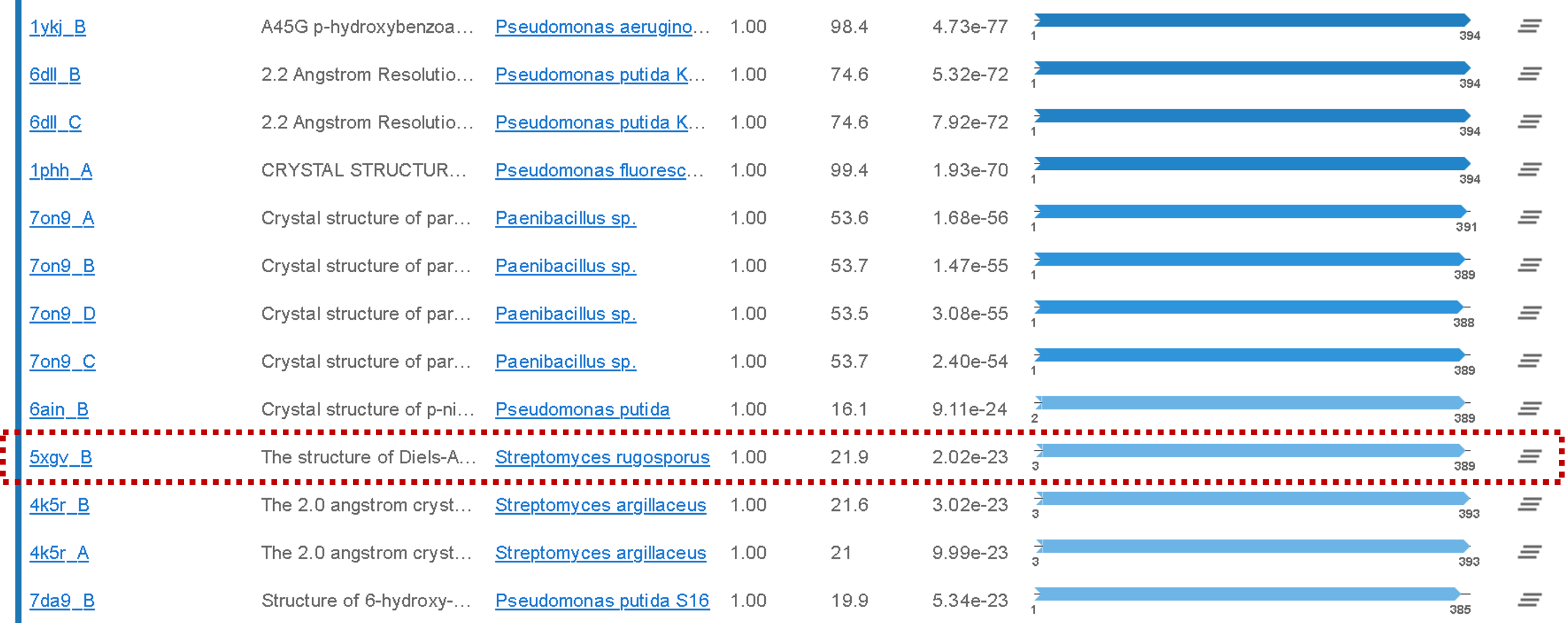
Foldseekを用いて構造ベースでホモログ酵素を検索し、それらの情報からFADポケットを予想してみます。FoldseekのページでUPLOAD PDBボタンからPobA.pdbをアップロードし、Databases & search settingsでPDB100以外の✔を外した後、Serchボタンをクリックします。結果を見ると、配列相同性が非常に高いホモログ酵素が上位にランクインしています。それらのFADポケットを参考にしても良いのですが、今回はあえて少し配列相同性の低いホモログ酵素を参考にしてみます(図2)。一つずつ構造を確認していくと、5xgvにFADが含まれていることがわかります。
2-2. FADを配置
5xgvはpyrroindomycinsの生合成に関与するPyrE3(Diels-Alderase)です。PobAのFADポケットとPyrE3のそれとが同じであると仮定し、PobA-FAD複合体の作成を試みます。
PyrE3に結合したFADをPobAへ移植するために、これら2つのタンパク質を重ね合わせる必要があります。重ね合わせにはPyMOLのalign機能が利用できますが、Foldseekのような構造ベースで検索されたホモログ酵素は、配列相同性が高くないために、綺麗に重ならないことが多いです(図3)。そこでTM-alignを利用して、構造ベースでタンパク質を重ね合わせます(図4)。
- PDBから5xgvをダウンロードする
- PyMOLで
5xgv.pdbを開く -
5xgv → A → remove watersにより水分子を削除する - Command Input Areaに
sele chain Bと入力しEnter -
(sele) → A → extract objectにより5xgvのB鎖を抽出する -
5xgv → A → delete objectにより5xgvのA鎖を削除する -
File → Export Molecule… → Save…によりファイル名PyrE3_chainB.pdbとして保存する -
TM-alignのページで2つの参照ボタンから
PobA.pdb→PyrE3_chainB.pdbの順でアップロードする - Run TM-align ボタンをクリックする
- 頭文字がGのPDBファイルをダウンロードする
- ファイル名を
PobA_vs_PyrE3.pdbに変更する
図3. PyMOL alignでの重ね合わせ(灰色:5xgv、水色:PobA)
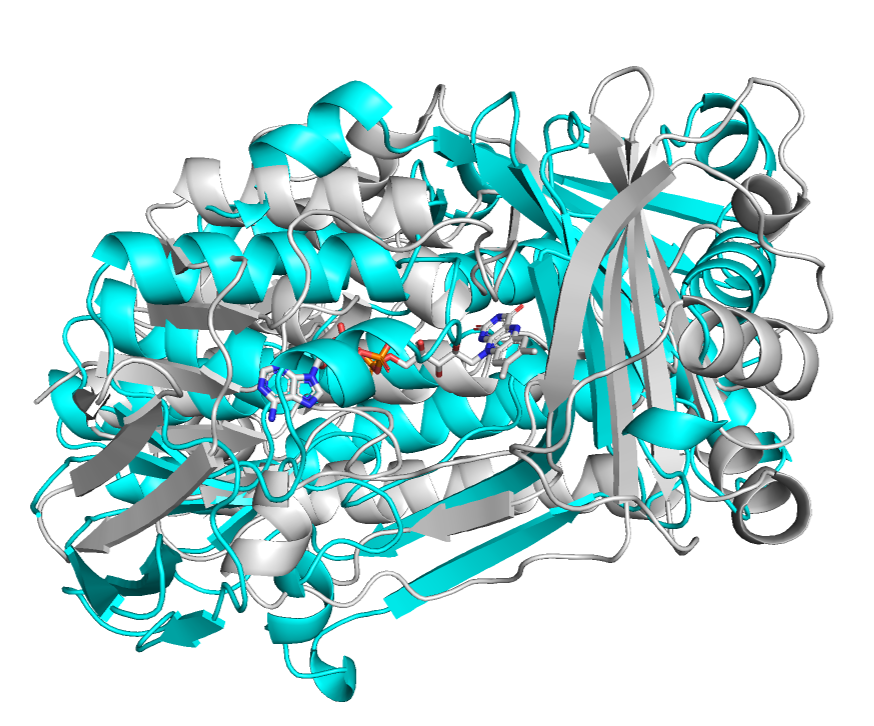
図4. TM-alignでの重ね合わせ(灰色:5xgv、緑色:PobA)
続いてPyrE3に結合したFADをPobAに移植します。
- PyMOLで
PobA_vs_PyrE3.pdbを開く - Command Input Areaに
sele chain Aと入力しEnter -
(sele) → A → extract objectによりPobAを抽出する - FADをクリックする
-
(sele) → A → extract objectによりFADを抽出する -
PobA_vs_PyrE3 → A → delete objectによりPyrE3を削除する -
File → Export Molecule… → Save…によりファイル名PobA_FAD.pdbとして保存する
2-3. FADを評価
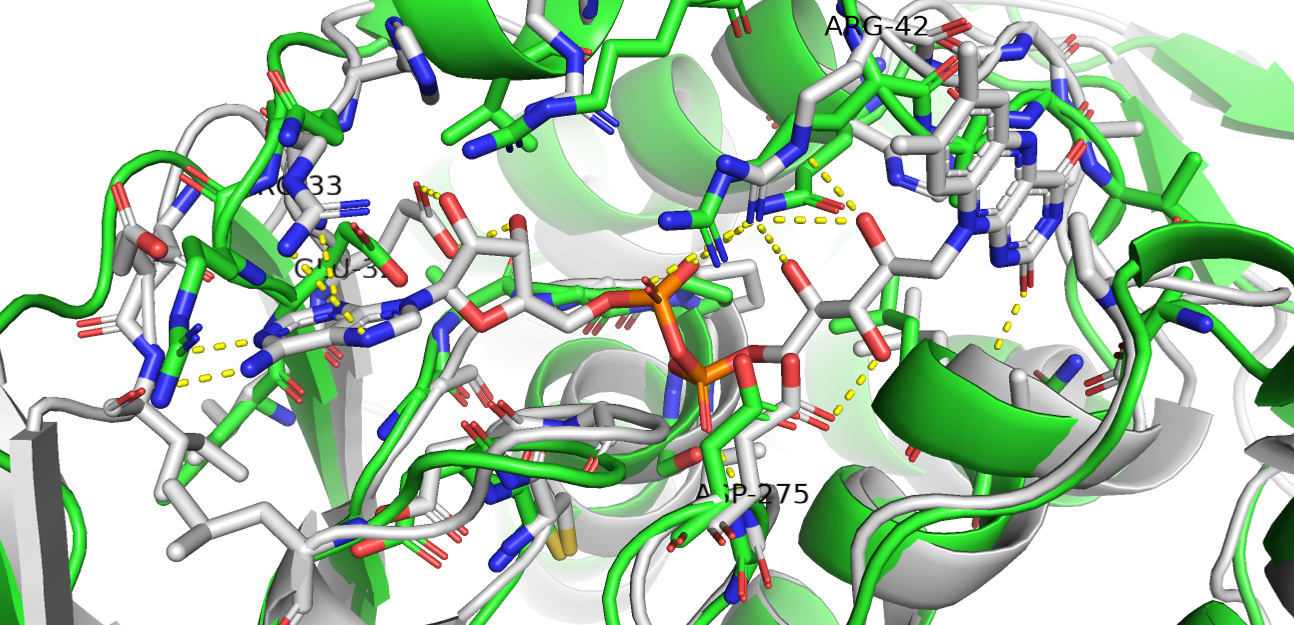
PyrE3に結合したFADをPobAに移植した構造が妥当であるかを評価します。それにあたり、PyrE3とFADとの相互作用に関与するアミノ酸残基がPobAにおいても保存されているのかを確認します。
- PyMOLで
PobA_vs_PyrE3.pdbを開く - Command Input Areaに
sele chain Aと入力しEnter -
(sele) → A → extract objectによりPobAを抽出する - FADをクリックする
-
(sele) → A → find → polar contacts → to other atoms in objectによりFAD-PyrE3間の相互作用を表示する -
(sele) → A → modify → around → residues within 4 AによりFADの4Å以内にあるアミノ酸残基を選択する -
(sele) → S → licoriceにより表示する
PyrE3においてFADと相互作用するGLU32、ARG33、ARG42、ASP275が、PobAにおいても保存されていることがわかります(図5)。このことから、PyrE3に結合したFADをPobAに移植した構造は妥当であると判断します。
図5. FAD周辺のアミノ酸残基(灰色:5xgv、緑色:PobA)
3. PHAについて
FADを配置できたので、次にPHAを配置します。Foldseekの結果(6ju1、1d7lなど)から、PHAもFADと同様の手法で配置できそうですが、今回はあえてドッキングによりPHAを配置してみます。
3-1. PHAをドッキング
初めにPHAの立体構造を取得します。PyMOL等で自作できますが、今回はPubChemからダウンロードします。
1.PHAの立体構造をPubChemからSDF形式でダウンロードする
2.PyMOLでダウンロードしたSDFファイルを開く
3.all → A → hydrogens → removeにより水素原子を削除する
4.File → Export Molecule… → Save…によりファイル名PHA.pdbとして保存する
続いてAutodock Vinaを利用し、PHA.pdbをligand、PobA_FAD.pdbをreceptorとしてドッキングします。この作業はコマンドプロンプトで行います。なお仮想環境mgltoolsにはmgltoolsが、仮想環autodockにはAutodock vinaがインストールされています。
# 1. 仮想環境mgltools入る
conda activate mgltools
# 2. PobA_FAD.pdbをpdbqt形式に変換(receptor preparation)
pythonsh /path/to/envs/mgltools/MGLToolsPckgs/AutoDockTools/Utilities24/prepare_receptor4.py \
-r PobA_FAD.pdb \
-o receptor.pdbqt \
-U nphs_lps_waters \
-A hydrogens \
-v
# 3. PHA.pdbをpdbqt形式に変換(ligand preparation)
pythonsh /path/to/envs/mgltools/MGLToolsPckgs/AutoDockTools/Utilities24/prepare_ligand4.py \
-l PHA.pdb \
-o ligand.pdbqt \
-A hydrogens \
-v
# 4. auto dock toolsでgrid boxを設定する
adt
- 画面右下の”Remind Me Later”が赤くなり次第、クリックする
-
File → Read Moleculeでreceptor.pdbqtを開く -
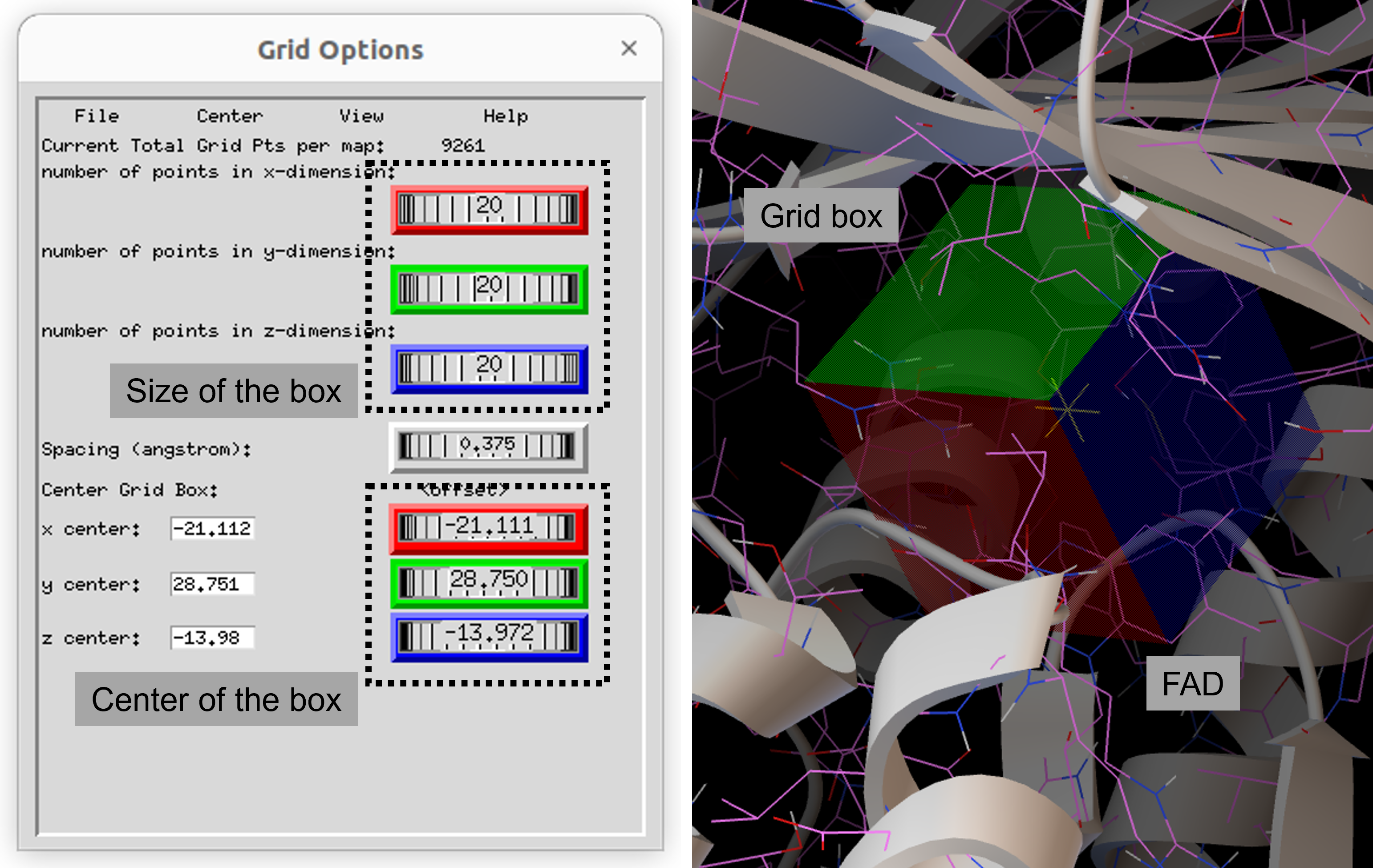
Grid → Grid Box...でgrid boxを表示する - grid boxの位置をFADのC4位付近まで移動し、サイズを調整する
- メモ帳でconfig_dockを開き、grid boxの位置とサイズとを書き込む(図6)
#CONFIGURATION FILE (options not used are commented)
#INPUT OPTIONS
receptor = receptor.pdbqt
ligand = ligand.pdbqt
#CHANGE THE FOLLOWING DATA WITH YOUR BOX DIMENSIONS
#Size of the box (values bxf, byf and bzf)
size_x = 20
size_y = 20
size_z = 20
#SEARCH SPACE CONFIGURATIONS
#CHANGE THE FOLLOWING DATA WITH YOUR BOX CENTER COORDINATES
#Center of the box (values bxi, byi and bzi)
center_x = -21.111
center_y = 28.750
center_z = -13.972
#OUTPUT OPTIONS
#out =
#log =
#OTHER OPTIONS
cpu = 4
exhaustiveness = 8
num_modes = 10
# 5. 仮想環autodockに入る
conda activate autodock
# 6. config_dockの条件でドッキングを実行する
vina --config config_dock --out output.pdbqt
ドッキングが正常に終了するとoutput.pdbqtが出力されます。
3-2. PHAを評価
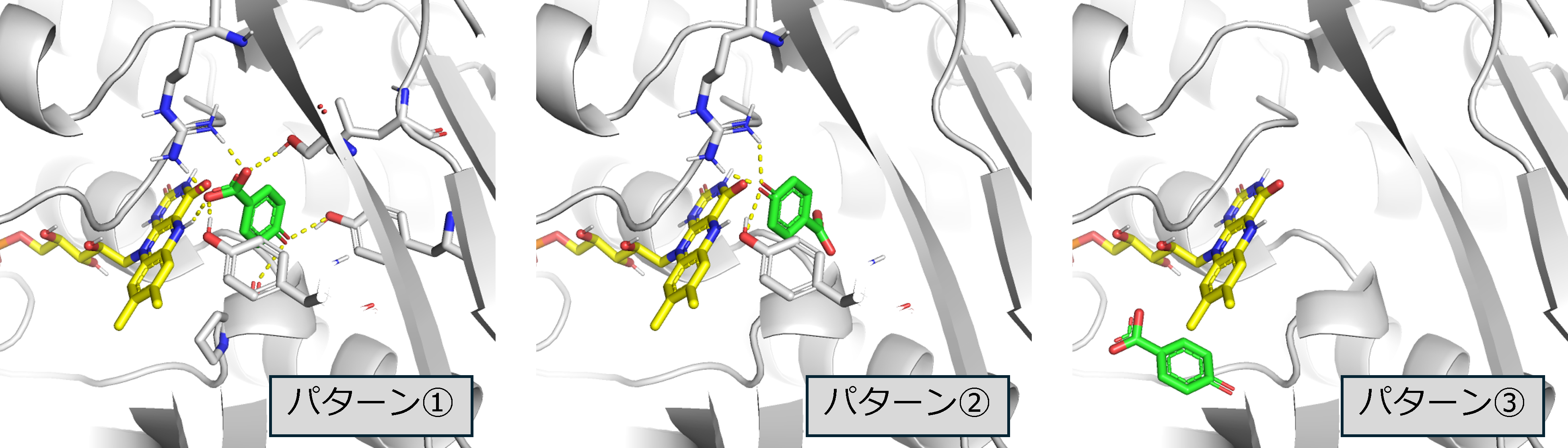
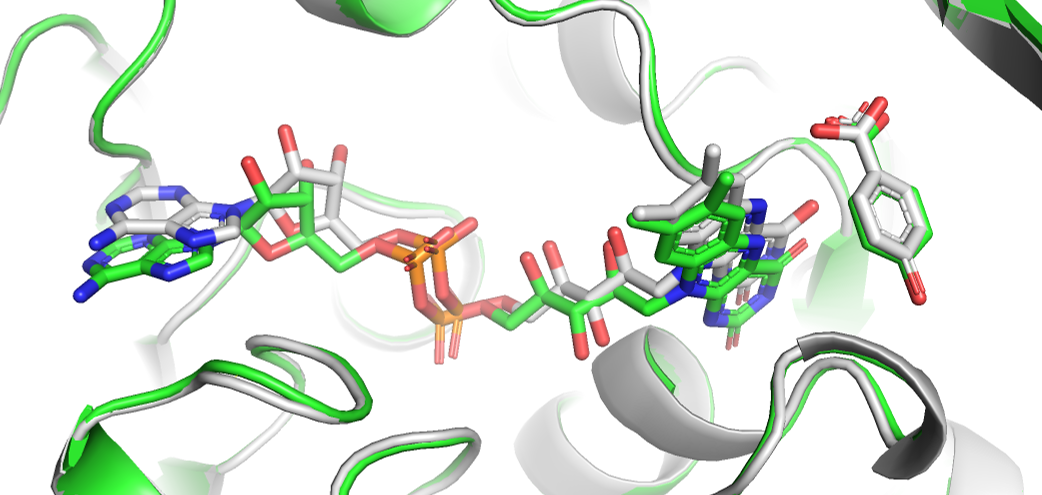
PobA_FADにPHAをドッキングした構造が妥当であるかを評価します。PyMOLでreceptor.pdbqtとoutput.pdbqtとを開き、ドッキング結果を確認します。PyMOLの画面右下ある➤ボタンをクリックして一つずつ構造を確認したところ、ドッキング結果を3パターンに大別できることがわかりました(図7)。パターン①、②ではPHAの反応点とFADのC4a位とが近いことがわかります。また①は、PHAのカルボン酸とPobAのARGが相互作用している点から、②に比べてより合理的だと考えます。これらのことから、①がPobA_FADにPHAをドッキングした構造として妥当であると判断します。この構造をExport Molecule…からPobA_FAD_PHA.pdbとして保存します。
4. 答え合わせ
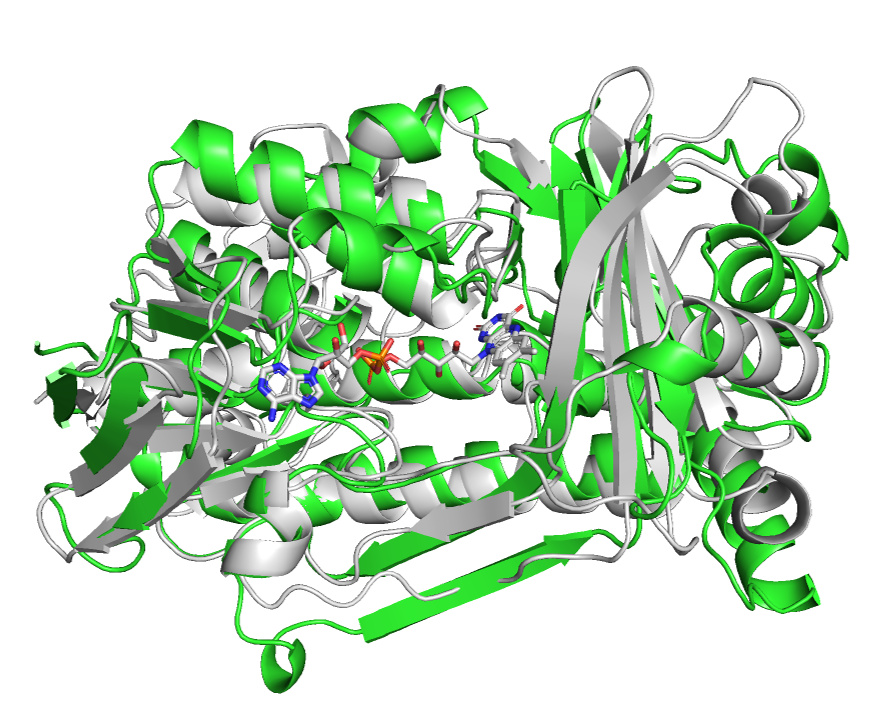
最後に、作成したモデルPobA_FAD_PHA.pdbとX線結晶構造とを比較します。両者をPyMOLのalign機能で重ね合わせると、良く一致することがわかります(図8)。
図8. PobA_FAD_PHA.pdbとX線結晶構造との重ね合わせ(灰色:5xgv、緑色:PobA)
参考
これらのWebページが大変参考になります。併せてご参照ください。