結果
data = !ls *.dat
pd.DataFrame({d: pd.read_table(d, index_col=0).squeeze() for d in data})
よくある実験データ取込シチュエーション
実験データなんか同じような条件で何度も実験して、 横軸が同じで縦軸とファイル名が異なるファイルをいくつも取り込む ような作業を何度も繰り返します。
varR_Gain_C15n_R100.dat
varR_Gain_C15n_R1000.dat
varR_Gain_C15n_R10000.dat
varR_Gain_C15p2n_R10.dat
varR_Gain_C2718n_R10.dat
varR_Gain_C2718n_R100.dat
varR_Gain_C2718n_R1000.dat
varR_Gain_C2718n_R10000.dat
varR_Gain_C4718n_R10.dat
varR_Gain_C4718n_R100.dat
varR_Gain_C4718n_R1000.dat
varR_Gain_C4718n_R10000.dat
varR_Gain_C71n_R10.dat
varR_Gain_C71n_R100.dat
varR_Gain_C71n_R1000.dat
varR_Gain_C71n_R10000.dat
ファイルの中身は0行目が横軸、1列目が縦軸です。
Freq. V(out)
7.52+005 -2.47e+001
7.79+005 -2.61e+001
8.06+005 -2.75e+001
8.35+005 -2.89e+001
8.64+005 -3.04e+001
8.95+005 -3.18e+001
9.26+005 -3.32e+001
9.59+005 -3.46e+001
9.93+005 -3.60e+001
1.00+006 -3.63e+001
これまではpandas read_table()関数を使って一個一個読み込んで、最後にDataFrameに結合していました。
このようなファイルたちを一発で読み込みたいのです。
そこで編みだしたディクショナリとsqueeze()メソッドを合わせる技
files = !ls *.dat
pd.DataFrame({f: pd.read_table(f, index_col=0).squeeze() for f in files})
filesはファイル名を格納するリストになります。ipython上で!lsと打ってshellのlsコマンドの結果をリスト型としてfilesオブジェクトに格納します。
{f: pd.read_table... for f in files}はディクショナリ形式にする内包表記で、filesの中身を一つ一つfに入れていって、f(一個のファイル名)をカラム名に、カラムをpd.read_tableで読み込んだpd.DataFrameオブジェクトにします。
squeeze()はあまり使わないかもしれませんが、DataFrame型をSeries型に変えるメソッドです。
!lsの代わりにglobを使えば真のワンライナーになります。
from glob import glob
pd.DataFrame({f: pd.read_table(f, index_col=0).squeeze() for f in glob('/path/to/files/*.dat')})
インポートが必要な時点で、
!lsと変わらず2行必要なのでは!?
タイプ数が!lsよりglobの方が多いのでは!?
でもglobなら相対パス、絶対パスがcdしなくても使えるので、汎用的ではある。
いつでも使えるコマンドに
pandas.read_table()の拡張版をprofile_default/startup/に.ipyとして保存します。
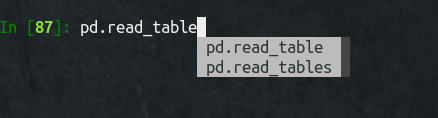
ipythonやjupyter notebookを開いたときに以下のスクリプトが自動実行されるので、次回からはpd.read_tTabでpd.read_tablesが予測に表示されます。
表示されることはすなわち、pandasのメソッドとして認識されているということです。しかもスタートアップとして登録したので、pandasさえインポートされていれば、read_tables()用のimport文を書かずに済みます。
def _read_tables(*files, **kwargs):
""" Read general delimited files into DataFrame
Super function of `pd.read_table()`
"""
return pd.DataFrame(
{f: pd.read_table(f, **kwargs).squeeze() for f in files})
setattr(pd, 'read_tables', _read_tables) # pd.read_tables()として使えるようにする
キーワード引数**kwargsをつけてやることで、pandas read_table()関数のすべての引数が扱えます。
以上のようにして取り込んだpd.read_tables()を使うときは、dataのリストをスター*をつけて展開し、引数のindex_col=0を入れてあげます。
data = !ls *.dat
df = pd.read_tables(*data, index_col=0)
from glob import iglob
df = pd.read_tables(*iglob('./*.dat'), index_col=0)
glob()を使っても良いですが、iglobはイテレータとして読み込むので、私は好み的にiglob()の方が好きです。
リストやイテレータで読み込む場合は*を忘れずにつけて可変長引数として展開してあげましょう。
またはデータファイルパスを直入力もできます。
df = pd.read_tables(
'varR_Gain_C71n_R100.dat',
'varR_Gain_C71n_R1000.dat',
index_col=0)
取り込んだDataFrameのcolumnsはファイル名になります。
Freq. varR_Gain_C15n_R100.dat varR_Gain_C15n_R1000.dat
779 -26.1 -26.0
806 -27.5 -27.4
835 -28.9 -28.9
864 -30.4 -30.3
895 -31.8 -31.7
926 -33.2 -33.1
959 -34.6 -34.6
993 -36.0 -36.0
100 -36.3 -36.3