PyTorchを使ってLSTMでコロナ陽性者数を予測してみる
はじめに
概要
PyTorchを使ってLSTMネットワークでPCR検査結果が陽性となった人の日別の人数を予測するモデルを作成しました。
モデルを作成するためのコードと予測結果を紹介します。
学習データには厚生労働省オープンデータと気象庁の気象データを利用しています。
学習データに使う特徴量は日毎のPCR検査結果が陽性の人の数、東京の平均気温、PCR検査実施人数の3次元です。予測するのは日毎のPCR検査結果が陽性の人の人数です。最終的には、このような予測結果が出ます。
2020/10/31から2020/11/30までのPCR検査結果が陽性の人の数を予測して、実際のデータと比較したグラフです。
実行環境はGoogle Colabを想定しています。
背景
最近PyTorchを覚えたのですが、どこかにアウトプットして頭を整理したいと思いました。
また、PyTorchはニューラルネットワークの構築とコーディングがとても直結していて書きやすいと感じました。この体験を少しでも広めて、この分野に少しでも貢献できればいいなとも思っています。
いろいろ実装が怪しかったり、非効率なところがあるかもしれませんが、その際は、ぜひご指摘いただけると嬉しいです。
対象読者
- PyTorch初心者の方
- LSTMで時系列データを使った予測をやってみたい方
簡単にLSTMについて
LSTMはRNNの発展系で、短期/長期の傾向の情報を学習できたり、不要な傾向の情報を忘れたり、どれくらい覚えるかを調整するLSTM層が中間層としてあります。情報をどれくらい取り入れるかだったり、忘れるかだったりはtanhやシグモイド関数を利用しています
それでは、データを準備して、コードを説明していきます。
データ準備
データはそれぞれ以下から入手します。
| 特徴量 | 入手先 |
|---|---|
| PCR検査陽性者数/日 | https://www.mhlw.go.jp/stf/covid-19/open-data.html |
| 東京の平均気温/日 | https://www.data.jma.go.jp/gmd/risk/obsdl/# |
| PCR検査実施人数/日 | https://www.mhlw.go.jp/stf/covid-19/open-data.html |
ダウンロードした後、それぞれ以下の形になるように手で加工します。
- PCR検査陽性者数/日

- 東京の平均気温/日

- PCR検査実施人数/日

- **それぞれのデータは2020/1/16から2020/11/30までとなっています。**始まりの2020/1/16はPCR検査陽性者数が記録された最初の日付に合わせています。
- PCR検査実施人数は2/4以前のデータはなかったので、0で埋めました。
- 本記事で構築するLSTMは予測したいPCR検査陽性者数/日に加えて、東京の平均気温/日とPCR検査実施人数/日も学習データとして入力します。
- このデータを今回使うGoogle Colabのアカウントに紐づくGoogle Driveにアップロードします。
コード紹介
Google Colabを使ってコードを書いていきます。ランタイムのタイプはGPUにしましょう。
ライブラリimport
まずは今回使用するライブラリをimportします。
import torch
import torch.nn as nn
import torch.optim as optim
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
%matplotlib inline
device変数の作成
後でTensorに送るデバイスを定義しておきます、
device = 'cuda' if torch.cuda.is_available else 'cpu'
Google Driveをマウント
データ準備で準備した3つのデータファイルをGoogle Driveにアップロードします。ここではGoogle Driveの直下に置くことを想定しています。
from google.colab import drive
drive.mount('/content/drive')
データをロード
Google Driveにアップロードしたデータ準備で準備した3つのデータファイルをロードします。
# PCR陽性者数データ
# 厚生労働省オープンデータを利用
# https://www.mhlw.go.jp/stf/covid-19/open-data.html
data1 = pd.read_csv('/content/drive/My Drive/pcr_positive_daily.csv')
# 東京都の平均気温データ
# 気象庁のデータを利用
# https://www.data.jma.go.jp/gmd/risk/obsdl/#
data2 = pd.read_csv('/content/drive/My Drive/covid19_temperature.csv')
# PCR検査実施人数データ
# 厚生労働省オープンデータを利用
# https://www.mhlw.go.jp/stf/covid-19/open-data.html
data3 = pd.read_csv('/content/drive/My Drive/pcr_tested_daily.csv').fillna(0)
3つのデータファイルを結合
ロードした3つのデータファイルを結合して1つのデータファイルにします。
# 3つのデータを結合する
data4 = pd.merge(data1,data2)
data = pd.merge(data4, data3)
結合したデータを確認
データのshapeは(全日数, (日付,PCR 検査陽性者数(単日),東京平均気温,PCR 検査実施件数(単日)))です。
print(data.shape)
print(data.tail(5))
以下のように出力されるかと思います。
(320, 4)
日付 PCR 検査陽性者数(単日) 東京平均気温 PCR 検査実施件数(単日)
315 2020/11/26 2499 12.8 42634.0
316 2020/11/27 2510 11.6 43351.0
317 2020/11/28 2674 11.8 22307.0
318 2020/11/29 2041 10.1 11975.0
319 2020/11/30 1429 9.9 41335.0
全データ数を取得
全データの数をlen_dataに入れます。
len_data = data.shape[0]
結合したデータから必要な特徴量を抽出
必要な特徴量はPCR 検査陽性者数(単日), 東京平均気温, PCR 検査実施件数(単日)の3つです。
covid19_data = data[['PCR 検査陽性者数(単日)', '東京平均気温', 'PCR 検査実施件数(単日)']]
特徴量を時系列にグラフ表示
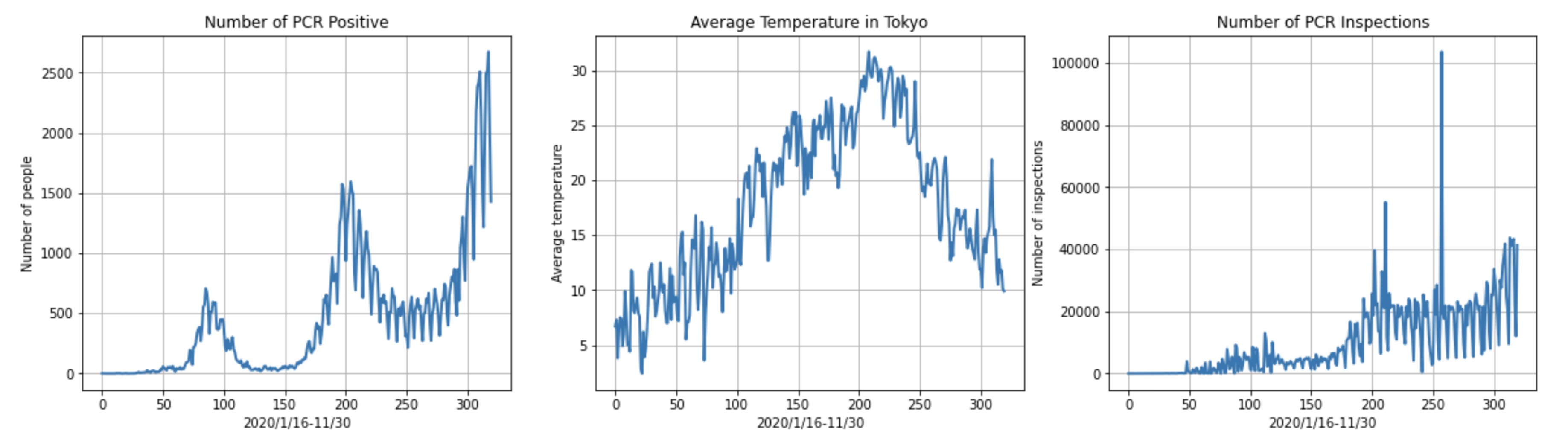
左からPCR 検査陽性者数(単日), 東京平均気温, PCR 検査実施件数(単日)をグラフ表示します。x軸は2020/1/16から数えて320日分のindex番号になっています。
fig, (axL, axM, axR) = plt.subplots(ncols=3, figsize=(20,5))
axL.plot(covid19_data['PCR 検査陽性者数(単日)'], linewidth=2)
axL.set_title('Number of PCR Positive')
axL.set_xlabel('2020/1/16-11/30')
axL.set_ylabel('Number of people')
axL.grid(True)
axM.plot(covid19_data['東京平均気温'], linewidth=2)
axM.set_title('Average Temperature in Tokyo')
axM.set_xlabel('2020/1/16-11/30')
axM.set_ylabel('Average temperature')
axM.grid(True)
axR.plot(covid19_data['PCR 検査実施件数(単日)'], linewidth=2)
axR.set_title('Number of PCR Inspections')
axR.set_xlabel('2020/1/16-11/30')
axR.set_ylabel('Number of inspections')
axR.grid(True)
fig.show()
float型に変換
covid19_data = covid19_data.values.astype(float)
covid19_data.shape
特徴量は日付がなくなり3つになっています。
(320, 3)
学習データとテストデータに分割する
直近の30日をテストデータにして、これ以前を学習データにします。
# 直近30日をテストデータにする
test_data_size = 30
train_data = covid19_data[:-test_data_size]
test_data = covid19_data[-test_data_size:]
学習データを正規化
特徴量を時系列にグラフ表示に示した通り、割とデータには開きがあります。なので、最小値と最大値を決めた正規化を学習データに行います。
# データセットの正規化を行う。最小値0と最大値1の範囲で行う。
from sklearn.preprocessing import MinMaxScaler
scaler = MinMaxScaler(feature_range=(0, 1))
train_data_normalized = scaler.fit_transform(train_data)
Tensor型に変換
PyTorchを使うのでTensor型にします。
# Tensor型に変換
train_data_normalized = torch.FloatTensor(train_data_normalized)
シーケンスデータ作成関数を定義
LSTMに学習させるデータは時系列情報を持たせる必要があリます。**この関数の目的はnum_sequence日分にまとめてあげることです。**イメージは下のような感じです。
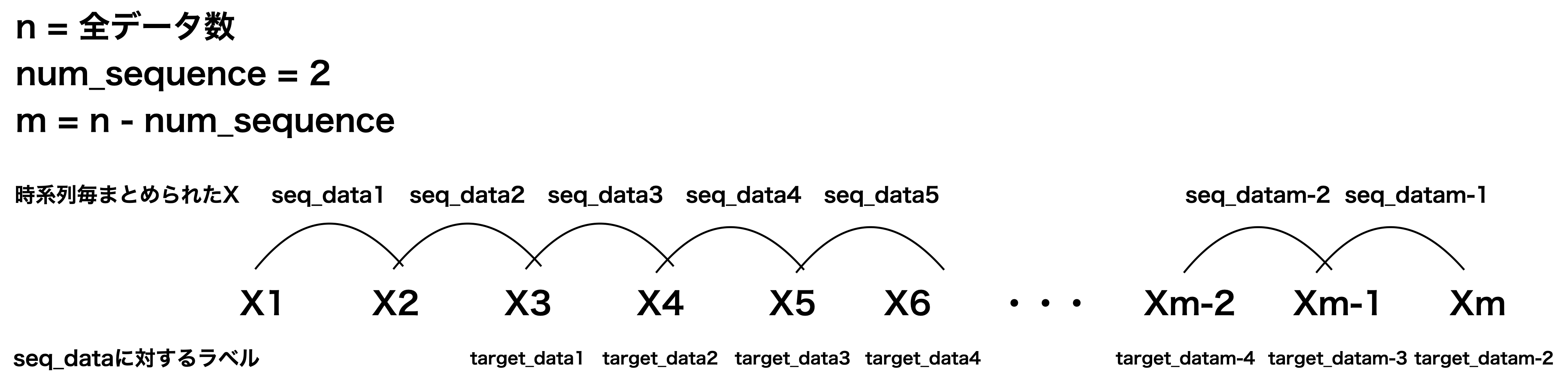
上の図はnum_sequence=2の例です。seq_dataにはnum_sequence分のデータを入れて、target_dataにはseq_dataの次の日のデータを入れます。seq_dataをLSTMにインプットして、LSTMからのアウトプット(予測値)とtarget_dataを比較して学習させていきます。
num_sequence日分のデータ(seq_data)を使ってその次の日のデータ(target_data)を予測しようとしています。
target_dataに入れるのは特徴量のうちPCR 検査陽性者数(単日)のみです。
# シーケンスに沿ったデータを作成する関数
def make_sequence_data(input_data, num_sequence):
# シーケンスデータとラベルデータの器
data = []
# 全体のデータ数取得
num_data = len(input_data)
# 全体からシーケンス分引いた数までループする
for i in range(num_data - num_sequence):
# 1個ずらして、シーケンス分のデータを取得していく
seq_data = input_data[i:i+num_sequence]
# シーケンスの次の要素のデータ(ラベルデータとして1個目の陽性者数のみ)を取得していく
target_data = input_data[:,0][i+num_sequence:i+num_sequence+1]
# シーケンスデータとラベルデータをタプルとして取得していく
data.append((seq_data, target_data))
return data
学習データのシーケンスデータを取得
seq_lengthは時系列の長さです。
**本記事では30日分のシーケンスデータからその1日後を予測します。**そのため、seq_lengthは30にします。
# シーケンス長は1ヶ月分30日とする
seq_length = 30
# train_seq_data=最初のデータを1個ずらしてシーケンス分のデータ(時系列の学習データ群)、train_target=train_seq_dataの次のデータ(ラベルデータ)
train_seq_data = make_sequence_data(train_data_normalized, seq_length)
LSTMネットワークを構築
- 特徴量のサイズ(
input_size)は3(PCR 検査陽性者数(単日),東京平均気温,PCR 検査実施件数(単日))、隠れ層のサイズ(hidden_layer_size)は100、LSTM層のサイズ(num_layer)はデフォルトの1、出力のサイズ(output_size)は1(PCR 検査陽性者数(単日)、batch_firstはTrueとします。 -
batch_firstはTrueなので、LSTMへの入力データxのshapeを(batch_size, seq_length, input_size)です。 - LSTMは
lstm_outと(hn, cn)を出力しますが、hnとcnにはNoneを渡して0ベクトルで初期化します。 -
forward関数ではLSTMにxを入力して、seq_length分の出力lstm_outを得ます。 - seq_length分ある
lstm_outのseq_length方向の最後の値を全結合層に渡して、その結果(prediction)を予測値として返却します。
class LSTM(nn.Module):
def __init__(self, input_size=3, hidden_layer_size=100, output_size=1):
super().__init__()
self.hidden_layer_size = hidden_layer_size
self.lstm = nn.LSTM(input_size=input_size, hidden_size=hidden_layer_size, batch_first=True)
self.linear = nn.Linear(in_features=hidden_layer_size, out_features=output_size)
def forward(self, x):
# LSTMのinputは(batch_size, seq_len, input_size)にする
# LSTMのoutputは(batch_size, seq_len, hidden_layer_size)となる
# hidden stateとcell stateにはNoneを渡して0ベクトルを渡す
lstm_out, (hn, cn) = self.lstm(x, None)
# Linearのinputは(N,∗,in_features)にする
# lstm_out(batch_size, seq_len, hidden_layer_size)のseq_len方向の最後の値をLinearに入力する
prediction = self.linear(lstm_out[:, -1, :])
return prediction
LSTMネットワークインスタンスを生成
LSTMクラスのインスタンスを生成して、GPUデバイスに送ります。
model = LSTM()
model.to(device)
損失関数と最適化関数を定義
損失関数は、平均二乗誤差、最適化関数はAdamとします。Adamの学習率(lr)はデフォルト0.001です。
# 損失関数と最適化関数を定義
criterion = nn.MSELoss()
optimizer = optim.Adam(model.parameters(), lr=0.001)
学習
-
train_seq_dataは学習データのシーケンスデータを取得で取得したデータでシーケンスデータとラベルデータのタプルのリストです。 - 1epochでシーケンスデータ全てのデータを学習するバッチ学習になります。
- 学習ごとに勾配初期化、予測、誤差計算、逆伝搬、勾配計算を行って、1epochごとに誤差を
lossesに入れてあとで表示します。 -
seqとlabelsはshapeが(seq_length, 特徴量)なのでLSTMに渡すためにunsqueezeして(batch_size, seq_length, 特徴量)にします。本記事ではbatch_sizeは1です。
epochs = 50
losses = []
for i in range(epochs):
for seq, labels in train_seq_data:
# seq, labelsのshapeは(seq_length, 特徴量)なのでLSTMに渡すために(batch, seq_length, 特徴量)にする。(batch=1)
seq, labels = torch.unsqueeze(seq, 0), torch.unsqueeze(labels, 0)
seq, labels = seq.to(device), labels.to(device)
optimizer.zero_grad()
y_pred = model(seq)
single_loss = criterion(y_pred, labels)
single_loss.backward()
optimizer.step()
losses.append(single_loss.item())
print(f'epoch: {i}, loss : {single_loss.item()}')
学習時の損失をグラフ表示
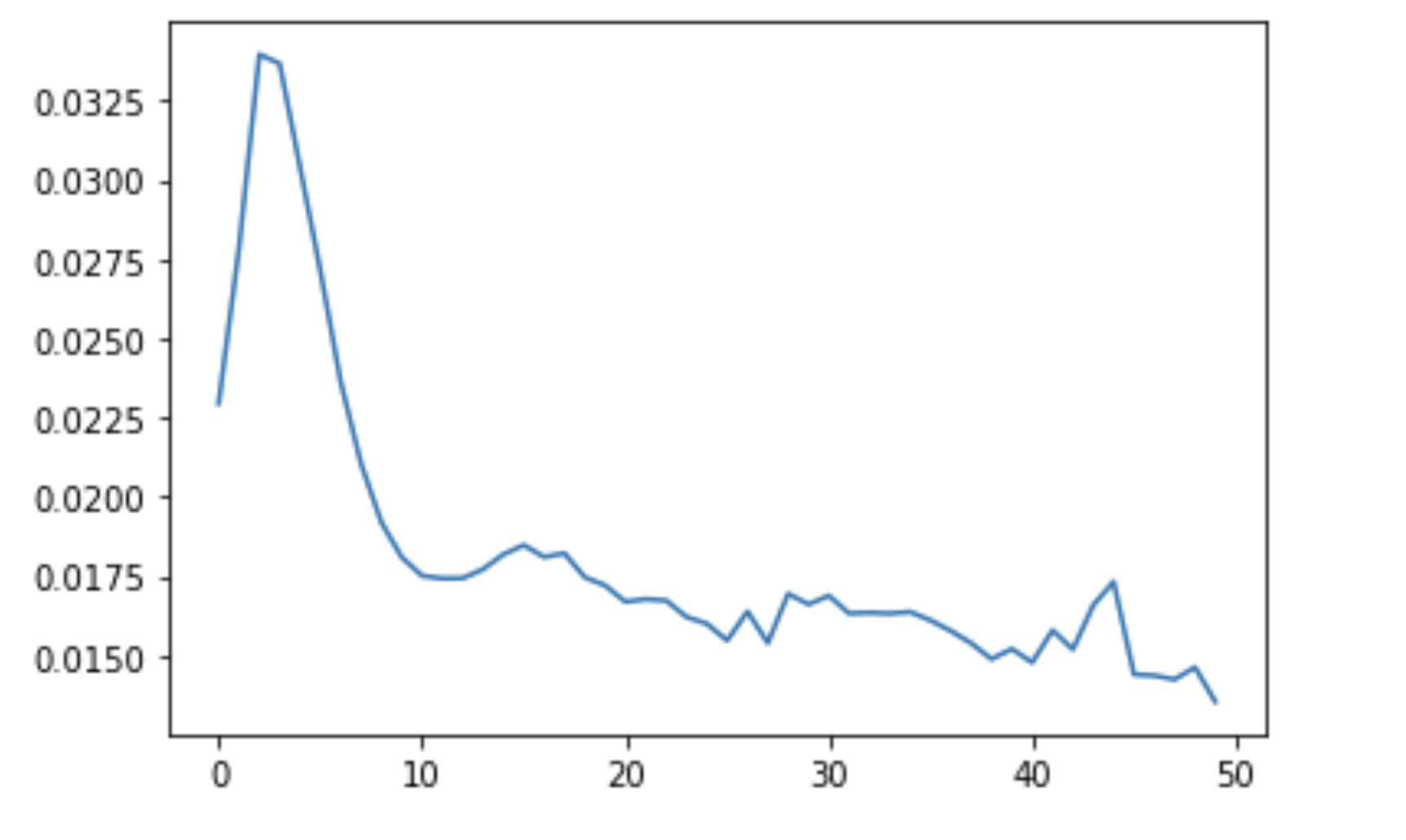
学習データの損失をグラフ表示します。順調に下がっています。
plt.plot(losses)
予測のためのデータ準備
学習によってパラメータが決定したので、このモデルを使って予測してみます。そのために学習データとテストデータに分割するにて分割したtest_dataをデータを正規化で作成したscalerを用いて変換します。train_dataの正規化で取得した統計情報をtest_data正規化時にも使って変換します。(scaler.fit_transform)
また、pred_daysは予測する日数です。本記事では、30日分のtest_dataとこれから予測する30日分を比較してみます。
さらに、test_inputsというリストを用意します。test_inputsには学習データを正規化で正規化した学習データ(train_data_normalized)の直近seq_length(=30)日分を入れます。これは予測時に使います。
# 予測する日数
pred_days = 30
# テストデータの正規化を行う。最小値0と最大値1の範囲で行ってTensor型に変換する。
test_data_normalized = scaler.fit_transform(test_data)
test_data_normalized = torch.FloatTensor(test_data_normalized)
# 予測するためのデータの最初のseq_length分はtrain_dataを使う
test_inputs = train_data_normalized[-seq_length:].tolist()
予測
- LSTMモデルを評価モードにします。
- 予測値を入れる
test_outputsを初期化します。 - 予測したい日数分(
pred_days)ループします。 - LSTMモデルに渡すデータは
test_inputsの後ろからseq_length(=30)日分のデータです。渡した後に、test_inputsにtest_data_normalizedを追加してやります。 - **つまり、予測時に使うデータは全て、結合したデータから必要な特徴量を抽出で作成した実際のデータ
covid19_dataを使います。**予測値は予測のためには使いません。そして予測値はtest_outputsに入れます。
# モデルを評価モードとする
model.eval()
# 予測値を入れるリスト
test_outputs = []
for i in range(pred_days):
seq = torch.FloatTensor(test_inputs[-seq_length:])
seq = torch.unsqueeze(seq, 0)
seq = seq.to(device)
with torch.no_grad():
test_inputs.append(test_data_normalized.tolist()[i])
test_outputs.append(model(seq).item())
予測結果の整形
予測値test_outputsを正規化したデータから元のデータに戻して、actual_predictionsとします。予測値test_outputsを列方向に同じ列を2回足して(30, 3)に変換していますが、これはデータを正規化で使用した統計情報は学習データに対応した特徴量が3次元のため、1次元の予測値を無理矢理3次元にしています。これは本来なら正規化に用いたscalerを学習データとラベル(=予測値)とで分けるべきなのですが、本記事では一緒にしちゃってます。
np_test_outputs = np.array(test_outputs).reshape(-1,1)
# 列方向に同じ値を追加して(30, 3)にする
np_test_outputs2 = np.hstack((np_test_outputs, np_test_outputs))
np_test_outputs3 = np.hstack((np_test_outputs2, np_test_outputs))
actual_predictions = scaler.inverse_transform(np_test_outputs3)
予測結果グラフ表示のための準備
全データ数を取得で取得した全データ数(len_data)から予測したい日数(pred_days)を引いた数から最後までの連番を作成します。これは後で予測結果表示で使います。
x = np.arange(len_data-pred_days, len_data, 1)
print(x)
[290 291 292 293 294 295 296 297 298 299 300 301 302 303 304 305 306 307
308 309 310 311 312 313 314 315 316 317 318 319]
予測結果表示
まずはグラフのサイズを調整します。
fig_size = plt.rcParams['figure.figsize']
fig_size[0] = 10
fig_size[1] = 5
plt.rcParams['figure.figsize'] = fig_size
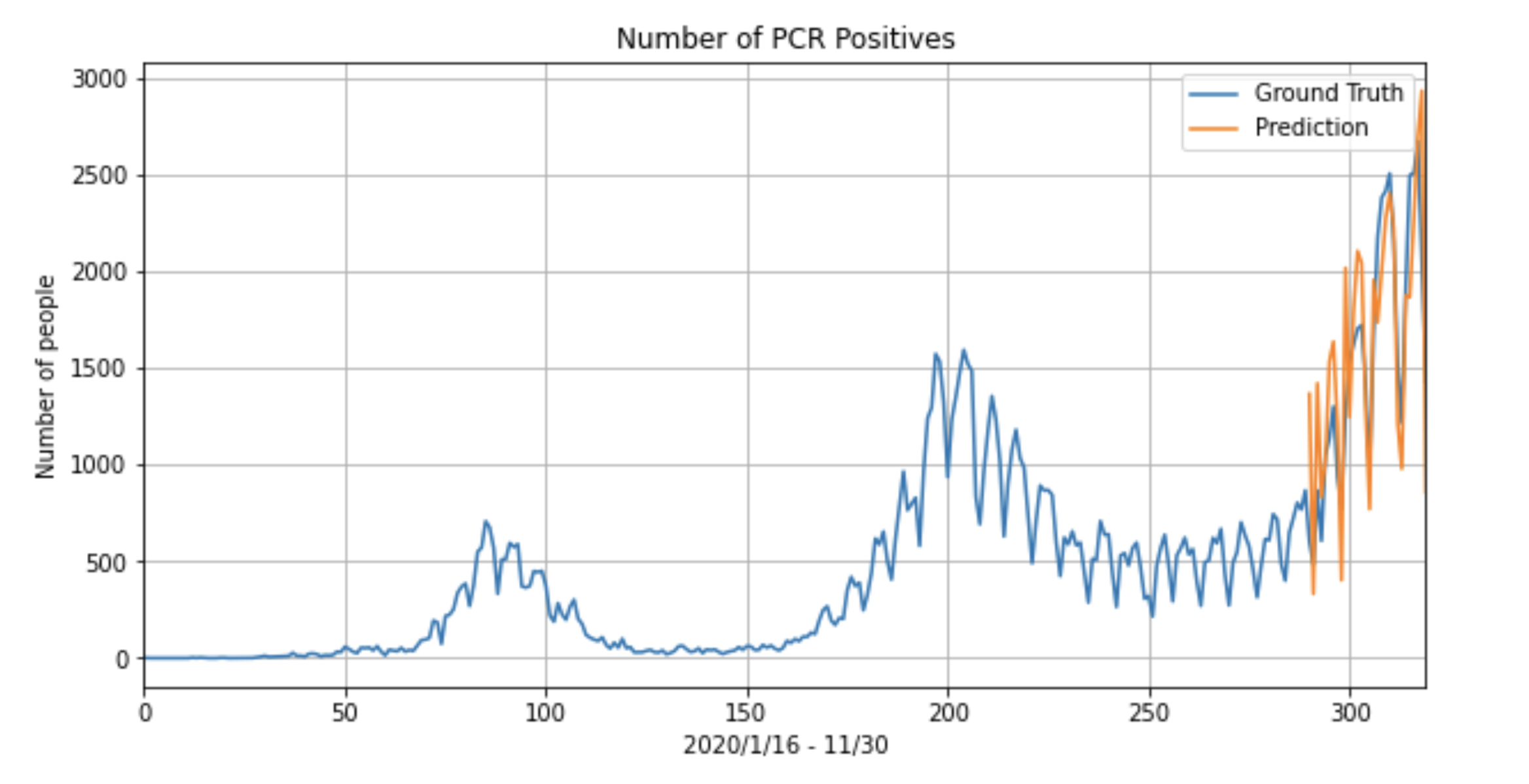
次に結合したデータから必要な特徴量を抽出で作成した実際のデータcovid19_dataをGround Truth、Predictionを予測値(actual_predictions)としてグラフを表示します。範囲は2020/1/16から2020/11/30です。
plt.title('Number of PCR Positives')
plt.ylabel('Number of people')
plt.grid(True)
plt.autoscale(axis='x', tight=True)
plt.plot(data['PCR 検査陽性者数(単日)'], label='Ground Truth')
plt.plot(x, actual_predictions[:,0], label='Prediction')
plt.xlabel('2020/1/16 - 11/30')
plt.legend()
plt.show()
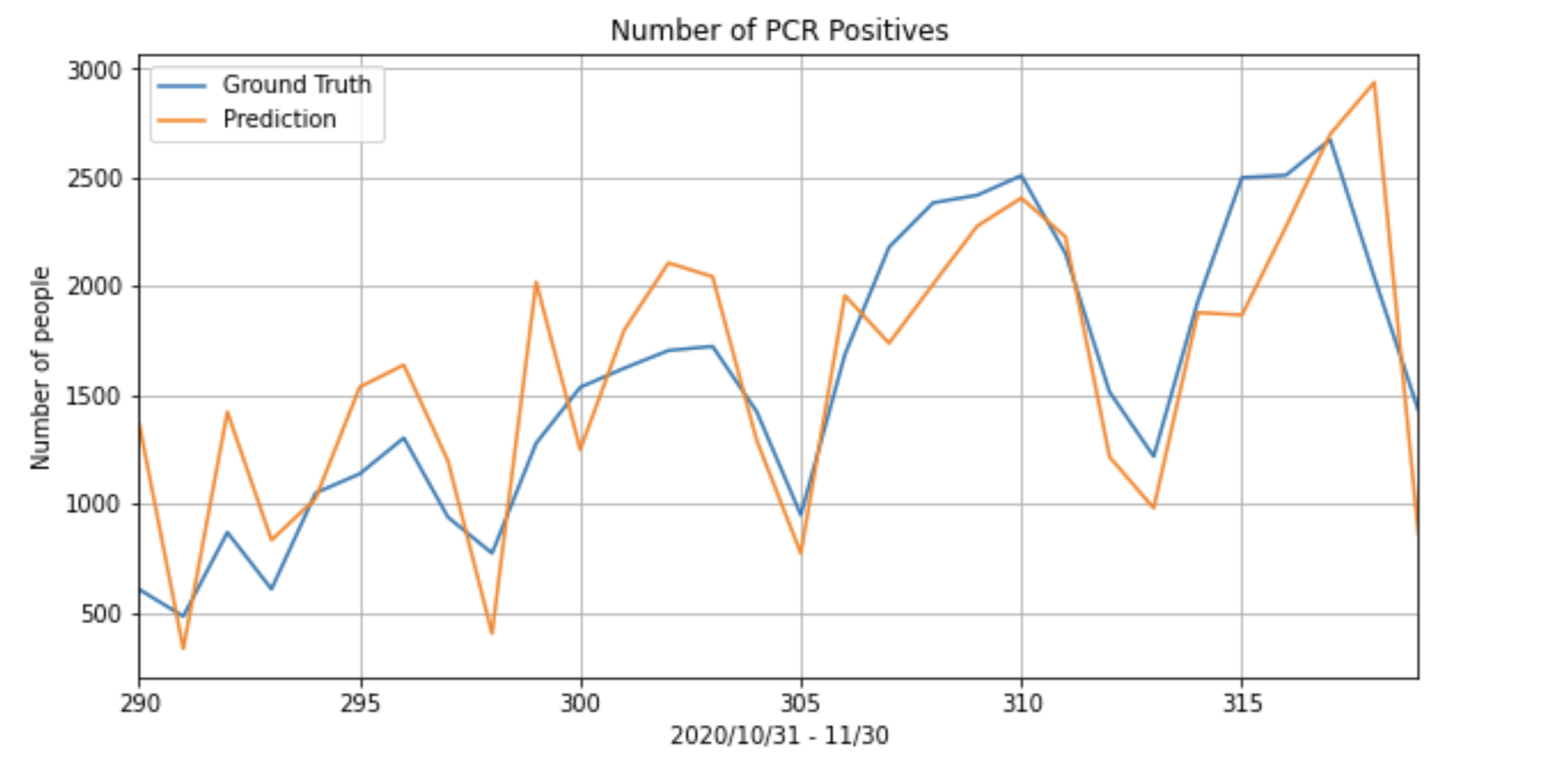
最後に上のグラフの範囲を2020/10/30から2020/11/30の30日間にしたものを表示します。
**いい感じに直近30日分を予測できました。**少なくとも増減傾向は読めていると思います。
plt.title('Number of PCR Positives')
plt.ylabel('Number of people')
plt.grid(True)
plt.autoscale(axis='x', tight=True)
plt.plot(x, data['PCR 検査陽性者数(単日)'][-1*pred_days:], label='Ground Truth')
plt.plot(x, actual_predictions[:,0], label='Prediction')
plt.xlabel('2020/10/31 - 11/30')
plt.legend()
plt.show()
終わりに
**本記事では、PyTorchを使ってLSTMを使った時系列予測をコロナという題材で行ってみました。**データ数が1年間もないということ、また、特徴量に、電車の乗車数や街への外出人数といったものがなかったというのもあり、また、これらの情報が取得できたときにトライしてみようと思います。
全てのコードはhttps://github.com/tsubauaaa/LSTM_covid19_nominibatch_multivariate/blob/master/LSTM_covid19_nominibatch_multivariate.ipynb こちらをご確認ください。
最後までお読みいただきまして、ありがとうございました。