創薬 Advent Calendar (Dry) 23日目記事
ねらい
- 有機化学のバックグラウンドを持つメドケムやケムインフォの人向け
- 生体高分子に含まれる低分子や水の構造がどれだけ妥当か確認する
まとめ
- PDB登録構造はけっこう不正確です
- 電子密度との一致度と(低分子なら)立体化学に気をつけましょう
- Cootで電子密度を見ましょう
- PDB登録構造ならValidation Reportを見ましょう
自己紹介
- アカデミアで生体高分子の構造解析をしています
- 阻害剤開発にも興味がありますが、企業の現場を知っているわけではありません
- 見当違いのことを言っていたら、どうぞご指摘くださいませ
Introduction
合理的な創薬において標的-低分子複合体の立体構造は有用な情報を提供します。現在、そのような複合体構造の決定にはX線結晶構造解析が主に使われています。ところが、PDB登録構造には低分子構造の妥当性に問題があるものが含まれており、利用の妨げになっています。123
精密化
下図にX線結晶構造解析の流れを示します(単純化しています)。
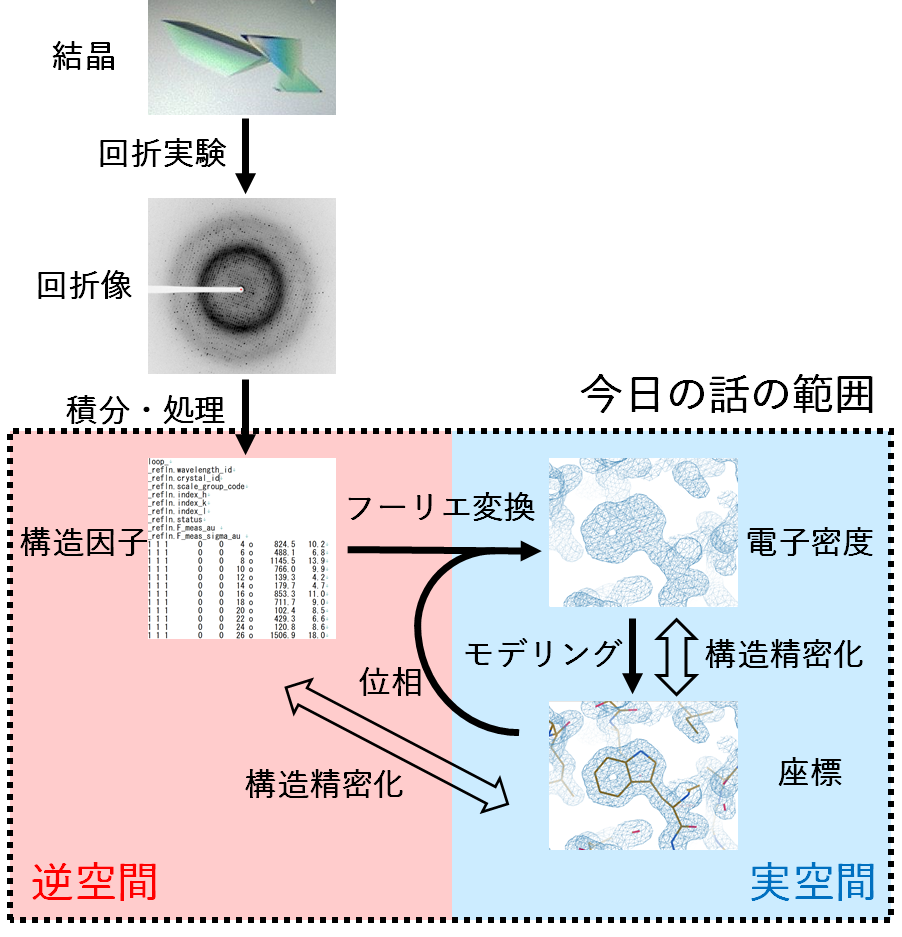
構造精密化は、原子モデル(座標と温度因子をもつ原子の集合)を動かして回折データ(やそこから得られる電子密度)と辻褄が合うものをつくるプロセスです。ところが、多くの生体高分子のX線結晶構造解析においては、回折データ数が少なすぎて、それだけでは原子モデルをうまく精密化することができません。
そのため、多くの場合では下式のような関数を用い、それを最小化することを目標とします。4
Etotal = wdata * Edata + Egeom
wdata: 重み
Edata: 回折データと原子モデルのずれを表す項
Egeom: 立体化学(共有結合長・角度・二面角など)の標準値からのずれを表す項
タンパク質や核酸やよくある低分子化合物については、立体化学の標準値がすでに求められており、結晶学者はそれらをそのまま利用することができます。しかし、稀な化合物については、結晶学者がその化学構造の標準値をいちいち計算してパラメータファイルを作成し、構造精密化に用いなければいけません。しばしば起こることですが、間違ったパラメータファイルを用いると化合物構造にひずみが生じてしまいます。
低分子構造の正確さ
結晶構造の質を表す統計値として、しばしばRfreeなどが使われます。ところが、これらの値は構造全体の正確さを表すものであり、その致命的な間違い(空間群や主鎖トレースの間違い、不適切な精密化戦略など)を検出することはできますが、局所的な正確さにはあまり影響を受けません。低分子構造の正確さを検討するには局所的な指標が必要となるのですが、適した指標を選ぶのは難しく、識者の間で議論が行われています。12
ここでは、低分子構造の正確さの指標として
・構造モデルと電子密度との一致度
・立体化学の標準値からのずれ
の2種類について紹介します。
Cootで電子密度を見ましょう
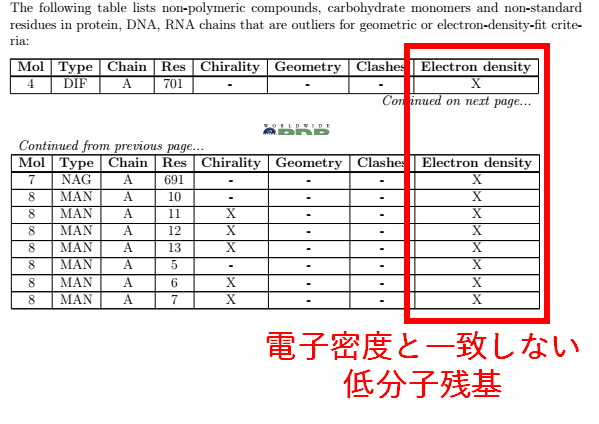
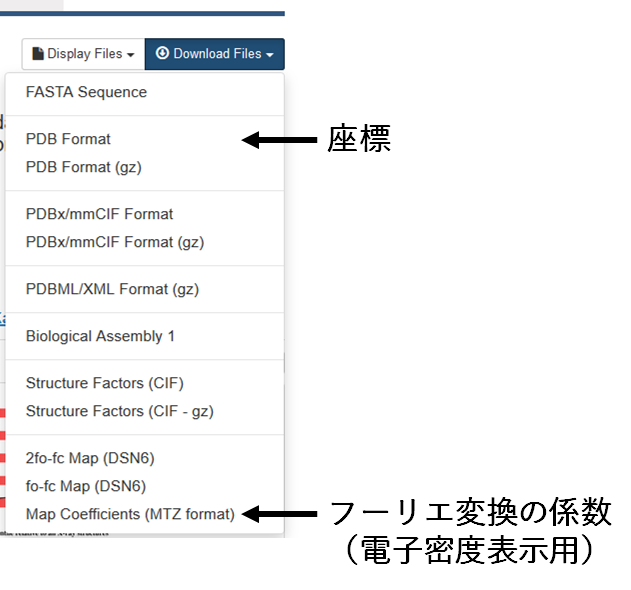
構造モデルと電子密度との一致度を確認するためには、実際に電子密度を見てみるのが手っ取り早いです。PDBサイトから構造3IB0の座標と電子密度を表示するためのファイルをダウンロードしましょう。
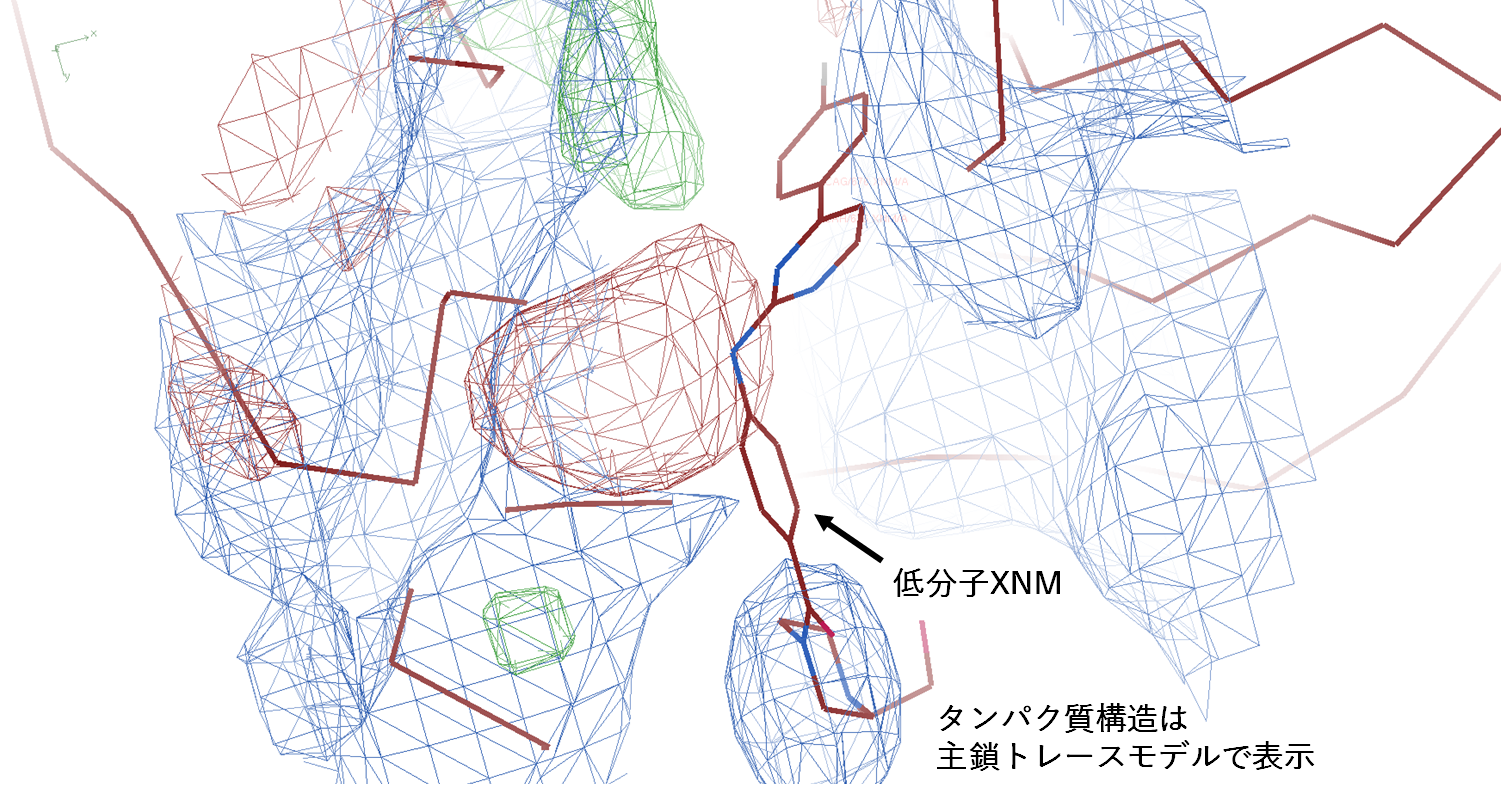
次に、プログラムCootを用いて、ダウンロードしたファイルを開き、化合物DIF(A鎖、残基番号701)周辺の構造を見てみましょう。magattacaさんのブログにCootの使い方が詳しく書いてあります。
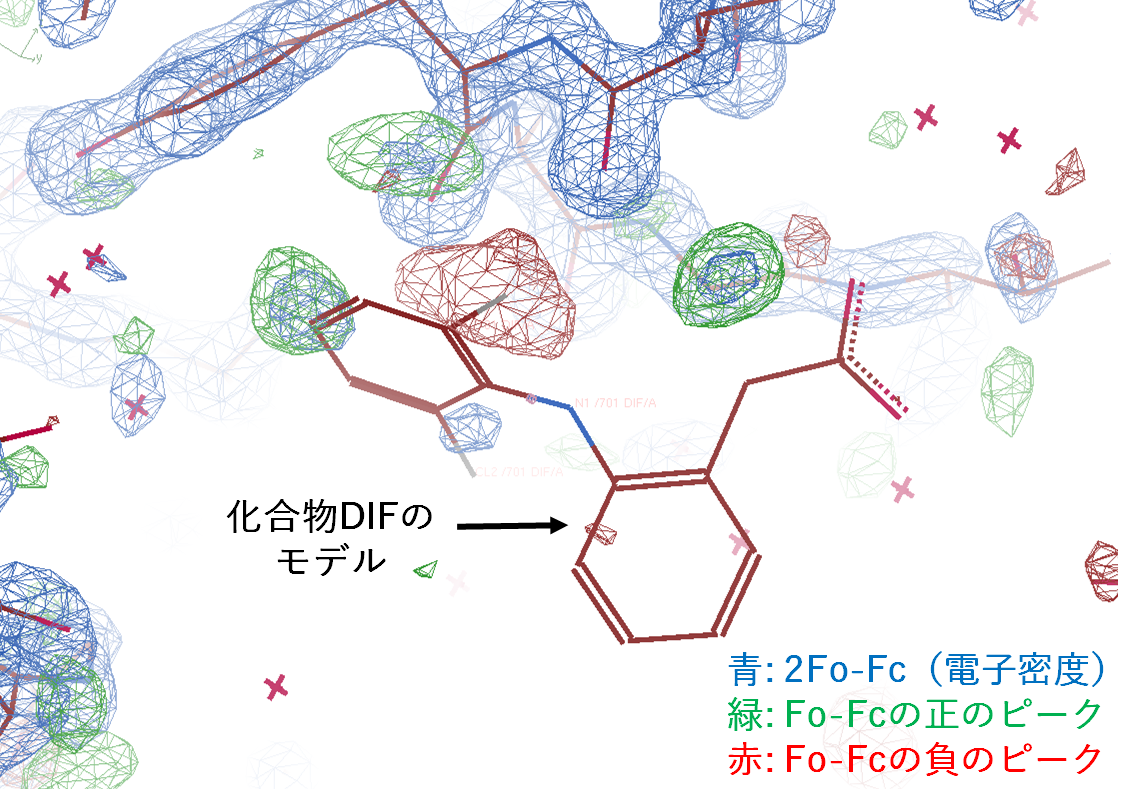
電子密度マップ(2Fo-Fcマップ、青色で表示)と化合物DIFのモデルがまったく一致していないことが分かります。おそらくこの部分にはDIFでなく水分子が結合しているのに、むりやりDIFを置いてしまったのでしょう。緑と赤で示されるマップは、それぞれFo-Fcマップの正と負の密度を示しています。正密度は電子密度があるはずなのに原子モデルが置かれていない部分を強調して示し、負密度はその逆(電子密度がないはずなのに原子モデルが置かれてしまった部分)を示します。ひとつの塩素原子(原子番号17、電子数が多い)の位置にFo-Fcマップの強い負ピークが観測されています。
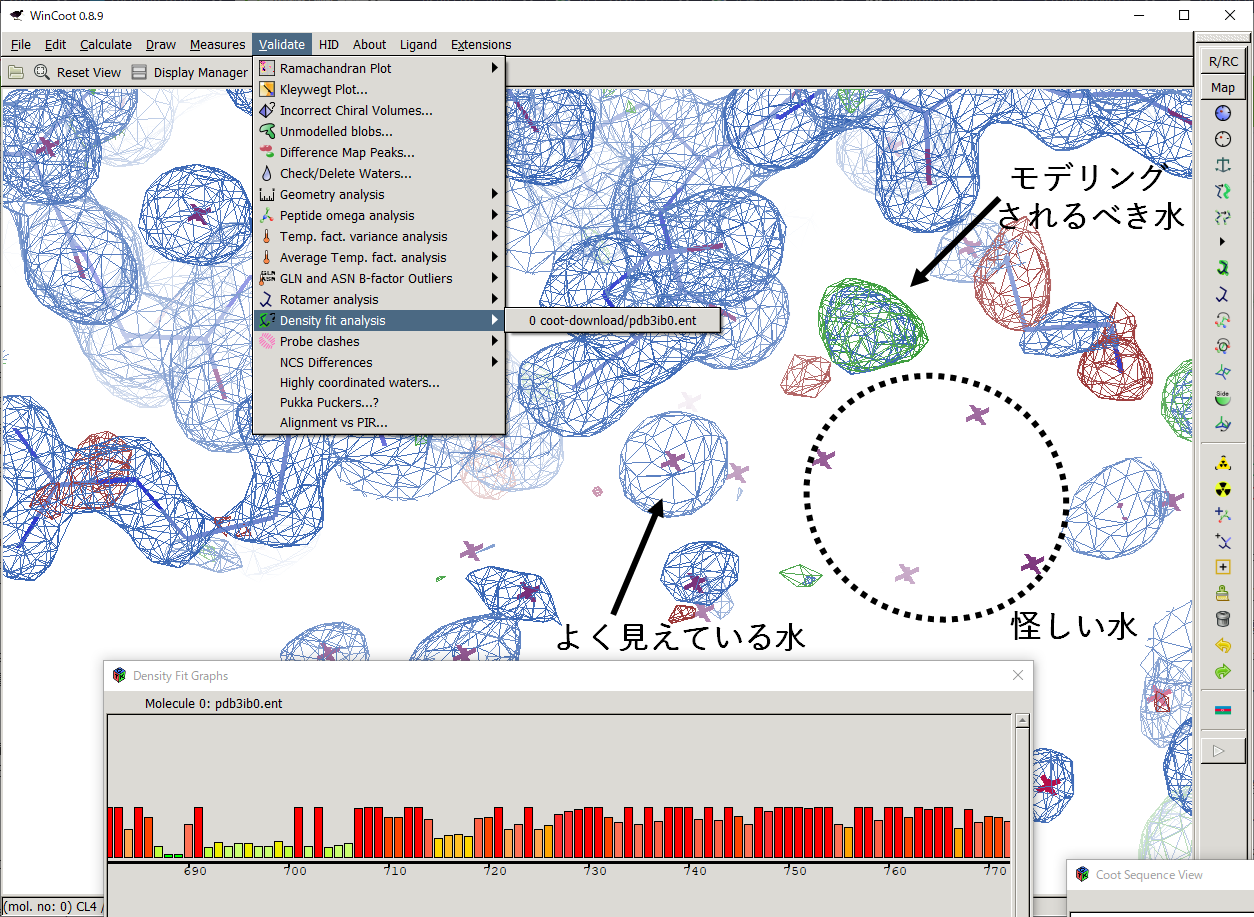
次に、上部のValidateタブから"Density fit analysis"を選んでみましょう。座標と電子密度との一致度が残基単位で表示されます。いくつかの水分子について電子密度との一致度が低いことがわかります(実際にいくつかの水分子の周りの電子密度を観察してみましょう)。また、2Fo-FcとFo-Fcの両マップで観察される丸い密度は、おそらく存在するはずなのにまだモデリングされていない水です。
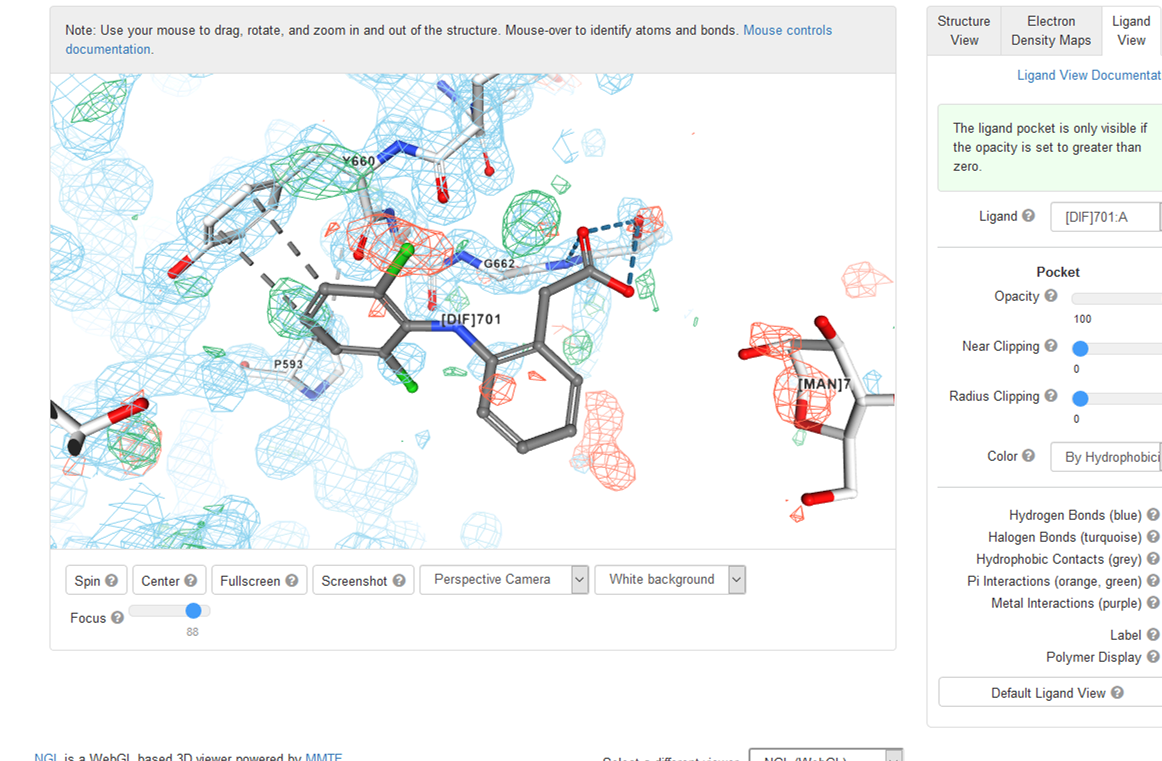
実はブラウザを用いてもPDBサイトの「3D View」タブから同種の観察を行えます。私はCootのほうが多機能で好きですが。
Validation Reportを見る
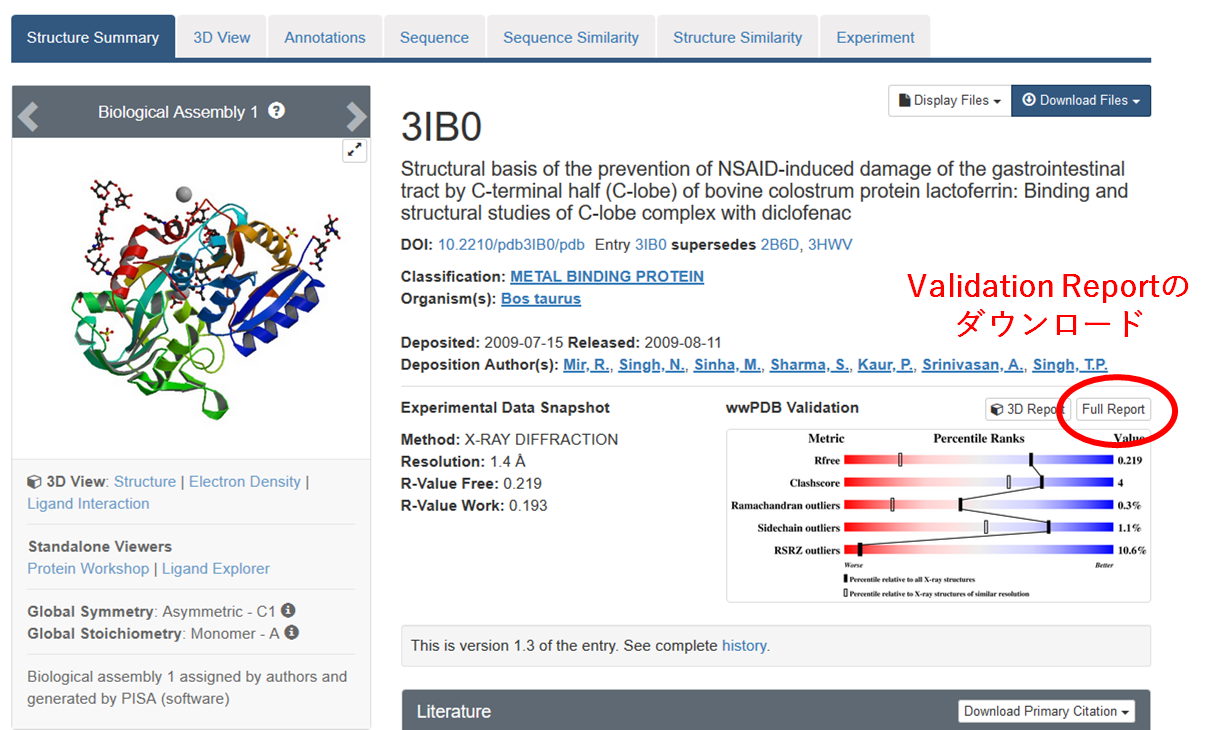
PDBは登録を受け付けた構造についてValidation Reportを作成し、もし問題があればそれを登録者に喚起することになっています。構造3IB0のValidation Reportをダウンロードして見てみましょう。
電子密度とモデル構造の一致度を表す値
RSR (Real-space R value)とRSCC (Real-space correlation coefficient) が主に使われています。RSRは0.4以上、RCSSは0.8以下が不十分な一致を表すそうです2。これらの値は、モデル構造の信頼性を客観的に判断し、また多数の構造を自動で処理するのに役立つでしょう。
残基ごとのRSRやRSCCの値は、ヨーロッパのPDBサイトであるPDBeからダウンロードできるValidation dataファイルに掲載されています(RCSB PDBサイトからダウンロードできるかどうかは分かりませんでした)。
Validation dataファイルによると、DIFのRSRは0.506、RSCCは0.264であることが分かります。
resname="DIF" resnum="701" rscc="0.264" rsr="0.506"
インハウスの構造を扱う場合にも、多くの精密化プログラムはRSRとRSCCを書き出してくれるので、参考にするといいでしょう。
立体化学の妥当性評価
Validation Reportは低分子の立体化学の妥当性も判定します。ここでは、類似の部分構造を持つ低分子のX線構造から、複合体構造中の低分子の様々なパラメータを推定し評価しています。構造3RZFのValidation Reportを見てみましょう。
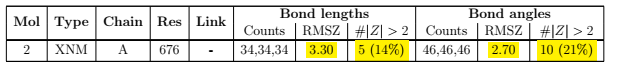
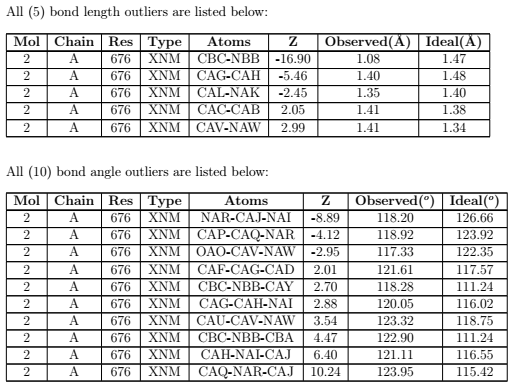
観測値が標準値からどれだけずれているかをZスコアで表し、それが2を超えている場所の数が示されています(2は厳しめの閾値のようです2)。Zスコアが-16.9や+10.24という値をとるものもありますね。このような場合、低分子の立体構造情報を用いるのには慎重になったほうがいいでしょう。
実はこの結晶構造は最初は構造3QADとして登録され、そのあとで修正が入り3RZFに差し替えられたという経緯があります。3QADと3RZFはけっこう低分子構造に違いがありますが、信頼性はどっちもどっちに思えます。3QADを報告した論文は2011年にNatureに発表されています5が、この複合体構造を信頼して解析や実験を行った人はどんな結果を得たのでしょうか。
複合体構造の精密化に用いる低分子パラメータファイルは注意深く作成しなければいけません。インハウスで構造解析が行われている場合には、どのようなパラメータファイルが使われているかを見せてもらてもいいかもしれません。
-
Outcome of the First wwPDB/CCDC/D3R Ligand Validation Workshop Structure 2016 ↩ ↩2
-
Validation of ligands in macromolecular structures determined by X-ray crystallography Acta Crystallogr D Struct Biol. 2018 ↩ ↩2 ↩3 ↩4
-
The good, the bad and the twisted: a survey of ligand geometry in protein crystal structures J Comput Aided Mol Des. 2012 ↩
-
Crystal structure of inhibitor of κB kinase β Nature 2011 ↩