概要
bioinformatics - ChEMBLから全化合物データをダウンロードして分割したsdfファイルをつくる - Qiita
http://qiita.com/tonets/items/272ed39b1cc663c47fac
の続きになります.
EMBL-EBIが運営する化合物/薬物のデータベース ChEMBL にはどんな化合物があるのだろう,というのを見ていきたいと思います.
手始めに全ての化合物の原子の数がどんな分布になっているかを見てみます.
ChEMBLからsdfをダウンロードしてバラす
こちらの内容をやっておきます.
bioinformatics - ChEMBLから全化合物データをダウンロードして分割したsdfファイルをつくる - Qiita
http://qiita.com/tonets/items/272ed39b1cc663c47fac
sdfファイルの原子数をカウントする
sdfファイルを開いてみると以下のようになっています.
CHEMBL601
SciTegic12111210002D
9 8 0 0 0 0 999 V2000
1.9000 -3.8875 0.0000 C 0 0
1.9000 -3.1792 0.0000 O 0 0
3.7167 -5.0000 0.0000 O 0 0
3.7375 -4.2875 0.0000 C 0 0
2.5042 -4.2542 0.0000 C 0 0
1.2750 -4.2375 0.0000 O 0 0
3.1375 -3.9250 0.0000 C 0 0
4.9750 -4.3292 0.0000 N 0 0
4.3625 -3.9542 0.0000 C 0 0
2 1 2 0
3 4 2 0
4 7 1 0
5 1 1 0
6 1 1 0
7 5 1 0
8 9 1 0
9 4 1 0
M END
> <chembl_id>
CHEMBL601
$$$$
今回のポイントは 4行目の最初の3文字が原子の数 になっているということです.4文字目~6文字目は結合数になっているので要注意です.何が言いたいかというと,下のようなファイルも存在します.
CHEMBL440060
11280716012D 1 1.00000 0.00000 0
206208 0 1 0 999 V2000
2.0792 -0.9500 0.0000 N 0 0 0 0 0 0 0 0 0
1.4671 -0.5958 0.0000 C 0 0 2 0 0 0 0 0 0
0.8510 -0.9500 0.0000 C 0 0 0 0 0 0 0 0 0
以下略
原子数206と結合数208がくっついてしまっています.というわけで,丁寧に切り取ってあげましょう.今回は以下のpythonスクリプトを使いました.普通にワンライナーでできますが.
# !/usr/bin/python
import re
import sys
import linecache
if (len(sys.argv) != 2):
print 'Usage: # python %s sdf' % sys.argv[0]
quit()
sdffile = sys.argv[1]
fi = open(sdffile, 'r')
line = fi.readlines()
Natom = int(line[3][0:3])
print sdffile, ", ", Natom
fi.close()
$ python count.py CHEMBL440060.sdf
> CHEMBL440060.sdf , 206
ChEMBLの全部の化合物の原子数をカウントする
これを1,404,752個全てに適用します.
とりあえず32コアのサーバで以下のシェルスクリプトを使ってやりました.
# !/bin/sh
PROCMAX=32
np=$PROCMAX
FP=chembldata.txt
DIR=chembl_sdfs
if [ -e $FP ]; then
rm -f $FP
echo "rm -f $FP"
fi
touch $FP
for i in $DIR/*; do
python count.py $i >> $FP &
let np=np-1
if [ $np -eq 0 ]; then
wait
np=$PROCMAX
fi
done
これでchembldata.txtというファイルに全部の原子数が書き込まれます.6時間くらい*かかりました.バラさないでchembl_19.sdfから直接やった方が絶対速いですね(てへぺろ).
*追記:やり直してみたら2時間13分で終わりました.
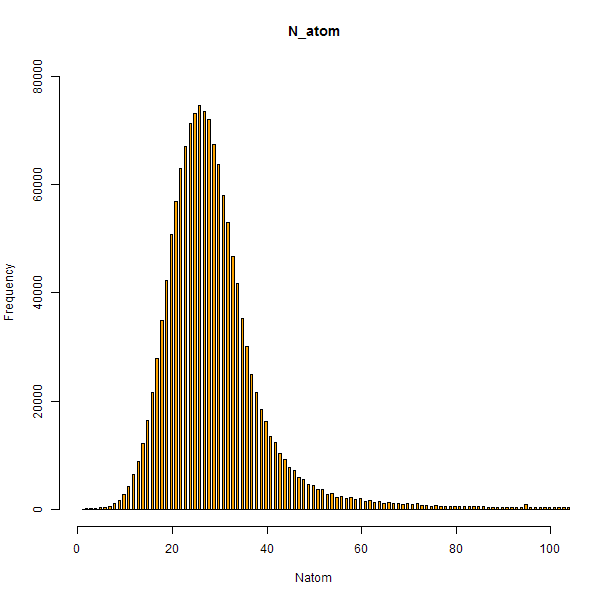
ヒストグラムを描く(R)
Rを使ってヒストグラムを描いてみます.
data <- read.csv("chembldata.txt", header=FALSE)
Natom <- t(t(data[2]))
png("plot.png", width = 600, height = 600)
hist(Natom, col = "orange", main = "N_atom", breaks = "Scott", xlim=c(0,100), ylim=c(0,80000))
dev.off()
ちなみに $N_{atom}>100$ の化合物はだいたい1万個ぐらいあります.
> sum(Natom > 100)
[1] 11443
ピークは $N_{atom}=26$ で,原子数26の化合物は74,693個ありました.